RIBO-seq i bakterier: en prøvesamling og bibliotekforberedelsesprotokoll for NGS-sekvensering
Summary
Her beskriver vi stadier av prøvetaking og forberedelse for RIBO-seq i bakterier. Sekvensering av bibliotekene utarbeidet i henhold til disse retningslinjene resulterer i tilstrekkelige data for omfattende bioinformatisk analyse. Protokollen vi presenterer er enkel, bruker standard laboratorieutstyr og tar syv dager fra lysis til å skaffe biblioteker.
Abstract
Ribosomets profileringsteknikk (RIBO-seq) er for tiden det mest effektive verktøyet for å studere prosessen med proteinsyntese in vivo. Fordelen med denne metoden, sammenlignet med andre tilnærminger, er dens evne til å overvåke oversettelse ved å kartlegge nøyaktig plasseringen og antall ribosomer på en mRNA-transkripsjon.
I denne artikkelen beskriver vi de påfølgende stadiene av prøveinnsamling og forberedelse for RIBO-seq-metoden i bakterier, og fremhever detaljene som er relevante for planlegging og gjennomføring av eksperimentet.
Siden RIBO-seq er avhengig av intakte ribosomer og relaterte mRNAer, er nøkkeltrinnet rask hemming av oversettelse og tilstrekkelig oppløsning av celler. Dermed foreslår vi filtrering og flash-frysing i flytende nitrogen for cellehøsting med en valgfri forbehandling med kloramfenikol for å arrestere oversettelse i bakterier. For oppløsningen foreslår vi sliping av frosne celler med mørtel og pestle i nærvær av aluminiumoksid for å forstyrre celleveggen mekanisk. I denne protokollen er det ikke nødvendig med sukrosepute eller en sukrose gradient ultracentrifugation for monosom rensing. I stedet påføres mRNA-separasjon ved hjelp av polyakrylamidgelelektroforese (PAGE) etterfulgt av det ribosomale fotavtrykket (28-30 nt-bånd) og gir tilfredsstillende resultater. Dette forenkler i stor grad metoden, samt reduserer tids- og utstyrskravene for prosedyren. For bibliotekforberedelse anbefaler vi at du bruker det kommersielt tilgjengelige lille RNA-settet for Illumina-sekvensering fra New England Biolabs, etter produsentens retningslinjer med en viss grad av optimalisering.
De resulterende cDNA-bibliotekene presenterer passende mengde og kvalitet som kreves for neste generasjons sekvensering (NGS). Sekvensering av bibliotekene utarbeidet i henhold til de beskrevne protokollresultatene i 2 til 10 mln unikt kartlagte lesninger per prøve som gir tilstrekkelige data for omfattende bioinformatisk analyse. Protokollen vi presenterer er rask og relativt enkel og kan utføres med standard laboratorieutstyr.
Introduction
Ribosomprofileringsteknikken (RIBO-seq) ble utviklet i laboratoriet til Jonathan Weissman ved University of California, San Francisco1. Sammenlignet med andre metoder som brukes til å studere genuttrykk på translasjonsnivå, fokuserer RIBO-seq på hvert ribosome som binder seg til mRNA og gir informasjon om plasseringen og det relative antallet ribosomer på en transkripsjon. Det muliggjør overvåking av prosessen med proteinsyntese in vivo og kan gi enkel kodonoppløsning og nøyaktighet som tillater måling av ribosomets tetthet på begge, den enkelte mRNA og langs hele transkripsjonen i cellen. Ved grunnlaget for RIBO-seq-teknikken ligger det faktum at ribbeosomet under oversettelse binder mRNA-molekylet og dermed beskytter det begravde fragmentet av transkripsjonen fra en ribonuklease fordøyelse. Ved tilsetning av ribonuklease fordøyes den ubeskyttede mRNA og fragmentene vedlagt av ribosomer – vanligvis ~ 28-30 nt lange – forblir intakte. Disse fragmentene, kalt ribosomale fotavtrykk (RF), kan deretter isoleres, sekvenseres og kartlegges på transkripsjonen de stammer fra, noe som resulterer i deteksjon av ribosomenes nøyaktige posisjon. Faktisk har ribososom evnen til å beskytte mRNA fragmenter blitt brukt siden 1960-tallet for å studere ribosomal binding og oversettelse initieringssteder (TIS)2,3,4. Men med fremskrittet i dyp sekvenseringsteknologi har RIBO-seq blitt en gullstandard for oversettelsesovervåking5 som gjennom ribosomets engasjement kan gi en genom-bred informasjon om proteinsyntese6. Ribosomprofilering fylte det teknologiske gapet som eksisterte mellom kvantifisering av transkripsjonen og proteomet6.
For å gjennomføre ribosomprofilering må vi få cellelys av organismen som hadde vokst under de undersøkte forholdene. Å forstyrre disse forholdene under celleinnsamling og lysis kan gi upålitelige data. For å forhindre dette brukes ofte oversettelseshemmere, rask høsting og blitsfrysing i flytende nitrogen. Celler kan lyses ved kryogen sliping i en mekanisk homogenisator som en miksermølle7,8 eller en perle beater9, og ved triturasjon gjennom en pipette10 eller med en nål11. Lysisbufferen kan tilsetts like før eller kort tid etter pulverisering av cellene. I vår protokoll bruker vi flytende nitrogen til prekolmørtel og pestle, samt aluminiumoksid som en mildere tilnærming til forstyrrelse av bakteriell cellevegg, noe som forhindrer RNA-skjæring ofte oppstår når metoder som sonifisering påføres. Etter pulverisering legger vi til en iskald lysisbuffer i det avkjølte innholdet i mørtelet. Valg av en passende lysisbuffer er viktig for å oppnå den beste oppløsningen av ribosomale fotavtrykk. Siden ionstyrke påvirker både RF-størrelsen og leserammepresisjonen, anbefales det for tiden å bruke lysisbuffere med lav ionisk styrke og bufferkapasitet, selv om det ser ut til at buffersammensetningen ikke påvirker ribosomal belegg på mRNAs11,12. Viktige komponenter i lysisbufferen er magnesiumioner, hvis tilstedeværelse forhindrer dissosiasjon av ribosomale underenheter og hemmer spontane konformasjonsendringer i bakterielle ribosomer11,13. Kalsiumioner spiller også en betydelig rolle og er avgjørende for aktiviteten til mikrokokkkjerne (MNase) som brukes i bakteriell ribosomprofileringsmetode14. Tillegg av guanosin 5′-[β,γ-imido]triphosfat (GMP-PNP), en ikke-hydrolysert analog av GTP, sammen med kloramfenikol hemmer oversettelse under lysis15.
Når lysatet oppnås, avklares det ved sentrifugering og deles inn i to deler, hver for en RIBO-seq og en total mRNA-sekvensering med høy gjennomstrømning (RNA-seq) siden de utføres samtidig (figur 1). RNA-seq gir et referansepunkt som gjør det mulig å sammenligne data fra både RIBO-seq og RNA-seq under dataanalyse. Den undersøkte translatome er definert ved normalisering av ribosomale fotavtrykk til mRNA overflod16. Data fra RNA-seq kan også bidra til å identifisere kloning eller sekvensering av artefakter17.
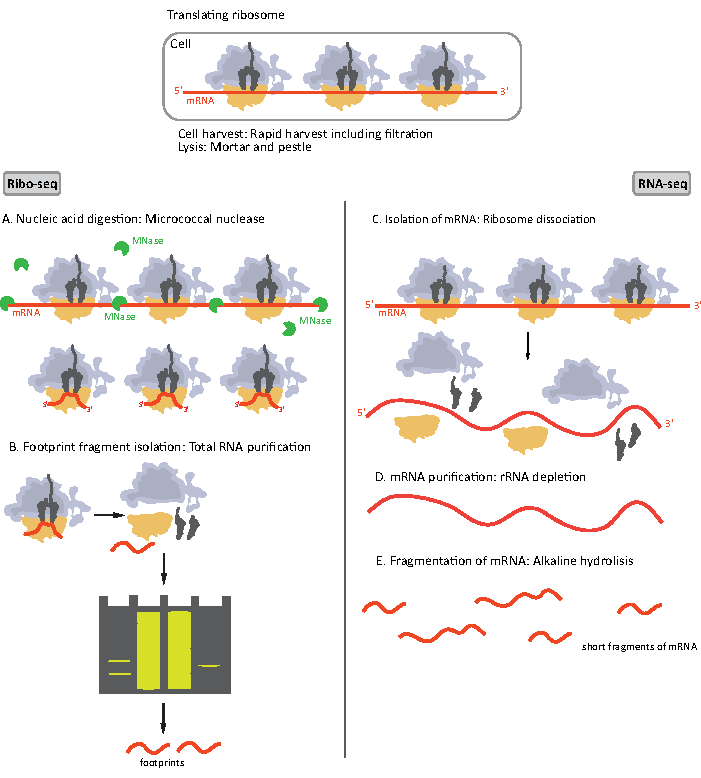
Figur 1. Skjema for mRNA prøvepreparering for RIBO-seq og RNA-seq. For RIBO-seq bibliotek forberedelse fordøyes RNA med MNase (A), etterfulgt av størrelsesvalget av RF på ~ 28-30 nt lengde (B); for RNA-seq RNA er isolert (C), utarmet av rRNA (D), og den resulterende mRNA fragmenteres tilfeldig i fragmenter av varierende lengde (E). Klikk her for å se en større versjon av denne figuren.
De første trinnene i prosedyren for prøvepreparering for RIBO-seq og RNA-seq varierer noe (figur 1). For ribosomal profilering må lysatet fordøyes av en bestemt endonuklease for å forringe mRNA-molekylene som ikke er beskyttet av ribosomene. I standardprotokoller gjenvinnes de oppnådde monosomene av en sukrosepute ultracentrifugation eller en sukrose gradient ultracentrifugation8,14. I denne artikkelen viser vi at dette trinnet ikke er nødvendig for å isolere RF som kreves for RIBO-seq i bakterier, på samme måte for eukaryote celler18, og at størrelsesvalg av riktig lengde mRNA-fragmenter fra polyakrylamidgelen er tilstrekkelig.
For RNA-seq oppnås mRNA ved uttømming av rRNA fra de totale RNA – rRNA-molekylene hybridiserer til de biotinylerte oligonukleotidsondene som binder seg til de streptavidinbelagte magnetiske perlene. RRNA-oligonukleotid-perlekompleksene fjernes deretter fra prøven med en magnet som resulterer i en rRNA utarmet prøve19,20. De rensede mRNA-molekylene fragmenteres deretter tilfeldig av alkalisk hydrolyse. De oppnådde fragmentene av mRNA samt ribosomale fotavtrykk konverteres til cDNA-biblioteker og fremstilles for dyp sekvensering (figur 2). Dette innebærer reparasjon av ender som trengs etter alkalisk hydrolyse (for mRNA) og enzymatisk fordøyelse (for RF): defosforylering av 3′ ender etterfulgt av fosforylering av 5′ ender. De neste trinnene er adaptere ligation og omvendt transkripsjon for å lage cDNA innsatser innrammet av sekvenser som kreves for neste generasjon sekvensering (NGS) ved hjelp av Illumina plattform. Den siste fasen av bibliotekforberedelse er en PCR-reaksjon der konstruksjonene forsterkes og merkes med prøvespesifikke strekkoder for å tillate multipleksing og sekvensering av ulike prøver på en kanal. Før sekvensering vurderes kvaliteten og mengden av bibliotekene av høysensitiv DNA on-chip elektroforese. cDNA-biblioteker med riktige parametere kan deretter grupperes og sekvenseres. Sekvensering kan utføres på forskjellige Illumina-plattformer, for eksempel MiSeq, NextSeq eller HighSeq, avhengig av antall biblioteker, nødvendig leselengde og sekvenseringsdybde. Etter sekvensering utføres den bioinformatiske analysen.

Figur 2. Klargjøring av biblioteket. Biblioteksforberedelse inkluderer endereparasjon, adaptere ligation, omvendt transkripsjon og forsterkning med barkoding. Klikk her for å se en større versjon av denne figuren.
Ribosomet profilering er en universell metode som lett kan modifiseres og justeres i henhold til det vitenskapelige spørsmålet. Opprinnelig ble den brukt i gjær1, men kort tid etter at den ble brukt på bakterieceller21 samt eukaryote modellorganismer, inkludert mus10, sebrafisk22, fruktflue23 og Arabidopsis thaliana24. Den ble også brukt til å studere forskjellige ribosomtyper: cytoplasmatisk, mitokondrie25,26 og kloroplast27,28. I eukaryoter er RIBO-seq vanligvis tilpasset og raffinert for å undersøke spesifikke aspekter ved oversettelse, inkludert initiering10,11,29,30,31,32, forlengelse1,10,11,31,33, ribosom stalling33 og konformasjonsendring33. De fleste modifikasjonene innebærer bruk av forskjellige oversettelseshemmere. I bakterier har imidlertid analoge studier vært vanskelige å gjennomføre på grunn av hemmeres paucity med den nødvendige virkningsmekanismen34. Den mest brukte oversettelseshemmeren i bakterier er kloramfenikol (CAM) som binder seg til peptidyloverføringssenteret (PTC) og forhindrer riktig posisjonering av aminoacyl-tRNA på A-området. Som et resultat forhindrer CAM dannelsen av en peptidbinding som fører til å arrestere de langstrakte ribosomene35. Andre eksempler på oversettelseshemmere i bakterier er tetracyklin (TET)36, retapamulin (RET)34 og Onc11237 som har blitt brukt til å undersøke oversettelsesinitieringssteder. TET, som forhindrer tRNA-levering til ribosomet ved å overlappe direkte med antikodonstammeløkken til tRNA på A-stedet, ble opprinnelig brukt for å verifisere resultatene oppnådd fra CAM-behandling siden de begge er antibiotika som hemmer oversettelsesforlengelse38. TET ble funnet for å oppdage primær TIS, men kunne ikke avsløre intern TIS36. RET binder seg i PTC av bakteriell ribosom, og forhindrer dannelse av den første peptidbindingen ved å forstyrre en langstrakt aminoacyl-tRNA på A-området. Bruk av RET resulterer i ribosomer arrestasjon på både primære og interne TISs34. Onc112, et prolinrikt antimikrobielt peptid, binder seg i utgangstunnelen og blokkerer aminoacyl-tRNA-binding i ribosomal A-området. Som et resultat forhindrer Onc112 initieringskomplekser fra å komme inn i forlengelsesfasen37.
Hovedinformasjonen ribosomeprofilering gir er ribosomer tetthet og deres posisjon på mRNA. Det ble brukt til å undersøke differensialgenuttrykk på oversettelsesnivå under ulike vekstforhold1,6, måle translasjonell effektivitet1,38,39 og oppdage oversettelsesreguleringshendelser som ribosomal pause10. RIBO-seq gjør det også mulig å avdekke oversettelsen av kommenterte ncRNA, pseudogenes og unannotated små åpne leserammer (ORF) som fører til identifisering av nye og / eller svært korte proteinkodingsgener10,12,22,30,37. I slike tilfeller kan RIBO-seq finjustere og forbedre genommerknaden. Med sin høye følsomhet for identifisering av oversatte ORFer og dens kvantitative natur, kan ribosomprofilering også fungere som proxy for proteombestemmelsen eller i å hjelpe proteomikkstudier31,34,39. Ved å kartlegge TIS avslører ribosomprofilering N-terminalt utvidede og avkortede isoformer av kjente proteiner10,32. RIBO-seq ble også tilpasset for å studere samtidig translasjonell folding av proteiner14,21,24. Denne metoden gjør det mulig å måle forlengelseshastigheter1,10,39 eller dekodingshastigheter for individuelle codons6 og hjelper til med å utvikle kvantitative modeller av oversettelse17. Ribosomet profilering metoden er også i stand til å gi mekanistisk innsikt i ribosomet pause i bakterier7,15,17, frameshifting40, stop-codon readthrough21, terminering / resirkulering defekter41,42 og ribosomal konformasjon endringer33 i eukaryoter. RIBO-seq ble også tilpasset for å undersøke effekten av spesifikke transvirkende faktorer på oversettelse som miRNAs6 og RNA-bindende proteiner i eukaryoter16,43. Det er imidlertid viktig å erkjenne at den eksperimentelle utformingen og den oppnådde oppløsningen av RIBO-seq bestemmer mengden informasjon som kan trekkes ut fra de resulterende sekvenseringsdataene12.
Protocol
Representative Results
Discussion
Den viktigste tekniske utfordringen med ribosomet profilering er behovet for å raskt hemme oversettelse for å fange et øyeblikksbilde av ribosomer på mRNAer på en bestemt fysiologisk tilstand av interesse. For å oppnå dette brukes ofte oversettelseshemmere, rask høsting og blitsfrysing i flytende nitrogen. Påføring av antibiotika er valgfritt siden de kan forårsake artefakter. Kloramfenikol er et vanlig brukt stoff for å arrestere forlengelse av ribosomer i bakteriell RIBO-seq. Det forhindrer imidlertid ikke …
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
ALS ønsker å anerkjenne den økonomiske støtten til EMBO Installation Grants IG 3914 og POIR. 04.04.00-00-3E9C/17-00 utført i First TEAM-programmet til Foundation for Polish Science finansiert av EU under European Regional Development Fund.
Materials
| 10X TBE (powder) | Invitrogen | AM9864 | |
| 2-Mercaptoethanol, 99%, pure | Acros Organics | 125472500 | |
| Adenosine 5'-Triphosphate (ATP) | New England Biolabs | P0756S | |
| Aluminium oxide calcinated pure p.a. | Chempur | 114560600 | |
| Calcium chloride dihydrate | Sigma-Aldrich | C3881-500G | |
| Chloramphenicol | MP Biomedicals | 190321 | |
| DNA Clean & Concentrator -5 | Zymo Research | D4004 | |
| Dnase I recombinant, Rnase-free | Roche | 4716728001 | |
| EDTA disodium salt | Fisher Scientific | E/P140/48 | |
| Ethyl Alcohol Absolut 99,8% Pure-P.A.-Basic | POCH Avantor Performance Materials Poland S.A | BA6480111 | |
| Filtration apparatus | VWR Collection | 511-0265 | all-glass filtration apparatus, with funnel, fritted base, cap, 47 mm Ø spring clamp and ground joint flask |
| Gel 40 (19:1) | Rotiphorese | 3030.1 | |
| Gel Loading Dye, Blue, 6X | New England Biolabs | E6138G | |
| Guanosine 5′-[β,γ-imido]triphosphate trisodium salt hydrate | Sigma-Aldrich | G0635-25MG | |
| labZAP | A&A Biotechnology | 040-500 | |
| Magnesium acetate tetrahydrate | Sigma-Aldrich | M5661-250G | |
| MCE membrane fiter | Alfatec Technology | M47MCE45GWS | pore size: 0.45um |
| MICROBExpress Bacterial mRNA Purification | Invitrogen | AM1905 | |
| Multiplex Small RNA Library Prep Set for Illumina | New England Biolabs | E7300S | |
| Nuclease-Free Water | Ambion | AM9937 | |
| Potassium Acetate Anhydrous Pure P.A. | POCH Avantor Performance Materials Poland S.A | 744330113 | |
| Quick-Load pBR322 DNA-MspI Digest | New England Biolabs | E7323A | |
| RNA Clean & Concentrator -25 | Zymo Research | R1018 | |
| Sodium acetate | Sigma-Aldrich | S2889-250G | |
| Sodium carbonate | Sigma-Aldrich | 223530-500G | |
| Sodium hydrogen carbonate pure p.a. | POCH Avantor Performance Materials Poland S.A | 810530115 | |
| SYBR Gold nucleic acid gel stain | Life Technologies | S11494 | |
| T4 Polynucleotide Kinase | New England Biolabs | M0201L | |
| T4 Polynucleotide Kinase Reaction Buffer | New England Biolabs | B0201S | |
| TBE-Urea Sample Buffer (2x) | Invitrogen | LC6876 | |
| Tris(hydroxymethyl)amino-methane, ultrapure, 99,9% | AlfaAesar | J65594 | |
| Triton X-100, 98% | Acros Organics | 327371000 | |
| Urea G.R. | lach:ner | 40096-AP0 |
Riferimenti
- Ingolia, N. T., Ghaemmaghami, S., Newman, J. R., Weissman, J. S. Genome-wide analysis in vivo of translation with nucleotide resolution using ribosome profiling. Science. 324 (5924), 218-223 (2009).
- Takanami, M., Yan, Y., Jukes, T. H. Studies on the site of ribosomal binding of f2 bacteriophage RNA. Journal of Molecular Biology. 12 (3), 761-773 (1965).
- Steitz, J. A. Polypeptide Chain Initiation: Nucleotide Sequences of the Three Ribosomal Binding Sites in Bacteriophage R17 RNA. Nature. 224 (5223), 957-964 (1969).
- Wolin, S. L., Walter, P. Ribosome pausing and stacking during translation of a eukaryotic mRNA. The EMBO journal. 7 (11), 3559-3569 (1988).
- Argüello, R. J., et al. SunRiSE – measuring translation elongation at single-cell resolution by means of flow cytometry. Journal of Cell Science. 131 (10), 214346 (2018).
- Michel, A. M., Baranov, P. V. Ribosome profiling: a Hi-Def monitor for protein synthesis at the genome-wide scale. Wiley Interdisciplinary Reviews: RNA. 4 (5), 473-490 (2013).
- Woolstenhulme, C. J., Guydosh, N. R., Green, R., Buskirk, A. R. High-precision analysis of translational pausing by ribosome profiling in bacteria lacking EFP. Cell reports. 11 (1), 13-21 (2015).
- McGlincy, N. J., Ingolia, N. T. Transcriptome-wide measurement of translation by ribosome profiling. Methods (San Diego, Calif). 126, 112-129 (2017).
- Gerashchenko, M. V., Gladyshev, V. N. Translation inhibitors cause abnormalities in ribosome profiling experiments. Nucleic acids research. 42 (17), 134 (2014).
- Ingolia, N. T., Lareau, L. F., Weissman, J. S. Ribosome Profiling of Mouse Embryonic Stem Cells Reveals the Complexity of Mammalian Proteomes. Cell. 147 (4), 789-802 (2011).
- Ingolia, N. T., Brar, G. A., Rouskin, S., McGeachy, A. M., Weissman, J. S. The ribosome profiling strategy for monitoring translation in vivo by deep sequencing of ribosome-protected mRNA fragments. Nature protocols. 7 (8), 1534-1550 (2012).
- Hsu, P. Y., et al. Super-resolution ribosome profiling reveals unannotated translation events in Arabidopsis. Proceedings of the National Academy of Sciences of the United States of America. 113 (45), 7126-7135 (2016).
- Blanchard, S. C., Kim, H. D., Gonzalez, R. L., Puglisi, J. D., Chu, S. tRNA dynamics on the ribosome during translation. Proceedings of the National Academy of Sciences of the United States of America. 101 (35), 12893-12898 (2004).
- Becker, A. H., Oh, E., Weissman, J. S., Kramer, G., Bukau, B. Selective ribosome profiling as a tool for studying the interaction of chaperones and targeting factors with nascent polypeptide chains and ribosomes. Nature protocols. 8 (11), 2212-2239 (2013).
- Li, G. W., Oh, E., Weissman, J. S. The anti-Shine-Dalgarno sequence drives translational pausing and codon choice in bacteria. Nature. 484 (7395), 538-541 (2012).
- King, H. A., Gerber, A. P. Translatome profiling: methods for genome-scale analysis of mRNA translation. Briefings in functional genomics. 15 (1), 22-31 (2014).
- Mohammad, F., Woolstenhulme, C. J., Green, R., Buskirk, A. R. Clarifying the Translational Pausing Landscape in Bacteria by Ribosome Profiling. Cell reports. 14 (4), 686-694 (2016).
- Reid, D. W., Shenolikar, S., Nicchitta, C. V. Simple and inexpensive ribosome profiling analysis of mRNA translation. Methods (San Diego, Calif). 91, 69-74 (2015).
- Sanz, E., Yang, L., Su, T., Morris, D. R., McKnight, G. S., Amieux, P. S. Cell-type-specific isolation of ribosome-associated mRNA from complex tissues. Proceedings of the National Academy of Sciences of the United States of America. 106 (33), 13939-13944 (2009).
- Petrova, O. E., Garcia-Alcalde, F., Zampaloni, C., Sauer, K. Comparative evaluation of rRNA depletion procedures for the improved analysis of bacterialbiofilm and mixed pathogen culture transcriptomes. Scientific Reports. 7, 41114 (2017).
- Oh, E. Selective ribosome profiling reveals the cotranslational chaperone action of trigger factor in vivo. Cell. 147 (6), 1295-1308 (2011).
- Chew, G. L. Ribosome profiling reveals resemblance between long non-coding RNAs and 5′ leaders of coding RNAs. Development. 140 (13), 2828-2834 (2013).
- Dunn, J. G., Foo, C. K., Belletier, N. G., Gavis, E. R., Weissman, J. S. Ribosome profiling reveals pervasive and regulated stop codon readthrough in Drosophila melanogaster. eLife. 2, 01179 (2013).
- Liu, M. J., et al. Translational landscape of photomorphogenic Arabidopsis. The Plant cell. 25 (10), 3699-3710 (2013).
- Rooijers, K., Loayza-Puch, F., Nijtmans, L. G., Agami, R. Ribosome profiling reveals features of normal and disease-associated mitochondrial translation. Nature Communications. 4, 2886 (2013).
- Danielle, L., et al. Fidelity of translation initiation is required for coordinated respiratory complex assembly. Science Advances. 5 (12), 2118 (2019).
- Zoschke, R., Watkins, K. P., Barkan, A. A rapid ribosome profiling method elucidates chloroplast ribosome behavior in vivo. The Plant cell. 25 (6), 2265-2275 (2013).
- Chotewutmontri, P., Barkan, A. Dynamics of chloroplast translation during chloroplast differentiation in maize. PLoS genetics. 12 (7), 1006106 (2016).
- Lee, S., Liu, B., Huang, S. X., Shen, B., Qian, S. B. Global mapping of translation initiation sites in mammalian cells at single-nucleotide resolution. Proceedings of the National Academy of Sciences of the United States of America. 109 (37), 2424-2432 (2012).
- Martinez, T. F., Chu, Q., Donaldson, C., Tan, D., Shokhirev, M. N., Saghatelian, A. Accurate annotation of human protein-coding small open reading frames. Nature chemical biology. 16 (4), 458-468 (2020).
- Simsek, D., et al. The Mammalian Ribo-interactome Reveals Ribosome Functional Diversity and Heterogeneity. Cell. 169 (6), 1051-1065 (2017).
- Fritsch, C., et al. Genome-wide search for novel human uORFs and N-terminal protein extensions using ribosomal footprinting. Genome research. 22 (11), 2208-2218 (2012).
- Lareau, L. F., Hite, D. H., Hogan, G. J., Brown, P. O. Distinct stages of the translation elongation cycle revealed by sequencing ribosome-protected mRNA fragments. eLife. 3, 01257 (2014).
- Meydan, S., et al. Retapamulin-Assisted Ribosome Profiling Reveals the Alternative Bacterial Proteome. Molecular Cell. 74 (3), 481-493 (2019).
- Wilson, D. N. The A-Z of bacterial translation inhibitors. Critical Reviews in Biochemistry and Molecular Biology. 44, 393-433 (2009).
- Nakahigashi, K. Comprehensive identification of translation start sites by tetracycline-inhibited ribosome profiling. DNA research : an international journal for rapid publication of reports on genes and genomes. 23 (3), 193-201 (2016).
- Weaver, J., Mohammad, F., Buskirk, A. R., Storz, G. Identifying Small Proteins by Ribosome Profiling with Stalled Initiation Complexes. mBio. 10 (2), 02819 (2019).
- Nakahigashi, K. Effect of codon adaptation on codon-level and gene-level translation efficiency in vivo. BMC genomics. 15 (1), 1115 (2014).
- Li, G. W., Burkhardt, D., Gross, C., Weissman, J. S. Quantifying absolute protein synthesis rates reveals principles underlying allocation of cellular resources. Cell. 157 (3), 624-635 (2014).
- Michel, A. M., et al. Observation of dually decoded regions of the human genome using ribosome profiling data. Genome research. 22 (11), 2219-2229 (2012).
- Guydosh, N. R., Green, R. Dom34 rescues ribosomes in 3′ untranslated regions. Cell. 156 (5), 950-962 (2014).
- Young, D. J., Guydosh, N. R., Zhang, F., Hinnebusch, A. G., Green, R. Rli1/ABCE1 Recycles Terminating Ribosomes and Controls Translation Reinitiation in 3’UTRs In Vivo. Cell. 162 (4), 872-884 (2015).
- Ingolia, N. T. Ribosome profiling: new views of translation, from single codons to genome scale. Nature Reviews Genetics. 15 (3), 205-213 (2014).
- Dobin, A., et al. ultrafast universal RNA seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- . . Babraham Bioinformatics. Fast QC. , (2021).
- Gerashchenko, M. V., Gladyshev, V. N. Ribonuclease selection for ribosome profiling. Nucleic acids research. 45 (2), 6 (2017).
- Verbruggen, S., Menschaert, G. mQC: A post-mapping data exploration tool for ribosome profiling. Computer Methods and Programs in Biomedicine. 181, 104806 (2019).
- Cui, H., Hu, H., Zeng, J., Chen, T. DeepShape: estimating isoform-level ribosome abundance and distribution with Ribo-seq data. BMC bioinformatics. 20, 678 (2019).
- Choi, J. . RiboToolkit: an integrated platform for analysis and annotation of ribosome profiling data to decode RNA translation at codon resolution. , (2021).
- Choi, J. Dynamics of the context-specific translation arrest by chloramphenicol and linezolid. Nature chemical biology. 16 (3), 310-317 (2020).
- Tompson, J., O’Connor, M., Mills, J. A., Dahlberg, A. E. The protein synthesis inhibitors, oxazolidinones and chloramphenicol, cause extensive translational inaccuracy in vivo. Journal of Molecular Biology. 322 (2), 273-279 (2002).
- Brar, G. A., Weissman, J. S. Ribosome profiling reveals the what, when, where and how of protein synthesis. Nature reviews. Molecular cell biology. 16 (11), 651-664 (2015).
- Glaub, A., Huptas, C., Neuhaus, K., Ardern, Z. Recommendations for bacterial ribosome profiling experiments based on bioinformatic evaluation of published data. Journal of Biological Chemistry. 295, 8999-9011 (2020).
- Sorour, M. H., Hani, H. A., Shaalan, H. F., El-Sayed, M. M. H. Experimental screening of some chelating agents for calcium and magnesium removal from saline solutions. Desalination and Water Treatment. 57 (48-49), 22799-22808 (2015).
- Mohammad, F., Green, R., Buskirk, A. R. A systematically-revised ribosome profiling method for bacteria reveals pauses at single-codon resolution. Elife. 8, 42591 (2019).
- O’Connor, P. B., Li, G. W., Weissman, J. S., Atkins, J. F., Baranov, P. V. rRNA:mRNA pairing alters the length and the symmetry of mRNA-protected fragments in ribosome profiling experiments. Bioinformatics. 29 (12), 1488-1491 (2013).
- Protocol for RNA Clean & Concentrator -25. Zymo Research Available from: https://files.zymoresearch.com/protocols/_r1017_r1018_rna_clean_concentrator-25.pdf (2021)
- Protocol for use with NEBNext Small RNA Library Prep Set for Illumina (E7300, E7580, E7560, E7330). New England Biolabs Available from: https://www.international.neb.com/protocols/2018/03/27/protocol-for-use-with-nebnext-small-rna-library-prep-set-for-illumina-e7300-e7580-e7560-e7330 (2021)

