멤브레인 운송은 단일 단백질 해상도에서 고도의 병렬 나노 기공 칩 시스템에 의해 분석 처리합니다
Summary
The presented protocol describes the analysis of membrane protein mediated transport on the single transporter level using pore-spanning solvent-free lipid bilayers. This is achieved by the creation of bulk produced nanopore array chips, combined with highly parallel data acquisition and analysis, enabling the future establishment of membrane protein effector screenings.
Abstract
전좌 기판 비 electrogenic 경우 단일 단백질 수준에서 세포막 단백질 수송 여전히 상세한 분석을 기피한다. 상당한 노력이 분야에서 제조되었지만, 막 수송의 분석에 필요한 무용제 지질 이중층 기술과 조합하여 높은 처리량 자동 반송 분석을 가능 기술들은 드물다. 운송의이 클래스는 그러나 세포 항상성에 중요한 따라서 신약 개발과 새로운 통찰력을 얻을 수있는 방법론의 주요 대상이 절실히 필요하다.
여기에 제시된 논문은 단일 트랜스 해상도 세포막 단백질 매개 수송 과정의 분석을위한 신규 한 바이오칩의 확립 및 처리를 설명. 바이오칩은 설계에서 매우 평행하고 산업 등급과 양으로 제조 할 수 나노 기공으로 둘러싸인 미세 공동으로 구성된다. 단백질 형질 리포좀에 직접 적용 할 수있다SSM-기술을 이용하여 자기 조립 기공 걸친 지질 이중층을 구성하는 칩면 (고체 지질 막을 지원됨). 막의 일부가 자립으로 또는 실시간 다중 분광 형광 판독 하였다 가능 캐비티 공간에서 기판 전위에 대한 인터페이스를 제공하는 세공은 스패닝. 표준 작업 절차 (SOP)의 설립은 기능적으로 재구성 될 수있는 거의 모든 막 단백질 칩 표면에 단백질 형질 지질 이중층의 간단한 구축을 허용한다. 유일한 필수 비 electrogenic 기판 반송 용 형광 판독 시스템의 구축이다.
하이 콘텐츠 스크리닝 응용 병렬 다중 칩 녹화 자동 반전 된 형광 현미경을 사용하여 accomplishable이다. 큰 데이터 세트는 자유롭게 사용할 맞춤 설계 해석 소프트웨어를 사용하여 분석 될 수있다. 세 가지 색 멀티 스펙트럼 형광판독 또한 거짓 긍정적 인 결과를 제거, 다른 이벤트 클래스로 편견 데이터 차별을 허용합니다.
칩 기술은 현재의 SiO2 표면을 기반으로하지만, 금 코팅 칩 표면을 사용하여 추가로 작용하는 것도 가능하다.
Introduction
막 단백질의 분석은 지난 20 년 동안 기본 및 제약 연구에 대한 관심이 증가되고있다. 신약 개발은 현재 제한 요인 중 하나 인, 식별 및 새로운 목표의 상세한 특성에 의존한다. 모든 약물 표적의 약 60 %는 단백질 막 한 사실이 기술의 개발은 그 기능이 가장 중요 규명 할 수있다.
4 – 과거 electrogenic 채널 및 수송을 연구하는 기술이 다수 개발되었다. 반면에 비 electrogenic 기판은 더 어려운 과제를 제시한다. 그들은 캐스케이드 5 시그널링 키 수용체로서 세포막 기능 및 전체 용질 및 영양소의 유동을 제어하는대로 그러나 프라임 약물 표적으로서 특별한 관심이다.
상당한 노력이 t의 개발에 투입하고있다echniques은 막 수송 단백질 (6), (7)의 기능을 연구한다. 몇 가지 이름 고체 지원 지질 이중층, 닿는 이중층 11, 12, microblack 지질 막 13, 14 및 기본 소포 어레이 (15, 16)을 포함하여 10 일 – 고체 지원 멤브레인을 사용하는 시스템은이 분야 8로 가장 유망한 도구가 등장했다. 그들 중 일부는 상업 설정 17, 18으로도 사용할 수 있습니다. 몇 가지 예는 고도의 병렬 14, 19, 심사 애플리케이션을위한 전제 조건에서 하나의 막 단백질을 연구 할 수있는 능력을 결합 발표되었다. 그러나 이러한 방법은 거의 산업 환경에 대한 기초 연구에서 다리 없다. 어려움은 종종 자동화가 될 수있는 시스템의 능력, 비용 집약적 생산 및 / 또는 준비에 수고 거짓말. 접근 오위에서 언급 한 모든 장애물을 vercoming하는 최종 목표이다.
22 – 여기에 제시된 기술은 단일 단백질 수준 (20)에 제어 된 환경에서 시험 관내에서 막 채널 수송을 연구하기 위해 개발되었다. 26 검은 지질 막 (27) – LUVs로 정제 막 단백질의 재구성 훨씬 더 GUVs (23)에 대한 비교 방법보다 설정됩니다. 이들은 직접 이중층의 형성이 자기 조립 과정을 통해 이루어지고, 칩 표면에 적용될 수있다. 나노 다공성 칩 (도. 1)의 유리 바닥 디자인은 시스템의 간단한 자동화 허용 공기 현미경을 허용한다. 전동 스테이지와 함께 다중 칩 분석 밀봉 공동 수천 함유 뷰의 각 필드와 동시에 측정 될 수있다.
<p class="jove_content" fo:keep-together.within-page= "1">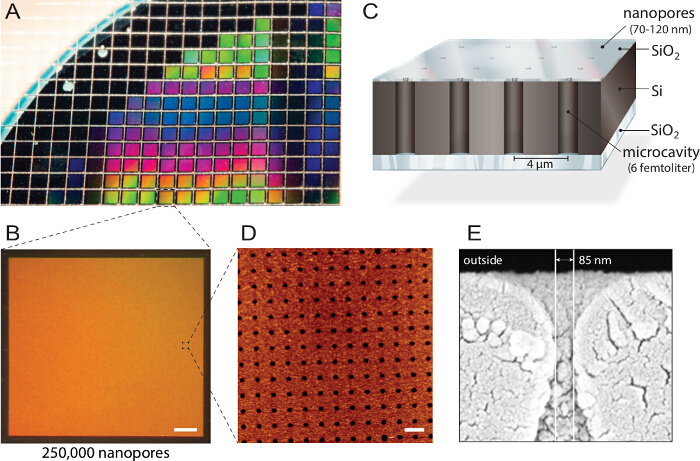
그림 1. 다중화 된 나노 기공 바이오칩의 설계. A)는 실리콘 – 온 – 인슐레이터 (SOI) 웨이퍼에 반응성 이온 에칭에 의해 구성된다. 약 1,150 개별 칩은 동일한 특성 및 품질. B) 각각의 칩은 나노 구멍에 25 만 개별 마이크로 공동을 포함하는 각각의 웨이퍼에서 제조된다. 스케일 바 :. 200 μm의 C) 각 공동 멀티 스펙트럼 형광 판독을 통해 주소입니다. 반전 된 형광 현미경. D) 원 자간 력 현미경으로 바이오칩 호환 만들기는 불투명 한 상부층 블록 버퍼 탱크에서 형광 신호 (AFM) 촬상 균등 (세공 개구 3.6 nm 인 이산화 규소 층의 표면 거칠기를 배치 계시 N = 소포 융합 40) 최적. 스케일 바 :. 5 μm의 E) 주사 전자 마이크roscopy (SEM) 이미지는 실리콘 칩 내부 femtoliter 캐비티에 대한 액세스를 허용 나노 기공을 통한 단면을 도시한다. 이 그림은 (21)로부터 허가를 재사용했다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
모든 데이터 분석은 최종 사용자가 제한되지 않은 액세스를 실현 프리웨어를 사용하여 수행된다. 시계열없는 화상 처리 소프트웨어 맞춤 제작 곡선 분석 소프트웨어 있도록 배치 프로세싱 곡선 여러 형광 채널 수천 대용량 데이터의 직접적인 상관 관계를 이용하여 분석 하였다.
이 프로토콜에서 사용되는 모델 단백질은 E. 유래의 컨덕턴스 (후 MsCl) 채널 단백질의 채널이다 mechanosensitive 대장균. 그것은 본질적으로 삼투압 충격을 해제하기위한 밸브로서 기능하지만 SYNT 합리적으로 설계하는 방식으로 수정hetic 기능은 공유 채널 수축 측에 부착 될 수있다. 나노 밸브를 만드는 채널이 열 트리거되는 공유 결합 활성제 (MTSET)의 전하 반발, 비아. 이온, 물, 작은 단백질뿐만 아니라, 작은 형광체 같은 작은 분자는 채널을 통해 투과 할 수있다. 여기서, 단백질은 단백질 – 매개 전위를 검출하기위한 시스템의 능력을 증명하기위한 모델로서 사용된다.
Protocol
Representative Results
Discussion
여기에 제시된 기술은 막 단백질 수송의 고도의 병렬 분석을 할 수 있습니다. 재구성 세포막 단백질 시스템은 직접 이론적마다 막 수송 체의 적응을 가능 또는 채널은 바이오 칩에 적용될 수있다. 전송 분석은 직접 형광 변화 (형광의 전위 또는 형광 표지 된 기판) 또는 간접 형광 변화 (pH 민감성 염료, 보조 효소 반응)을 통해 중, 형광 판독 시스템의 구축에 의해 제한된다. 후자는 그러나 아직 확?…
Disclosures
The authors have nothing to disclose.
Acknowledgements
We thank Barbara Windschiegl for her help in establishing SOPs; Dennis Remme for his work on the NanoCalcFX software and Alina Kollmannsperger, Markus Braner and Milan Gerovac for helpful suggestions on the manuscript. The German-Israeli Project Cooperation (DIP) provided by the DFG and the Federal Ministry of Education and Research to R.T., as well as the Federal Ministry of Economics and Technology (ZIM R&D Project) to R.T. and Nanospot GmbH supported this work.
Materials
| Reagent | |||
| [2-(Trimethylammonium)ethyl] methanethiosulfonate |
Toronto Research Chemicals Inc. | T792900 | MTSET; hydrolized by water. Keep as dry pouder aliquot at -80 °C. Use immediately (30 minutes) after solubilization in buffer. |
| 1 ml gas-tight syringe | Hamilton | #1001 | |
| 10 ml round flask | Schott Duran | ||
| 2.7 mm glas beads | Roth | N032.1 | |
| 2-Propanole | Roth | 9866.5 | |
| 30 cm Luer-Lock Extension Tube | Sarstedt | 744304 | |
| Acetone | Roth | 5025.5 | |
| Bio-Beads SM-2 Adsorbent | Bio-Rad | 152-3920 | need to be activated before first use |
| CaCl2 Dihydrat | Roth | HN04.3 | |
| Calcein | Sigma | C0875 | store dark at -20 °C |
| Chloroform reagent grade | VWR Chemicals | 22711324 | |
| DOPEATTO390 | ATTO-TEC | AD 390-165 | store dark at -20 °C |
| Ethanol absolute | Sigma-Aldrich | 32205 | |
| Injekt Single-use syringe | Braun | 460 60 51V | |
| Injekt-F single-use syringe | Braun | 91 66 017V | |
| Keck clips | Schott | KC29 | |
| L-α-Phosphatidylcholine, 20% (Soy) | Avanti Polar Lipids | 5416016 | store under inert gas at -20 °C |
| NaCl 99.5% p.a. | Roth | 3957.2 | |
| Nanopore E100 wafer/chips | Micromotive (Mainz/Germany) | available on request | |
| Nucleopore Track-Etch Membrane 0.4 µm | Whatman | 800282 | |
| Oregon Green Dextran 488 (70 kDa) | life Technologies | D-7173 | store dark at -20 °C |
| Oy647 | Luminartis (Münster/Germany) | OY-647-T-1mg | store dark at -20 °C |
| Rotilabo-syringe filtration, unsterile, pore-size 0.22 µm | Roth | P 818.1 | |
| Sephadex G-50 | Sigma-Aldrich | G5080 | column material for size exclusion chromatography |
| Silastic MDX4-4210 | Dow Corning | curing agent for chip fixation onto cover glass support | |
| sticky-Slide 8-well | ibidi | 80828 | multi-well chamber for the mounting onto glass slides (chip holder) |
| Three-way stopcock blue | Sarstedt | 744410001 | |
| Tris Pufferan 99.9% Ultra Quality | Roth | 5429.2 | |
| Triton-X 100 | Roth | 6683.1 | |
| Whatman 0.2 µm cellulose nitrate membrane filter | Roth | NH69.1 | |
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| Büchi 461 water bath | Büchi | ||
| Büchi Rotavapor RE 111 | Büchi | ||
| Cary Eclipse Fluorescence Spectrophtometer | Varian | ||
| LiposoFast Mini Extruder | Avestin | ||
| Membrane pump | Vaccubrand | 15430 | |
| Nanosight Nanoparticle Tracking Microscope | Malvern / Nanosight | LM 14C | |
| NyONE microscope | Synentec | available on request | |
| Pump control | Vaccubrand | CVC 2II | |
| Sonicator bath Sonorex RK100H | Brandelin electronic | 31200001107477 | |
| Vaccum pump RC5 | Vaccubrand | 1805400204 | |
| Water bath W13 | Haake | 002-9910 | |
| Plasma Cleaner PDC-37G | Harrick Plasma | PDC-37G | |
| Name | Company | Catalog Number | Comments |
| Software | |||
| ImageJ | Open Source | http://imagej.nih.gov/ij/ | scientific image processing software |
| NanoCalcFX | Freeware | http://sourceforge.net/projects/nanocalc/ | data analysis/evaluation software for massive transport kinetic datasets |
| NTA 2.3 Analytical Software | Nanosight | data acquisition and analysis software for nanoparticle tracking microscope | |
| NTA 2.3 Temperature Comms | Nanosight | temperature controle software for nanoparticle tracking microscope |
References
- Yildirim, M. A., Goh, K. I., Cusick, M. E., Barabasi, A. L., Vidal, M. Drug-target network. Nat Biotechnol. 25, 1119 (2007).
- Hamill, O. P., Marty, A., Neher, E., Sakmann, B., Sigworth, F. Improved patch-clamp techniques for high-resolution current recording from cells and cell-free membrane patches. Pflugers Arch. 391, 85-100 (1981).
- Osaki, T., Suzuki, H., Le Pioufle, B., Takeuchi, S. Multichannel simultaneous measurements of single-molecule translocation in α-hemolysin nanopore array. Anal. Chem. 81, 9866-9870 (2009).
- Carrillo, L., et al. High-resolution membrane capacitance measurements for studying endocytosis and exocytosis in yeast. Traffic. , (2015).
- Giacomini, K. M., et al. Membrane transporters in drug development. Nat. Rev. Drug Discov. 9, 215-236 (2010).
- Castell, O. K., Berridge, J., Wallace, M. I. Quantification of membrane protein inhibition by optical ion flux in a droplet interface bilayer array. Angew. Chem. Int. Ed. 51, 3134-3138 (2012).
- Zollmann, T., et al. Single liposome analysis of peptide translocation by the ABC transporter TAPL. Proc. Natl. Acad. Sci. U.S.A. 112, 2046-2051 (2015).
- Tamm, L. K., McConnell, H. M. Supported phospholipid bilayers. Biophys. J. 47, 105-113 (1985).
- Sackmann, E. Supported membranes: scientific and practical applications. Science. 271, 43-48 (1996).
- Castellana, E. T., Cremer, P. S. Solid supported lipid bilayers: From biophysical studies to sensor design. Surf. Sci. Rep. 61, 429-444 (2006).
- Wagner, M. L., Tamm, L. K. Tethered polymer-supported planar lipid bilayers for reconstitution of integral membrane proteins: silane-polyethyleneglycol-lipid as a cushion and covalent linker. Biophys. J. 79, 1400-1414 (2000).
- Naumann, C. A., et al. The polymer-supported phospholipid bilayer: tethering as a new approach to substrate-membrane stabilization. Biomacromolecules. 3, 27-35 (2002).
- Weiskopf, D., Schmitt, E. K., Klühr, M. H., Dertinger, S. K., Steinem, C. Micro-BLMs on highly ordered porous silicon substrates: Rupture process and lateral mobility. Langmuir. 23, 9134-9139 (2007).
- Watanabe, R., et al. Arrayed lipid bilayer chambers allow single-molecule analysis of membrane transporter activity. Nat. Commun. 5, (2014).
- Stamou, D., Duschl, C., Delamarche, E., Vogel, H. Self-Assembled Microarrays of Attoliter Molecular Vessels. Angew. Chem. Int. Ed. 115, 5738-5741 (2003).
- Lohr, C., et al. Single Liposomes Used to Study the Activity of Individual Transporters. Biophysical Journal. 106, 229a (2014).
- Dunlop, J., Bowlby, M., Peri, R., Vasilyev, D., Arias, R. High-throughput electrophysiology: an emerging paradigm for ion-channel screening and physiology. Nat. Rev. Drug Discov. 7, 358-368 (2008).
- Milligan, C. J., et al. Robotic multiwell planar patch-clamp for native and primary mammalian cells. Nat. Protoc. 4, 244-255 (2009).
- Soga, N., Watanabe, R., Noji, H. Attolitre-sized lipid bilayer chamber array for rapid detection of single transporters. Sci. Rep. 5, (2015).
- Kleefen, A., et al. Multiplexed parallel single transport recordings on nanopore arrays. Nano Lett. 10, 5080-5087 (2010).
- Wei, R., Gatterdam, V., Wieneke, R., Tampé, R., Rant, U. Stochastic sensing of proteins with receptor-modified solid-state nanopores. Nat. Nanotechnol. 7, 257-263 (2012).
- Urban, M., et al. Highly parallel transport recordings on a membrane-on-nanopore chip at single molecule resolution. Nano Lett. 14, 1674-1680 (2014).
- Kusters, I., Van Oijen, A. M., Driessen, A. J. Membrane-on-a-Chip: Microstructured Silicon/Silicon-Dioxide Chips for High-Throughput Screening of Membrane Transport and Viral Membrane Fusion. ACS Nano. 8, 3380-3392 (2014).
- Hansen, J. S., Thompson, J. R., Hélix-Nielsen, C., Malmstadt, N. Lipid directed intrinsic membrane protein segregation. J. Am. Chem. Soc. 135, 17294-17297 (2013).
- Heinemann, F., Schwille, P. Preparation of Micrometer-Sized Free-Standing Membranes. ChemPhysChem. 12, 2568-2571 (2011).
- Lazzara, T. D., Carnarius, C., Kocun, M., Janshoff, A., Steinem, C. Separating attoliter-sized compartments using fluid pore-spanning lipid bilayers. ACS nano. 5, 6935-6944 (2011).
- Winterhalter, M. Black lipid membranes. Curr. Opin. Colloid Interface Sci. 5, 250-255 (2000).
- Koçer, A., Walko, M., Feringa, B. L. Synthesis and utilization of reversible and irreversible light-activated nanovalves derived from the channel protein MscL. Nat. Protoc. 2, 1426-1437 (2007).

