Membrana de transporte de procesos analizados por un sistema de nanoporos viruta altamente paralela a la Resolución de la proteína individual
Summary
The presented protocol describes the analysis of membrane protein mediated transport on the single transporter level using pore-spanning solvent-free lipid bilayers. This is achieved by the creation of bulk produced nanopore array chips, combined with highly parallel data acquisition and analysis, enabling the future establishment of membrane protein effector screenings.
Abstract
Membrana de transporte de proteínas en el nivel de proteína única sigue evade análisis detallado, si el sustrato es translocado no electrógeno. considerables esfuerzos se han hecho en este campo, pero las técnicas que permiten el análisis automatizado de transporte de alto rendimiento en combinación con técnicas de bicapa de lípidos libres de disolvente necesarias para el análisis de los transportadores de membrana son raros. Esta clase de transportadores sin embargo, es crucial en la homeostasis celular y, por tanto, un objetivo clave en el desarrollo de fármacos y metodologías para obtener nuevos conocimientos necesita desesperadamente.
El manuscrito que aquí se presenta describe el establecimiento y manejo de un nuevo biochip para el análisis de proteínas mediada por procesos de transporte de membrana a una resolución de transportador sola. El biochip se compone de microcavidades encerradas por nanoporos que es altamente paralelo en su diseño y se pueden producir en grado industrial y cantidad. liposomas de proteínas que albergan directamente se pueden aplicar ala superficie del chip de formación de poros que abarca bicapas lipídicas auto-ensambladas utilizando técnicas SSM-(sólido apoyado membranas lipídicas). Pore-que abarca partes de la membrana son independientes, que proporciona la interfaz para la translocación de sustrato en o fuera del espacio de la cavidad, que puede ser seguido por la lectura fluorescente multiespectral en tiempo real. El establecimiento de procedimientos normalizados de trabajo (PNT) permite la creación directa de bicapas de lípidos proteínas albergar sobre la superficie del chip de prácticamente todas las proteínas de membrana que puede ser reconstituida funcionalmente. El único requisito previo es el establecimiento de un sistema de lectura de fluorescencia para sustratos de transporte no electrógenos.
aplicaciones de alto contenido de cribado son realizable por el uso de microscopios fluorescentes invertidas automatizados de grabación chips múltiples en paralelo. grandes conjuntos de datos pueden ser analizados utilizando el software de análisis de diseño personalizado de libre disposición. Tres colores fluorescentes múltiples espectralde lectura, además, permite la discriminación de datos imparcial en diferentes clases de eventos, la eliminación de resultados falsos positivos.
La tecnología de chip se basa actualmente en SiO2 superficies, pero funcionalización adicional el uso de superficies de chips recubiertas de oro también es posible.
Introduction
El análisis de las proteínas de la membrana ha sido de creciente interés para la investigación básica y farmacéutica en los últimos 20 años. El desarrollo de nuevos fármacos depende de la identificación y caracterización detallada de nuevas dianas, siendo en la actualidad uno de los factores limitantes. El hecho de que alrededor del 60% de todas las dianas farmacológicas son proteínas de membrana 1, hace que el desarrollo de técnicas para dilucidar su función más importante.
En el pasado, las técnicas para el estudio de los canales electrógenos y transportadores se han desarrollado multitud 2-4. Los substratos no electrógenos en contrario presentan una tarea más difícil. Sin embargo, son de especial interés como dianas de fármacos de primera, ya que controlan el flujo de solutos y nutrientes a través de la membrana celular y la función como receptores clave en las cascadas de señalización 5.
Un esfuerzo considerable se ha puesto en el desarrollo de techniques para estudiar la función de las proteínas de transporte de membrana 6, 7. Los sistemas que utilizan membranas soporte sólido han surgido como herramientas más prometedoras en este campo 8 – 10, incluyendo las bicapas lipídicas de soporte sólido, bicapas atados 11, 12, membranas lipídicas microblack 13, 14 y nativas matrices de vesículas 15, 16 para nombrar unos pocos. Algunos de ellos son incluso disponibles como configuraciones comerciales 17, 18. Algunos ejemplos se han publicado la combinación de la capacidad para estudiar las proteínas de membrana individuales de una manera altamente paralelo 14, 19, un requisito previo para aplicaciones de cribado. Sin embargo, estos métodos raramente puente desde la investigación básica hasta el entorno industrial. Las dificultades se encuentran a menudo en la capacidad del sistema para ser automatizable, la producción de alto coste y / o preparación laboriosa. Un enfoque overcoming todos los obstáculos mencionados anteriormente es el objetivo final.
La técnica que aquí se presenta se ha desarrollado para estudiar los canales de membrana y transportadores in vitro en un ambiente controlado en el nivel de proteína única 20 – 22. La reconstitución de proteínas de membrana purificadas en las LUV se estableció mucho más que los enfoques comparables para GUVs 23 – 26 o lípidos negro membranas 27. Ellos se pueden aplicar directamente a la superficie del chip, donde la formación de bicapa está llevando a cabo a través de un proceso de autoensamblaje. El diseño de fondo de vidrio del chip nanoporoso (Fig. 1) permite la microscopía de aire, que permite la automatización sencilla del sistema. En combinación con una etapa motorizada múltiples chips pueden ser medidas al mismo tiempo, con cada campo de visión que contiene miles de cavidades selladas para el análisis.
<p class="jove_content" fo:keep-together.within-page= "1">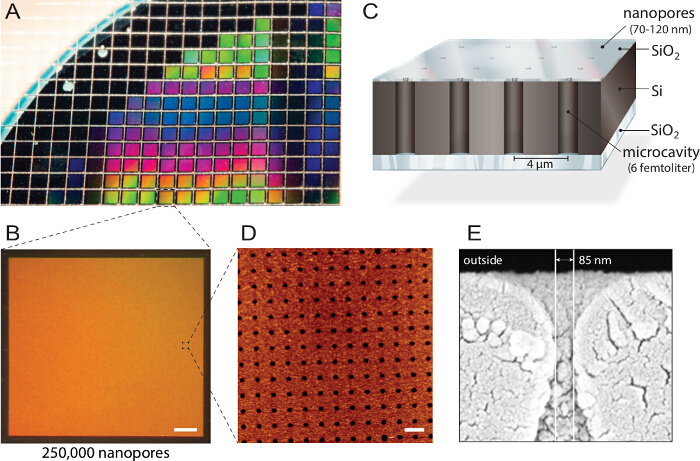
Figura 1. Diseño de biochips nanoporos multiplexados. A) A de silicio sobre aislante (SOI) de la oblea está estructurada por ataque de iones reactivos. Aproximadamente 1.150 fichas individuales se fabrican a partir de cada oblea con idénticas propiedades y calidad. B) Cada chip comprende 250.000 microcavidades individuales con aberturas nano. Barra de escala:. 200 micras C) Cada cavidad es direccionable a través de fluorescencia multiespectral de lectura. Un bloques de la capa superior carentes de las señales fluorescentes desde el depósito de regulación, haciendo que el biochip compatible con los microscopios de fluorescencia invertido. D) microscopía de fuerza atómica (AFM de imágenes) revela uniformemente dispuestas aberturas de los poros y rugosidad de la superficie de la capa de dióxido de silicio de 3,6 nm (n = 40) óptimo para la fusión de vesículas. Barra de escala:. 5 micras E) de micro electrónica de barridoimagen roscopy (SEM) muestra una sección transversal a través de la nanopore permitiendo el acceso a las cavidades femtoliter dentro del chip de silicio. Esta cifra se reutiliza con permiso de 21 años. Por favor, haga clic aquí para ver una versión más grande de esta figura.
Todos los análisis de los datos se realiza utilizando software gratuito para garantizar el acceso sin restricciones para los usuarios finales. Las series temporales se analizaron utilizando el software de procesamiento de imágenes gratuito y un procesamiento por lotes que permite el software de análisis de la curva de generación personalizada y la correlación directa de grandes conjuntos de datos con múltiples canales fluorescentes y miles de curvas.
La proteína modelo utilizado en este protocolo es el canal mechanosensitive de proteínas de gran conductancia (MscL) canal derivado de E. coli. Funciona como una válvula para liberar choque osmótico en la naturaleza, pero se ha modificado de tal manera que racionalmente diseñada syntfuncionalidades hetic covalentemente se pueden unir a la banda de los canales de constricción. Via carga repulsión del activador unido covalentemente (MTSET) el canal se activa para abrir, la creación de una válvula de nano. Las pequeñas moléculas como iones, agua, proteínas pequeñas, pero también pequeños fluoróforos pueden permear a través del canal. Aquí, la proteína se usa como un modelo para demostrar la capacidad del sistema para detectar la translocación mediada por proteínas.
Protocol
Representative Results
Discussion
La técnica que aquí se presenta permite un análisis altamente paralela de transporte de proteínas de membrana. sistemas de proteínas de membrana reconstituidas directamente se pueden aplicar a la biochip, por lo que la adaptación de teóricamente cada transportador de membrana o canalizar posible. el análisis del transporte sólo está limitado por el establecimiento de un sistema de lectura de fluorescencia, ya sea a través de los cambios directos de fluorescencia (translocación de fluoróforos o sustratos mar…
Disclosures
The authors have nothing to disclose.
Acknowledgements
We thank Barbara Windschiegl for her help in establishing SOPs; Dennis Remme for his work on the NanoCalcFX software and Alina Kollmannsperger, Markus Braner and Milan Gerovac for helpful suggestions on the manuscript. The German-Israeli Project Cooperation (DIP) provided by the DFG and the Federal Ministry of Education and Research to R.T., as well as the Federal Ministry of Economics and Technology (ZIM R&D Project) to R.T. and Nanospot GmbH supported this work.
Materials
| Reagent | |||
| [2-(Trimethylammonium)ethyl] methanethiosulfonate |
Toronto Research Chemicals Inc. | T792900 | MTSET; hydrolized by water. Keep as dry pouder aliquot at -80 °C. Use immediately (30 minutes) after solubilization in buffer. |
| 1 ml gas-tight syringe | Hamilton | #1001 | |
| 10 ml round flask | Schott Duran | ||
| 2.7 mm glas beads | Roth | N032.1 | |
| 2-Propanole | Roth | 9866.5 | |
| 30 cm Luer-Lock Extension Tube | Sarstedt | 744304 | |
| Acetone | Roth | 5025.5 | |
| Bio-Beads SM-2 Adsorbent | Bio-Rad | 152-3920 | need to be activated before first use |
| CaCl2 Dihydrat | Roth | HN04.3 | |
| Calcein | Sigma | C0875 | store dark at -20 °C |
| Chloroform reagent grade | VWR Chemicals | 22711324 | |
| DOPEATTO390 | ATTO-TEC | AD 390-165 | store dark at -20 °C |
| Ethanol absolute | Sigma-Aldrich | 32205 | |
| Injekt Single-use syringe | Braun | 460 60 51V | |
| Injekt-F single-use syringe | Braun | 91 66 017V | |
| Keck clips | Schott | KC29 | |
| L-α-Phosphatidylcholine, 20% (Soy) | Avanti Polar Lipids | 5416016 | store under inert gas at -20 °C |
| NaCl 99.5% p.a. | Roth | 3957.2 | |
| Nanopore E100 wafer/chips | Micromotive (Mainz/Germany) | available on request | |
| Nucleopore Track-Etch Membrane 0.4 µm | Whatman | 800282 | |
| Oregon Green Dextran 488 (70 kDa) | life Technologies | D-7173 | store dark at -20 °C |
| Oy647 | Luminartis (Münster/Germany) | OY-647-T-1mg | store dark at -20 °C |
| Rotilabo-syringe filtration, unsterile, pore-size 0.22 µm | Roth | P 818.1 | |
| Sephadex G-50 | Sigma-Aldrich | G5080 | column material for size exclusion chromatography |
| Silastic MDX4-4210 | Dow Corning | curing agent for chip fixation onto cover glass support | |
| sticky-Slide 8-well | ibidi | 80828 | multi-well chamber for the mounting onto glass slides (chip holder) |
| Three-way stopcock blue | Sarstedt | 744410001 | |
| Tris Pufferan 99.9% Ultra Quality | Roth | 5429.2 | |
| Triton-X 100 | Roth | 6683.1 | |
| Whatman 0.2 µm cellulose nitrate membrane filter | Roth | NH69.1 | |
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| Büchi 461 water bath | Büchi | ||
| Büchi Rotavapor RE 111 | Büchi | ||
| Cary Eclipse Fluorescence Spectrophtometer | Varian | ||
| LiposoFast Mini Extruder | Avestin | ||
| Membrane pump | Vaccubrand | 15430 | |
| Nanosight Nanoparticle Tracking Microscope | Malvern / Nanosight | LM 14C | |
| NyONE microscope | Synentec | available on request | |
| Pump control | Vaccubrand | CVC 2II | |
| Sonicator bath Sonorex RK100H | Brandelin electronic | 31200001107477 | |
| Vaccum pump RC5 | Vaccubrand | 1805400204 | |
| Water bath W13 | Haake | 002-9910 | |
| Plasma Cleaner PDC-37G | Harrick Plasma | PDC-37G | |
| Name | Company | Catalog Number | Comments |
| Software | |||
| ImageJ | Open Source | http://imagej.nih.gov/ij/ | scientific image processing software |
| NanoCalcFX | Freeware | http://sourceforge.net/projects/nanocalc/ | data analysis/evaluation software for massive transport kinetic datasets |
| NTA 2.3 Analytical Software | Nanosight | data acquisition and analysis software for nanoparticle tracking microscope | |
| NTA 2.3 Temperature Comms | Nanosight | temperature controle software for nanoparticle tracking microscope |
References
- Yildirim, M. A., Goh, K. I., Cusick, M. E., Barabasi, A. L., Vidal, M. Drug-target network. Nat Biotechnol. 25, 1119 (2007).
- Hamill, O. P., Marty, A., Neher, E., Sakmann, B., Sigworth, F. Improved patch-clamp techniques for high-resolution current recording from cells and cell-free membrane patches. Pflugers Arch. 391, 85-100 (1981).
- Osaki, T., Suzuki, H., Le Pioufle, B., Takeuchi, S. Multichannel simultaneous measurements of single-molecule translocation in α-hemolysin nanopore array. Anal. Chem. 81, 9866-9870 (2009).
- Carrillo, L., et al. High-resolution membrane capacitance measurements for studying endocytosis and exocytosis in yeast. Traffic. , (2015).
- Giacomini, K. M., et al. Membrane transporters in drug development. Nat. Rev. Drug Discov. 9, 215-236 (2010).
- Castell, O. K., Berridge, J., Wallace, M. I. Quantification of membrane protein inhibition by optical ion flux in a droplet interface bilayer array. Angew. Chem. Int. Ed. 51, 3134-3138 (2012).
- Zollmann, T., et al. Single liposome analysis of peptide translocation by the ABC transporter TAPL. Proc. Natl. Acad. Sci. U.S.A. 112, 2046-2051 (2015).
- Tamm, L. K., McConnell, H. M. Supported phospholipid bilayers. Biophys. J. 47, 105-113 (1985).
- Sackmann, E. Supported membranes: scientific and practical applications. Science. 271, 43-48 (1996).
- Castellana, E. T., Cremer, P. S. Solid supported lipid bilayers: From biophysical studies to sensor design. Surf. Sci. Rep. 61, 429-444 (2006).
- Wagner, M. L., Tamm, L. K. Tethered polymer-supported planar lipid bilayers for reconstitution of integral membrane proteins: silane-polyethyleneglycol-lipid as a cushion and covalent linker. Biophys. J. 79, 1400-1414 (2000).
- Naumann, C. A., et al. The polymer-supported phospholipid bilayer: tethering as a new approach to substrate-membrane stabilization. Biomacromolecules. 3, 27-35 (2002).
- Weiskopf, D., Schmitt, E. K., Klühr, M. H., Dertinger, S. K., Steinem, C. Micro-BLMs on highly ordered porous silicon substrates: Rupture process and lateral mobility. Langmuir. 23, 9134-9139 (2007).
- Watanabe, R., et al. Arrayed lipid bilayer chambers allow single-molecule analysis of membrane transporter activity. Nat. Commun. 5, (2014).
- Stamou, D., Duschl, C., Delamarche, E., Vogel, H. Self-Assembled Microarrays of Attoliter Molecular Vessels. Angew. Chem. Int. Ed. 115, 5738-5741 (2003).
- Lohr, C., et al. Single Liposomes Used to Study the Activity of Individual Transporters. Biophysical Journal. 106, 229a (2014).
- Dunlop, J., Bowlby, M., Peri, R., Vasilyev, D., Arias, R. High-throughput electrophysiology: an emerging paradigm for ion-channel screening and physiology. Nat. Rev. Drug Discov. 7, 358-368 (2008).
- Milligan, C. J., et al. Robotic multiwell planar patch-clamp for native and primary mammalian cells. Nat. Protoc. 4, 244-255 (2009).
- Soga, N., Watanabe, R., Noji, H. Attolitre-sized lipid bilayer chamber array for rapid detection of single transporters. Sci. Rep. 5, (2015).
- Kleefen, A., et al. Multiplexed parallel single transport recordings on nanopore arrays. Nano Lett. 10, 5080-5087 (2010).
- Wei, R., Gatterdam, V., Wieneke, R., Tampé, R., Rant, U. Stochastic sensing of proteins with receptor-modified solid-state nanopores. Nat. Nanotechnol. 7, 257-263 (2012).
- Urban, M., et al. Highly parallel transport recordings on a membrane-on-nanopore chip at single molecule resolution. Nano Lett. 14, 1674-1680 (2014).
- Kusters, I., Van Oijen, A. M., Driessen, A. J. Membrane-on-a-Chip: Microstructured Silicon/Silicon-Dioxide Chips for High-Throughput Screening of Membrane Transport and Viral Membrane Fusion. ACS Nano. 8, 3380-3392 (2014).
- Hansen, J. S., Thompson, J. R., Hélix-Nielsen, C., Malmstadt, N. Lipid directed intrinsic membrane protein segregation. J. Am. Chem. Soc. 135, 17294-17297 (2013).
- Heinemann, F., Schwille, P. Preparation of Micrometer-Sized Free-Standing Membranes. ChemPhysChem. 12, 2568-2571 (2011).
- Lazzara, T. D., Carnarius, C., Kocun, M., Janshoff, A., Steinem, C. Separating attoliter-sized compartments using fluid pore-spanning lipid bilayers. ACS nano. 5, 6935-6944 (2011).
- Winterhalter, M. Black lipid membranes. Curr. Opin. Colloid Interface Sci. 5, 250-255 (2000).
- Koçer, A., Walko, M., Feringa, B. L. Synthesis and utilization of reversible and irreversible light-activated nanovalves derived from the channel protein MscL. Nat. Protoc. 2, 1426-1437 (2007).

