Membrane Transport processus analysés par un système hautement parallèle Nanopore Chip à protéine unique Résolution
Summary
The presented protocol describes the analysis of membrane protein mediated transport on the single transporter level using pore-spanning solvent-free lipid bilayers. This is achieved by the creation of bulk produced nanopore array chips, combined with highly parallel data acquisition and analysis, enabling the future establishment of membrane protein effector screenings.
Abstract
Membrane transport des protéines au niveau de protéine unique échappe encore une analyse détaillée, si le substrat translocation est non-électrogénique. Des efforts considérables ont été faits dans ce domaine, mais les techniques permettant l'analyse automatisée de transport à haut débit en combinaison avec des techniques de bicouches lipidiques sans solvant requis pour l'analyse des transporteurs membranaires sont rares. Cette classe de transporteurs est toutefois crucial dans l'homéostasie cellulaire et donc une cible clé dans le développement de médicaments et de méthodologies pour acquérir de nouvelles connaissances désespérément besoin.
Le manuscrit présenté ici décrit la création et la manipulation d'un roman biopuce pour l'analyse des processus de transport de protéines membranaires médiation à la résolution du transporteur unique. La biopuce est composé de microcavités fermées par nanopores qui est hautement parallèle dans sa conception et peuvent être produits de qualité industrielle et de la quantité. liposomes Protein-hébergeant peuvent être directement appliquées àla surface de la puce formant des bicouches lipidiques pores transmembranaires auto-assemblées à l'aide de SSM-techniques (solide supporté membranes lipidiques). Pores transmembranaires parties de la membrane sont autonomes, fournissant l'interface substrat pour la translocation dans ou hors de l'espace de la cavité, qui peut être suivie par la lecture de fluorescence multispectral en temps réel. La mise en place de procédures normalisées d'exploitation (SOP) permet à la mise en place directe des bicouches lipidiques de protéines hébergeant sur la surface de la puce de pratiquement toutes les protéines de la membrane qui peut être reconstituée fonctionnellement. La seule condition préalable est la mise en place d'un système de lecture fluorescent pour les substrats de transport non électrogènes.
Les applications à haute teneur dépistage sont accomplishable par l'utilisation de microscopes automatisés fluorescents inversés d'enregistrement multiples puces en parallèle. Les grands ensembles de données peuvent être analysées en utilisant le logiciel d'analyse conçu sur mesure librement disponibles. Trois couleurs multiples spectrale fluorescentelecture permet en outre de discrimination de données impartiale dans différentes classes d'événements, ce qui élimine des résultats faussement positifs.
La technologie de la puce est actuellement basée sur SiO 2 surfaces, mais fonctionnalisation supplémentaire en utilisant des surfaces de puces revêtues d' or est également possible.
Introduction
L'analyse des protéines membranaires est devenue d'un intérêt croissant pour la recherche fondamentale et pharmaceutique au cours des 20 dernières années. Le développement de nouveaux médicaments dépend de l'identification et de la caractérisation détaillée des nouvelles cibles, étant actuellement l'un des facteurs limitants. Le fait que 60% de toutes les cibles médicamenteuses sont des protéines membranaires 1, rend le développement de techniques pour élucider leur fonction la plus importante.
Dans le passé, des techniques pour l'étude des canaux électrogènes et les transporteurs ont été développés dans la multitude 2-4. substrats non électrogènes en contraire présentent une tâche plus difficile. Ils sont cependant d' un intérêt particulier en tant que cibles de médicaments de choix, car ils contrôlent le flux de solutés et de nutriments à travers la membrane cellulaire et la fonction des récepteurs 5 clés en cascades de signalisation.
Des efforts considérables ont été mis dans le développement de techniques pour étudier la fonction des protéines de transport membranaire 6, 7. Les systèmes utilisant des membranes sur support solide ont émergé comme des outils les plus prometteurs dans ce domaine 8-10, y compris solides bicouches lipidiques supportées, bicouches captifs 11, 12, membranes microblack lipidiques 13, 14 et natifs tableaux vésiculaires 15, 16 pour ne citer que quelques – uns. Certains d'entre eux sont même disponibles en configurations commerciales 17, 18. Quelques exemples ont été publiés en combinant la capacité d'étudier les protéines membranaires simples d'une manière hautement parallèle 14, 19, une condition préalable pour les applications de dépistage. Cependant, ces méthodes combler rarement de la recherche fondamentale à l'environnement industriel. Les difficultés se situent souvent dans la capacité du système à être automatisable, la production coûteuse et / ou d'une préparation laborieuse. Une approche overcoming tous les obstacles mentionnés ci-dessus est le but final.
La technique présentée ici a été développé pour étudier les canaux de membrane et des transporteurs in vitro dans un environnement contrôlé , le niveau de protéine unique 20-22. Reconstitution des protéines membranaires purifiées en LUV est beaucoup plus établi que les approches comparables pour GUVs 23 – 26 ou lipides noir membranes 27. Ils peuvent être directement appliqués sur la surface de la puce, où la formation de deux couches se déroule par l'intermédiaire d'un processus d'auto-assemblage. La conception à fond de verre de la puce nanoporeux (Fig. 1) permet pour la microscopie de l' air, ce qui permet l'automatisation simple du système. En combinaison avec une platine motorisée plusieurs puces peuvent être mesurées en même temps, chaque champ de vision contenant des milliers de cavités scellées pour l'analyse.
<p class="jove_content" fo:keep-together.within-page= "1">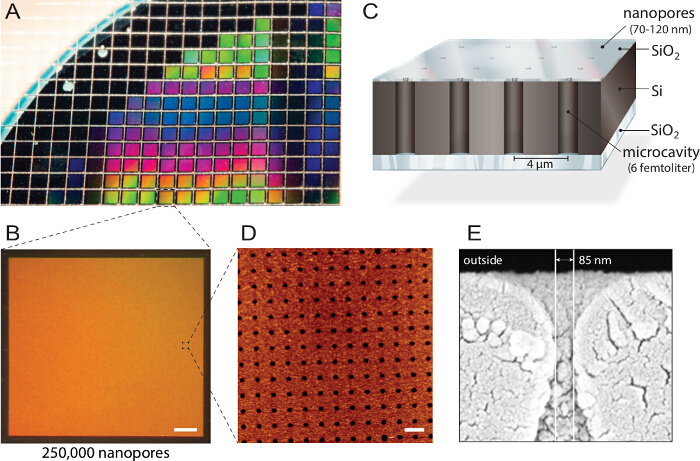
La figure 1. Conception de biopuces nanopore multiplexés. A) de silicium sur isolant (SOI) wafer est structuré par gravure ionique réactive. Environ 1.150 puces individuelles sont fabriquées à partir de chaque plaquette avec des propriétés identiques et de qualité. B) Chaque puce comprend 250.000 microcavités individuels avec des ouvertures nano. Barre d' échelle:. 200 um C) Chaque cavité est adressable par fluorescence multi-spectrale lecture. Un opaques blocs supérieurs de la couche les signaux fluorescents provenant du réservoir tampon, ce qui rend la biopuce compatible avec inversées microscopes à fluorescence. D) microscopie à force atomique (AFM) imagerie révèle disposés de manière uniforme des ouvertures de pores et la rugosité de la surface de la couche de dioxyde de silicium de 3,6 nm (n = 40) optimale pour la fusion des vésicules. Barre d' échelle: 5 um E) micro électronique à balayage.roscopie d'image (SEM) montre une coupe transversale à travers le nanopore permettant l'accès aux cavités femtolitre à l'intérieur de la puce de silicium. Ce chiffre a été réutilisé avec l' autorisation de 21. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
Toute analyse des données est effectuée en utilisant freeware pour garantir un accès sans restriction pour les utilisateurs finaux. Les séries temporelles sont analysées en utilisant un logiciel de traitement d'images gratuit et d'un traitement par lots logiciel d'analyse de la courbe de génération personnalisée permettant et la corrélation directe de grands ensembles de données avec de multiples canaux fluorescents et des milliers de courbes.
La protéine de modèle utilisé dans ce protocole est le canal mécanosensible de protéines grande conductance (MscL) de canal dérivé de E. coli. Il fonctionne comme une soupape pour libérer un choc osmotique dans la nature, mais a été modifiée d'une manière telle que conçue rationnellement syntfonctionnalités HETIC peuvent covalente être fixés sur le côté des canaux d'étranglement. Via charge la répulsion de l'activateur lié de façon covalente (MTSET) le canal est déclenché pour ouvrir, créer une nano-valve. Les petites molécules comme les ions, l'eau, de petites protéines, mais aussi les petites fluorophores peuvent passer à travers le canal. Ici, la protéine est utilisée comme modèle pour démontrer la capacité du système pour détecter la translocation de la protéine à médiation.
Protocol
Representative Results
Discussion
La technique présentée ici permet une analyse très parallèle de transport des protéines de la membrane. des systèmes protéiques de la membrane reconstituée peuvent être directement appliquées sur la biopuce, ce qui rend l'adaptation de la théorie, chaque transporteur membranaire ou canaux possibles. l'analyse de transport est seulement limité par la mise en place d'un système de lecture fluorescente, soit par changement direct de fluorescence (translocation des fluorophores ou des substrats marq…
Disclosures
The authors have nothing to disclose.
Acknowledgements
We thank Barbara Windschiegl for her help in establishing SOPs; Dennis Remme for his work on the NanoCalcFX software and Alina Kollmannsperger, Markus Braner and Milan Gerovac for helpful suggestions on the manuscript. The German-Israeli Project Cooperation (DIP) provided by the DFG and the Federal Ministry of Education and Research to R.T., as well as the Federal Ministry of Economics and Technology (ZIM R&D Project) to R.T. and Nanospot GmbH supported this work.
Materials
| Reagent | |||
| [2-(Trimethylammonium)ethyl] methanethiosulfonate |
Toronto Research Chemicals Inc. | T792900 | MTSET; hydrolized by water. Keep as dry pouder aliquot at -80 °C. Use immediately (30 minutes) after solubilization in buffer. |
| 1 ml gas-tight syringe | Hamilton | #1001 | |
| 10 ml round flask | Schott Duran | ||
| 2.7 mm glas beads | Roth | N032.1 | |
| 2-Propanole | Roth | 9866.5 | |
| 30 cm Luer-Lock Extension Tube | Sarstedt | 744304 | |
| Acetone | Roth | 5025.5 | |
| Bio-Beads SM-2 Adsorbent | Bio-Rad | 152-3920 | need to be activated before first use |
| CaCl2 Dihydrat | Roth | HN04.3 | |
| Calcein | Sigma | C0875 | store dark at -20 °C |
| Chloroform reagent grade | VWR Chemicals | 22711324 | |
| DOPEATTO390 | ATTO-TEC | AD 390-165 | store dark at -20 °C |
| Ethanol absolute | Sigma-Aldrich | 32205 | |
| Injekt Single-use syringe | Braun | 460 60 51V | |
| Injekt-F single-use syringe | Braun | 91 66 017V | |
| Keck clips | Schott | KC29 | |
| L-α-Phosphatidylcholine, 20% (Soy) | Avanti Polar Lipids | 5416016 | store under inert gas at -20 °C |
| NaCl 99.5% p.a. | Roth | 3957.2 | |
| Nanopore E100 wafer/chips | Micromotive (Mainz/Germany) | available on request | |
| Nucleopore Track-Etch Membrane 0.4 µm | Whatman | 800282 | |
| Oregon Green Dextran 488 (70 kDa) | life Technologies | D-7173 | store dark at -20 °C |
| Oy647 | Luminartis (Münster/Germany) | OY-647-T-1mg | store dark at -20 °C |
| Rotilabo-syringe filtration, unsterile, pore-size 0.22 µm | Roth | P 818.1 | |
| Sephadex G-50 | Sigma-Aldrich | G5080 | column material for size exclusion chromatography |
| Silastic MDX4-4210 | Dow Corning | curing agent for chip fixation onto cover glass support | |
| sticky-Slide 8-well | ibidi | 80828 | multi-well chamber for the mounting onto glass slides (chip holder) |
| Three-way stopcock blue | Sarstedt | 744410001 | |
| Tris Pufferan 99.9% Ultra Quality | Roth | 5429.2 | |
| Triton-X 100 | Roth | 6683.1 | |
| Whatman 0.2 µm cellulose nitrate membrane filter | Roth | NH69.1 | |
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| Büchi 461 water bath | Büchi | ||
| Büchi Rotavapor RE 111 | Büchi | ||
| Cary Eclipse Fluorescence Spectrophtometer | Varian | ||
| LiposoFast Mini Extruder | Avestin | ||
| Membrane pump | Vaccubrand | 15430 | |
| Nanosight Nanoparticle Tracking Microscope | Malvern / Nanosight | LM 14C | |
| NyONE microscope | Synentec | available on request | |
| Pump control | Vaccubrand | CVC 2II | |
| Sonicator bath Sonorex RK100H | Brandelin electronic | 31200001107477 | |
| Vaccum pump RC5 | Vaccubrand | 1805400204 | |
| Water bath W13 | Haake | 002-9910 | |
| Plasma Cleaner PDC-37G | Harrick Plasma | PDC-37G | |
| Name | Company | Catalog Number | Comments |
| Software | |||
| ImageJ | Open Source | http://imagej.nih.gov/ij/ | scientific image processing software |
| NanoCalcFX | Freeware | http://sourceforge.net/projects/nanocalc/ | data analysis/evaluation software for massive transport kinetic datasets |
| NTA 2.3 Analytical Software | Nanosight | data acquisition and analysis software for nanoparticle tracking microscope | |
| NTA 2.3 Temperature Comms | Nanosight | temperature controle software for nanoparticle tracking microscope |
References
- Yildirim, M. A., Goh, K. I., Cusick, M. E., Barabasi, A. L., Vidal, M. Drug-target network. Nat Biotechnol. 25, 1119 (2007).
- Hamill, O. P., Marty, A., Neher, E., Sakmann, B., Sigworth, F. Improved patch-clamp techniques for high-resolution current recording from cells and cell-free membrane patches. Pflugers Arch. 391, 85-100 (1981).
- Osaki, T., Suzuki, H., Le Pioufle, B., Takeuchi, S. Multichannel simultaneous measurements of single-molecule translocation in α-hemolysin nanopore array. Anal. Chem. 81, 9866-9870 (2009).
- Carrillo, L., et al. High-resolution membrane capacitance measurements for studying endocytosis and exocytosis in yeast. Traffic. , (2015).
- Giacomini, K. M., et al. Membrane transporters in drug development. Nat. Rev. Drug Discov. 9, 215-236 (2010).
- Castell, O. K., Berridge, J., Wallace, M. I. Quantification of membrane protein inhibition by optical ion flux in a droplet interface bilayer array. Angew. Chem. Int. Ed. 51, 3134-3138 (2012).
- Zollmann, T., et al. Single liposome analysis of peptide translocation by the ABC transporter TAPL. Proc. Natl. Acad. Sci. U.S.A. 112, 2046-2051 (2015).
- Tamm, L. K., McConnell, H. M. Supported phospholipid bilayers. Biophys. J. 47, 105-113 (1985).
- Sackmann, E. Supported membranes: scientific and practical applications. Science. 271, 43-48 (1996).
- Castellana, E. T., Cremer, P. S. Solid supported lipid bilayers: From biophysical studies to sensor design. Surf. Sci. Rep. 61, 429-444 (2006).
- Wagner, M. L., Tamm, L. K. Tethered polymer-supported planar lipid bilayers for reconstitution of integral membrane proteins: silane-polyethyleneglycol-lipid as a cushion and covalent linker. Biophys. J. 79, 1400-1414 (2000).
- Naumann, C. A., et al. The polymer-supported phospholipid bilayer: tethering as a new approach to substrate-membrane stabilization. Biomacromolecules. 3, 27-35 (2002).
- Weiskopf, D., Schmitt, E. K., Klühr, M. H., Dertinger, S. K., Steinem, C. Micro-BLMs on highly ordered porous silicon substrates: Rupture process and lateral mobility. Langmuir. 23, 9134-9139 (2007).
- Watanabe, R., et al. Arrayed lipid bilayer chambers allow single-molecule analysis of membrane transporter activity. Nat. Commun. 5, (2014).
- Stamou, D., Duschl, C., Delamarche, E., Vogel, H. Self-Assembled Microarrays of Attoliter Molecular Vessels. Angew. Chem. Int. Ed. 115, 5738-5741 (2003).
- Lohr, C., et al. Single Liposomes Used to Study the Activity of Individual Transporters. Biophysical Journal. 106, 229a (2014).
- Dunlop, J., Bowlby, M., Peri, R., Vasilyev, D., Arias, R. High-throughput electrophysiology: an emerging paradigm for ion-channel screening and physiology. Nat. Rev. Drug Discov. 7, 358-368 (2008).
- Milligan, C. J., et al. Robotic multiwell planar patch-clamp for native and primary mammalian cells. Nat. Protoc. 4, 244-255 (2009).
- Soga, N., Watanabe, R., Noji, H. Attolitre-sized lipid bilayer chamber array for rapid detection of single transporters. Sci. Rep. 5, (2015).
- Kleefen, A., et al. Multiplexed parallel single transport recordings on nanopore arrays. Nano Lett. 10, 5080-5087 (2010).
- Wei, R., Gatterdam, V., Wieneke, R., Tampé, R., Rant, U. Stochastic sensing of proteins with receptor-modified solid-state nanopores. Nat. Nanotechnol. 7, 257-263 (2012).
- Urban, M., et al. Highly parallel transport recordings on a membrane-on-nanopore chip at single molecule resolution. Nano Lett. 14, 1674-1680 (2014).
- Kusters, I., Van Oijen, A. M., Driessen, A. J. Membrane-on-a-Chip: Microstructured Silicon/Silicon-Dioxide Chips for High-Throughput Screening of Membrane Transport and Viral Membrane Fusion. ACS Nano. 8, 3380-3392 (2014).
- Hansen, J. S., Thompson, J. R., Hélix-Nielsen, C., Malmstadt, N. Lipid directed intrinsic membrane protein segregation. J. Am. Chem. Soc. 135, 17294-17297 (2013).
- Heinemann, F., Schwille, P. Preparation of Micrometer-Sized Free-Standing Membranes. ChemPhysChem. 12, 2568-2571 (2011).
- Lazzara, T. D., Carnarius, C., Kocun, M., Janshoff, A., Steinem, C. Separating attoliter-sized compartments using fluid pore-spanning lipid bilayers. ACS nano. 5, 6935-6944 (2011).
- Winterhalter, M. Black lipid membranes. Curr. Opin. Colloid Interface Sci. 5, 250-255 (2000).
- Koçer, A., Walko, M., Feringa, B. L. Synthesis and utilization of reversible and irreversible light-activated nanovalves derived from the channel protein MscL. Nat. Protoc. 2, 1426-1437 (2007).

