Membrantransportprozesse durch eine hochparallele Nanopore Chip-System bei Einzel Protein Resolution Analysierte
Summary
The presented protocol describes the analysis of membrane protein mediated transport on the single transporter level using pore-spanning solvent-free lipid bilayers. This is achieved by the creation of bulk produced nanopore array chips, combined with highly parallel data acquisition and analysis, enabling the future establishment of membrane protein effector screenings.
Abstract
Membranproteintransport auf dem einzelnen Proteinebene ausweicht noch detaillierte Analyse, wenn das transloziert Substrat nicht elektrogener ist. Erhebliche Anstrengungen wurden in diesem Bereich, sondern Techniken ermöglicht automatisierte Hochdurchsatz-Transport-Analyse in Kombination mit lösemittelfreien Techniken Lipid-Doppelschicht für die Analyse von Membrantransportern sind erforderlich selten. Diese Klasse von Transportern ist jedoch von entscheidender Bedeutung in der Zelle Homöostase und daher ein zentrales Ziel in der Medikamentenentwicklung und Methoden neue Erkenntnisse dringend benötigt zu gewinnen.
Die hier präsentierten Handschrift beschreibt den Aufbau und die Handhabung eines neuartigen Biochips für die Analyse von Membranprotein-vermittelte Transportprozesse auf Einzel transporter Auflösung. Der Biochip besteht aus Mikrokavitäten durch Nanoporen eingeschlossen, die stark parallel in seiner Gestaltung und kann in Industriequalität und Menge hergestellt werden. Protein-tragende Liposomen können direkt angewendet werdendie Chipoberfläche bilden selbstorganisierende poren Spanning Lipid-Doppelschichten SSM-Techniken (feste Lipidmembranen unterstützt). Pore-Spanning Teile der Membran sind freistehend, Bereitstellen der Schnittstelle für das Substrat Translokation in den oder aus dem Hohlraum, die durch multispektrale Fluoreszenzanzeige in Echtzeit verfolgt werden können. Die Einrichtung von Standard Operating Procedures (SOPs) ermöglicht die einfache Einrichtung von Protein-tragenden Lipid-Doppelschichten auf der Chipoberfläche praktisch jeder Membranprotein, das funktionell wiederhergestellt werden kann. Einzige Voraussetzung ist die Schaffung eines Fluoreszenzlesesystem für Nicht-elektrogener Transportgründen.
High-Content-Screening-Anwendungen sind erfüllbar durch den Einsatz von automatisierten inversen Fluoreszenzmikroskope mehrere Chips parallel aufzuzeichnen. Große Datenmengen können mit dem frei verfügbaren anwendungsspezifische Analyse-Software analysiert werden. Drei-Farben-Mehrspektrale FluoreszenzAuslesen erlaubt darüber hinaus für unvoreingenommene Daten Diskriminierung in verschiedenen Ereignisklassen, falsch positive Ergebnisse zu beseitigen.
Die Chip – Technologie basiert derzeit auf SiO 2 -Oberflächen, aber weitere Funktionalisierung goldbeschichteten Spanflächen ist ebenfalls möglich.
Introduction
Die Analyse von Membranproteinen ist von Interesse in den letzten 20 Jahren für die Grund- und pharmazeutischen Forschung erhöht werden. Die Entwicklung neuer Medikamente ist abhängig von der Identifizierung und detaillierte Charakterisierung neuer Ziele, die derzeit einer der limitierenden Faktoren zu sein. Die Tatsache , dass etwa 60% aller drug targets sind Membranproteine 1, macht die Entwicklung von Techniken , um ihre Funktion wichtigsten aufzuklären.
4 – In der Vergangenheit wurden in 2 Vielzahl entwickelten Techniken für die Untersuchung von elektrogenen Kanäle und Transporter. Nicht elektrogener Substrate im Gegensatz eine schwierige Aufgabe darstellen. Sie sind jedoch von besonderem Interesse als Hauptangriffspunkte für Medikamente, da sie den Fluss von gelösten Stoffen und Nährstoffen durch die Zellmembran und die Funktion als Schlüsselrezeptoren in Signalkaskaden 5 steuern.
Beträchtliche Anstrengungen wurden in die Entwicklung von t gesetztechniques die Funktion von Membrantransportproteinen 6, 7 zu untersuchen. 10, darunter auch feste unterstützten Lipid – Doppelschichten, tethered Bilayer 11, 12, microblack Lipidmembranen 13, 14 und nativen Vesikel – Arrays 15, 16 ein paar zu nennen – Systeme festen Träger unter Verwendung von Membranen wurden als vielversprechendsten Instrumente auf diesem Gebiet 8 entstanden. Einige von ihnen sind sogar als Handelsaufbauten 17, 18. Einige Beispiele wurden veröffentlicht Kombination die Fähigkeit einzelne Membranproteine in einer hochparallelen Weise 14 zu studieren, 19, Voraussetzung für Screening – Anwendungen. Allerdings überbrücken diese Methoden nur selten aus der Grundlagenforschung zur industriellen Umfeld. Die Schwierigkeiten liegen oft in der Fähigkeit des Systems, automatisierbar zu sein, die kostenintensive Herstellung und / oder aufwendige Vorbereitung. Ein Ansatz overcoming alle oben erwähnten Hindernisse ist das Endziel.
Die Technik , die hier vorgestellt wurde entwickelt Membrankanäle und Transporter in vitro in einer kontrollierten Umgebung auf dem einzelnen Protein – Ebene 20 zu studieren – 22. Die Rekonstitution von gereinigten Membranproteinen in LUVs ist viel mehr etabliert als vergleichbare Ansätze für GUVs 23-26 oder schwarzen Lipidmembranen 27. Sie können direkt auf die Chipoberfläche aufgebracht werden, in dem Doppelschichtbildung erfolgt über eine Selbstorganisationsprozess stattfindet. Der Glasboden Design des nanoporösen Chip (Fig. 1) ermöglicht eine Luft Mikroskopie, die die einfache Automatisierung des Systems ermöglicht. In Kombination mit einer motorisierten Stufe kann mehrere Chips gleichzeitig gemessen werden, wobei jedes Sichtfeld Tausende von abgedichteten Hohlräumen zur Analyse enthält.
<p class="jove_content" fo:keep-together.within-page= "1">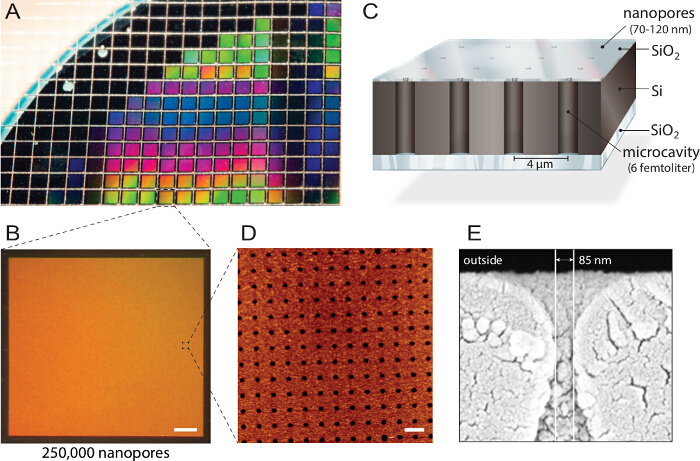
Abbildung 1. Design von gemultiplexten Nanopore Biochips. A) A silicon-on-insulator (SOI) Wafers durch reaktives Ionenätzen strukturiert. Ungefähr 1.150 einzelne Chips werden von jedem Wafer hergestellt mit identischen Eigenschaften und Qualität. B) Jeder Chip umfasst 250.000 einzelne Mikrokavitäten mit Nano – Öffnungen. Maßstabsbalken:. 200 & mgr; m C) Jeder Hohlraum ist adressierbar über multispektrale Fluoreszenz ausgelesen. Eine undurchsichtige Deckschicht blockiert die Fluoreszenzsignale aus dem Pufferspeicher, so dass der Biochip kompatibel mit invertierten Fluoreszenzmikroskop. D) Rasterkraftmikroskopie (AFM) Bildgebung Porenöffnungen und Oberflächenrauhigkeit der Siliziumdioxid – Schicht von 3,6 nm gleichmäßig angeordnet enthüllt (n = 40) optimal für Vesikelfusion. Maßstabsbalken:. 5 um E) Rasterelektronen microscopy (SEM) Aufnahme zeigt einen Querschnitt durch die Nanopore die den Zugang zu den Femtoliter Hohlräume innerhalb des Siliziumchips. Diese Zahl wurde mit Erlaubnis wiederverwendet werden von 21. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
Alle Datenanalyse wird mit Freeware durchgeführt uneingeschränkten Zugriff für Endanwender zu gewährleisten. Zeitreihen sind mit dem kostenlosen Bildverarbeitungssoftware analysiert und eine benutzerdefinierte Build-Kurvenanalyse-Software ermöglicht die Stapelverarbeitung und einfache Korrelation von großen Datensätzen mit mehreren Fluoreszenzkanälen und Tausende von Kurven.
Das Modellprotein in diesem Protokoll wird die mechanosensitive Kanal von großer Leitfähigkeit (MscL) Kanalproteins von E. abgeleitet coli. Es fungiert als ein Ventil in der Natur osmotischen Schock freizusetzen, wurde aber derart modifiziert, dass synt rational entworfenhetic Funktionalitäten können kovalent an die Kanäle Einschnürung Seite angebracht werden. Via Ladungs Abstoßung des kovalent gebundenen Aktivator (MTSET) der Kanal ausgelöst wird, zu öffnen, die Schaffung eines Nano-Ventil. Kleine Moleküle, wie Ionen, Wasser, kleine Proteine, aber auch kleine Fluorophore können durch den Kanal zu durchdringen. Hierbei wird das Protein als ein Modell verwendet, um die Fähigkeit des Systems zu demonstrieren Protein-vermittelte Translokation zu detektieren.
Protocol
Representative Results
Discussion
Die hier vorgestellte Technik ermöglicht eine hochparallele Analyse von Membranproteintransport. Rekonstituierten Membranprotein-Systeme können direkt auf dem Biochip aufgebracht werden, so dass die Anpassung der theoretisch jeder Membrantransporter oder möglichen Kanal. Transport-Analyse wird durch die Einrichtung eines fluoreszierenden Auslesesystem, entweder durch direkte Fluoreszenzänderung (Translokation von Fluorophore oder fluoreszenzmarkierte Substrate) oder indirekte Fluoreszenzänderung (pH-sensitiven Farb…
Disclosures
The authors have nothing to disclose.
Acknowledgements
We thank Barbara Windschiegl for her help in establishing SOPs; Dennis Remme for his work on the NanoCalcFX software and Alina Kollmannsperger, Markus Braner and Milan Gerovac for helpful suggestions on the manuscript. The German-Israeli Project Cooperation (DIP) provided by the DFG and the Federal Ministry of Education and Research to R.T., as well as the Federal Ministry of Economics and Technology (ZIM R&D Project) to R.T. and Nanospot GmbH supported this work.
Materials
| Reagent | |||
| [2-(Trimethylammonium)ethyl] methanethiosulfonate |
Toronto Research Chemicals Inc. | T792900 | MTSET; hydrolized by water. Keep as dry pouder aliquot at -80 °C. Use immediately (30 minutes) after solubilization in buffer. |
| 1 ml gas-tight syringe | Hamilton | #1001 | |
| 10 ml round flask | Schott Duran | ||
| 2.7 mm glas beads | Roth | N032.1 | |
| 2-Propanole | Roth | 9866.5 | |
| 30 cm Luer-Lock Extension Tube | Sarstedt | 744304 | |
| Acetone | Roth | 5025.5 | |
| Bio-Beads SM-2 Adsorbent | Bio-Rad | 152-3920 | need to be activated before first use |
| CaCl2 Dihydrat | Roth | HN04.3 | |
| Calcein | Sigma | C0875 | store dark at -20 °C |
| Chloroform reagent grade | VWR Chemicals | 22711324 | |
| DOPEATTO390 | ATTO-TEC | AD 390-165 | store dark at -20 °C |
| Ethanol absolute | Sigma-Aldrich | 32205 | |
| Injekt Single-use syringe | Braun | 460 60 51V | |
| Injekt-F single-use syringe | Braun | 91 66 017V | |
| Keck clips | Schott | KC29 | |
| L-α-Phosphatidylcholine, 20% (Soy) | Avanti Polar Lipids | 5416016 | store under inert gas at -20 °C |
| NaCl 99.5% p.a. | Roth | 3957.2 | |
| Nanopore E100 wafer/chips | Micromotive (Mainz/Germany) | available on request | |
| Nucleopore Track-Etch Membrane 0.4 µm | Whatman | 800282 | |
| Oregon Green Dextran 488 (70 kDa) | life Technologies | D-7173 | store dark at -20 °C |
| Oy647 | Luminartis (Münster/Germany) | OY-647-T-1mg | store dark at -20 °C |
| Rotilabo-syringe filtration, unsterile, pore-size 0.22 µm | Roth | P 818.1 | |
| Sephadex G-50 | Sigma-Aldrich | G5080 | column material for size exclusion chromatography |
| Silastic MDX4-4210 | Dow Corning | curing agent for chip fixation onto cover glass support | |
| sticky-Slide 8-well | ibidi | 80828 | multi-well chamber for the mounting onto glass slides (chip holder) |
| Three-way stopcock blue | Sarstedt | 744410001 | |
| Tris Pufferan 99.9% Ultra Quality | Roth | 5429.2 | |
| Triton-X 100 | Roth | 6683.1 | |
| Whatman 0.2 µm cellulose nitrate membrane filter | Roth | NH69.1 | |
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| Büchi 461 water bath | Büchi | ||
| Büchi Rotavapor RE 111 | Büchi | ||
| Cary Eclipse Fluorescence Spectrophtometer | Varian | ||
| LiposoFast Mini Extruder | Avestin | ||
| Membrane pump | Vaccubrand | 15430 | |
| Nanosight Nanoparticle Tracking Microscope | Malvern / Nanosight | LM 14C | |
| NyONE microscope | Synentec | available on request | |
| Pump control | Vaccubrand | CVC 2II | |
| Sonicator bath Sonorex RK100H | Brandelin electronic | 31200001107477 | |
| Vaccum pump RC5 | Vaccubrand | 1805400204 | |
| Water bath W13 | Haake | 002-9910 | |
| Plasma Cleaner PDC-37G | Harrick Plasma | PDC-37G | |
| Name | Company | Catalog Number | Comments |
| Software | |||
| ImageJ | Open Source | http://imagej.nih.gov/ij/ | scientific image processing software |
| NanoCalcFX | Freeware | http://sourceforge.net/projects/nanocalc/ | data analysis/evaluation software for massive transport kinetic datasets |
| NTA 2.3 Analytical Software | Nanosight | data acquisition and analysis software for nanoparticle tracking microscope | |
| NTA 2.3 Temperature Comms | Nanosight | temperature controle software for nanoparticle tracking microscope |
References
- Yildirim, M. A., Goh, K. I., Cusick, M. E., Barabasi, A. L., Vidal, M. Drug-target network. Nat Biotechnol. 25, 1119 (2007).
- Hamill, O. P., Marty, A., Neher, E., Sakmann, B., Sigworth, F. Improved patch-clamp techniques for high-resolution current recording from cells and cell-free membrane patches. Pflugers Arch. 391, 85-100 (1981).
- Osaki, T., Suzuki, H., Le Pioufle, B., Takeuchi, S. Multichannel simultaneous measurements of single-molecule translocation in α-hemolysin nanopore array. Anal. Chem. 81, 9866-9870 (2009).
- Carrillo, L., et al. High-resolution membrane capacitance measurements for studying endocytosis and exocytosis in yeast. Traffic. , (2015).
- Giacomini, K. M., et al. Membrane transporters in drug development. Nat. Rev. Drug Discov. 9, 215-236 (2010).
- Castell, O. K., Berridge, J., Wallace, M. I. Quantification of membrane protein inhibition by optical ion flux in a droplet interface bilayer array. Angew. Chem. Int. Ed. 51, 3134-3138 (2012).
- Zollmann, T., et al. Single liposome analysis of peptide translocation by the ABC transporter TAPL. Proc. Natl. Acad. Sci. U.S.A. 112, 2046-2051 (2015).
- Tamm, L. K., McConnell, H. M. Supported phospholipid bilayers. Biophys. J. 47, 105-113 (1985).
- Sackmann, E. Supported membranes: scientific and practical applications. Science. 271, 43-48 (1996).
- Castellana, E. T., Cremer, P. S. Solid supported lipid bilayers: From biophysical studies to sensor design. Surf. Sci. Rep. 61, 429-444 (2006).
- Wagner, M. L., Tamm, L. K. Tethered polymer-supported planar lipid bilayers for reconstitution of integral membrane proteins: silane-polyethyleneglycol-lipid as a cushion and covalent linker. Biophys. J. 79, 1400-1414 (2000).
- Naumann, C. A., et al. The polymer-supported phospholipid bilayer: tethering as a new approach to substrate-membrane stabilization. Biomacromolecules. 3, 27-35 (2002).
- Weiskopf, D., Schmitt, E. K., Klühr, M. H., Dertinger, S. K., Steinem, C. Micro-BLMs on highly ordered porous silicon substrates: Rupture process and lateral mobility. Langmuir. 23, 9134-9139 (2007).
- Watanabe, R., et al. Arrayed lipid bilayer chambers allow single-molecule analysis of membrane transporter activity. Nat. Commun. 5, (2014).
- Stamou, D., Duschl, C., Delamarche, E., Vogel, H. Self-Assembled Microarrays of Attoliter Molecular Vessels. Angew. Chem. Int. Ed. 115, 5738-5741 (2003).
- Lohr, C., et al. Single Liposomes Used to Study the Activity of Individual Transporters. Biophysical Journal. 106, 229a (2014).
- Dunlop, J., Bowlby, M., Peri, R., Vasilyev, D., Arias, R. High-throughput electrophysiology: an emerging paradigm for ion-channel screening and physiology. Nat. Rev. Drug Discov. 7, 358-368 (2008).
- Milligan, C. J., et al. Robotic multiwell planar patch-clamp for native and primary mammalian cells. Nat. Protoc. 4, 244-255 (2009).
- Soga, N., Watanabe, R., Noji, H. Attolitre-sized lipid bilayer chamber array for rapid detection of single transporters. Sci. Rep. 5, (2015).
- Kleefen, A., et al. Multiplexed parallel single transport recordings on nanopore arrays. Nano Lett. 10, 5080-5087 (2010).
- Wei, R., Gatterdam, V., Wieneke, R., Tampé, R., Rant, U. Stochastic sensing of proteins with receptor-modified solid-state nanopores. Nat. Nanotechnol. 7, 257-263 (2012).
- Urban, M., et al. Highly parallel transport recordings on a membrane-on-nanopore chip at single molecule resolution. Nano Lett. 14, 1674-1680 (2014).
- Kusters, I., Van Oijen, A. M., Driessen, A. J. Membrane-on-a-Chip: Microstructured Silicon/Silicon-Dioxide Chips for High-Throughput Screening of Membrane Transport and Viral Membrane Fusion. ACS Nano. 8, 3380-3392 (2014).
- Hansen, J. S., Thompson, J. R., Hélix-Nielsen, C., Malmstadt, N. Lipid directed intrinsic membrane protein segregation. J. Am. Chem. Soc. 135, 17294-17297 (2013).
- Heinemann, F., Schwille, P. Preparation of Micrometer-Sized Free-Standing Membranes. ChemPhysChem. 12, 2568-2571 (2011).
- Lazzara, T. D., Carnarius, C., Kocun, M., Janshoff, A., Steinem, C. Separating attoliter-sized compartments using fluid pore-spanning lipid bilayers. ACS nano. 5, 6935-6944 (2011).
- Winterhalter, M. Black lipid membranes. Curr. Opin. Colloid Interface Sci. 5, 250-255 (2000).
- Koçer, A., Walko, M., Feringa, B. L. Synthesis and utilization of reversible and irreversible light-activated nanovalves derived from the channel protein MscL. Nat. Protoc. 2, 1426-1437 (2007).

