Trasporto di membrana processi analizzati da un altamente parallelo sistema Nanopore Chip a risoluzione della proteina singolo
Summary
The presented protocol describes the analysis of membrane protein mediated transport on the single transporter level using pore-spanning solvent-free lipid bilayers. This is achieved by the creation of bulk produced nanopore array chips, combined with highly parallel data acquisition and analysis, enabling the future establishment of membrane protein effector screenings.
Abstract
trasporto proteina di membrana a livello della proteina singolo elude ancora un'analisi dettagliata, se il substrato traslocato non è elettrogenico. Notevoli sforzi sono stati fatti in questo campo, ma le tecniche che permettono automatizzato di analisi trasporto ad alta produttività in combinazione con lipidi tecniche di doppio strato privi di solventi necessari per l'analisi dei trasportatori di membrana sono rari. Questa classe di trasportatori, tuttavia, è cruciale nella omeostasi cellulare e quindi un obiettivo chiave nello sviluppo di farmaci e metodologie per acquisire nuove conoscenze disperatamente bisogno.
Il manoscritto qui presentato descrive la creazione e la gestione di un romanzo biochip per l'analisi di proteine mediata processi di trasporto di membrana con risoluzione singolo trasportatore. Il biochip è composto da microcavità racchiuse da nanopori che è altamente parallelo nel suo design e possono essere prodotti in grado industriale e quantità. liposomi Protein-nutrirebbero possono essere direttamente applicati aila superficie del chip che formano doppi strati lipidici poro-spanning auto-assemblate utilizzando SSM-tecniche (solido supportato membrane lipidiche). Pore-attraversa parti della membrana sono indipendenti, fornendo l'interfaccia per substrato traslocazione dentro o fuori lo spazio della cavità, che può essere seguita da lettura fluorescente multispettrale in tempo reale. La definizione di procedure operative standard (SOP) permette la creazione diretta di doppi strati lipidici proteine ospitare sulla superficie del chip di proteine virtualmente ogni membrana che può essere ricostituito in modo funzionale. L'unico requisito è la creazione di un sistema di read-out fluorescente per substrati di trasporto non elettrogeniche.
applicazioni ad alto contenuto di screening sono attuabile mediante l'uso di microscopi a fluorescenza automatizzati invertiti registrazione chip multipli in parallelo. Grandi insiemi di dati possono essere analizzati utilizzando il software di analisi custom-designed liberamente disponibile. Tre colori multi spettrale fluorescenteread-out, inoltre, consente una discriminazione dati imparziale in diverse classi di eventi, eliminando falsi risultati positivi.
La tecnologia dei chip si basa attualmente su SiO 2 superfici, ma un'ulteriore funzionalizzazione con superfici di chip d'oro rivestite è inoltre possibile.
Introduction
L'analisi delle proteine di membrana è diventato di crescente interesse per la ricerca di base e farmaceutica negli ultimi 20 anni. Lo sviluppo di nuovi farmaci dipende identificazione e caratterizzazione dettagliata di nuovi bersagli, attualmente essendo uno dei fattori limitanti. Il fatto che circa il 60% di tutti i bersagli farmacologici sono proteine di membrana 1, rende lo sviluppo di tecniche per chiarire la loro funzione più importante.
In passato, le tecniche per lo studio dei canali elettrogeniche e trasportatori sono stati sviluppati in moltitudine di 2 – 4. substrati non elettrogeniche in contrario presentano un compito più impegnativo. Sono comunque di particolare interesse come obiettivi primari droga, come controllano il flusso di soluti e nutrienti attraverso la membrana cellulare e la funzione come recettori chiave in cascate di segnalazione 5.
considerevole sforzo è stato messo nello sviluppo di techniques per studiare la funzione di trasportatore di membrana 6, 7. I sistemi che utilizzano membrane solidi supportati sono emersi come strumenti più promettenti in questo campo 8 – 10, compreso solidi doppi strati lipidici supportati, bistrati frenati 11, 12, membrane lipidiche microblack 13, 14 e nativi array vescicola 15, 16 solo per citarne alcuni. Alcuni di loro sono anche disponibili come configurazioni commerciali 17, 18. Alcuni esempi sono stati pubblicati combinando la capacità di studiare singole proteine di membrana in modo altamente paralleli 14, 19, un prerequisito per applicazioni di screening. Tuttavia, questi metodi raramente colmare dalla ricerca di base per l'ambiente industriale. Le difficoltà spesso trovano nella capacità del sistema di essere automatizzabili, la produzione costose e / o preparazione laboriosa. Un approccio overcoming tutti gli ostacoli di cui sopra è l'obiettivo finale.
La tecnica qui presentata è stato sviluppato per studiare canali di membrana e trasportatori in vitro in un ambiente controllato al livello proteico singola 20 – 22. Ricostituzione di proteine di membrana purificate in luvs è molto più stabilito di approcci comparabili per GUVs 23 – 26 o lipidi nero membrane 27. Essi possono essere applicati direttamente alla superficie del chip, dove la formazione doppio strato avviene attraverso un processo di auto-assemblaggio. Il design fondo di vetro del chip nanoporoso (Fig. 1) permette per microscopia dell'aria, che permette l'automazione semplice del sistema. In combinazione con uno stadio motorizzato chip multipli possono essere misurate allo stesso tempo, con ogni campo di vista contenente migliaia di cavità sigillate per l'analisi.
<p class="jove_content" fo:keep-together.within-page= "1">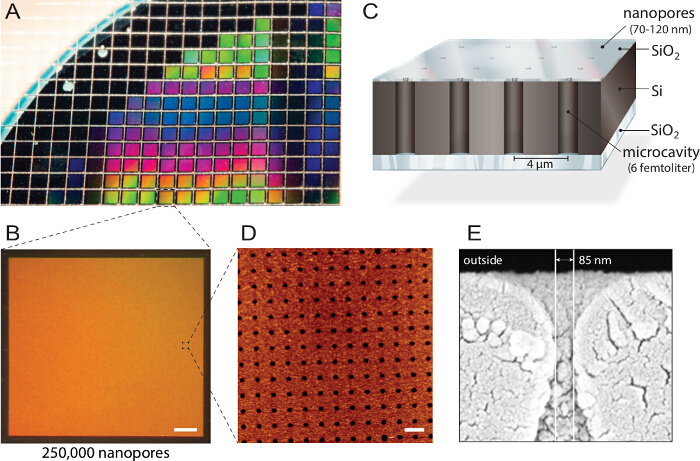
Figura 1. Progettazione di biochip nanopori multiplex. A) una Silicon-on-Insulator (SOI) wafer è strutturata da incisione reattiva ioni di litio. Circa 1.150 singoli chip sono fabbricati da ogni wafer con proprietà identiche e qualità. B) Ogni chip comprende 250.000 microcavità individuali con aperture nano. Barra di scala:. 200 micron C) Ogni cavità è indirizzabile tramite fluorescenza multispettrale read-out. Un blocchi strato superiore non trasparenti i segnali fluorescenti dal serbatoio tampone, rendendo il biochip compatibile con microscopi a fluorescenza invertiti. D) microscopia a forza atomica (AFM) di imaging rivela organizzati uniformemente aperture dei pori e la rugosità superficiale dello strato di biossido di silicio di 3,6 nm (n = 40) ottimale per la fusione delle vescicole. Barra di scala:. 5 micron E) mic elettronico a scansioneimmagine roscopy (SEM) mostra una sezione trasversale attraverso il nanoporo consentendo l'accesso alla cavità femtoliter all'interno del chip di silicio. Questo dato è stato riutilizzato con il permesso di 21. Si prega di cliccare qui per vedere una versione più grande di questa figura.
Tutte le analisi dei dati viene effettuata utilizzando freeware per garantire l'accesso senza restrizioni per gli utenti finali. Le serie temporali vengono analizzati utilizzando il software gratuito di elaborazione delle immagini e di una elaborazione batch curva di generazione personalizzata software di analisi che permette e la correlazione diretta di dati di grandi dimensioni con canali fluorescenti multiple e migliaia di curve.
La proteina modello utilizzato in questo protocollo è il canale meccanosensibili di proteine grande conduttanza (MscL) canale derivato da E. coli. Esso funziona come una valvola per rilasciare shock osmotico in natura, ma è stato modificato in modo tale che razionalmente progettato syntfunzionalità hetic possono covalentemente essere fissati al cielo canali costrizione. Via carica-repulsione dell'attivatore legame covalente (MTSET) il canale viene attivato per aprire, la creazione di un nano-valvola. Piccole molecole come ioni, acqua, piccole proteine, ma anche piccoli fluorofori possono permeare attraverso il canale. Qui, la proteina viene utilizzato come modello per dimostrare la capacità del sistema di rilevare la traslocazione di proteine mediata.
Protocol
Representative Results
Discussion
La tecnica qui presentata permette un'analisi altamente parallela di trasporto delle proteine di membrana. sistemi proteici di membrana ricostituiti possono essere direttamente applicati al biochip, rendendo l'adattamento teoricamente ogni trasportatore di membrana o canale possibile. Analisi dei trasporti è limitata solo dalla creazione di un sistema di read-out a fluorescenza, sia attraverso il passaggio diretto di fluorescenza (traslocazione di fluorofori o substrati fluorescente) o il cambiamento di f…
Disclosures
The authors have nothing to disclose.
Acknowledgements
We thank Barbara Windschiegl for her help in establishing SOPs; Dennis Remme for his work on the NanoCalcFX software and Alina Kollmannsperger, Markus Braner and Milan Gerovac for helpful suggestions on the manuscript. The German-Israeli Project Cooperation (DIP) provided by the DFG and the Federal Ministry of Education and Research to R.T., as well as the Federal Ministry of Economics and Technology (ZIM R&D Project) to R.T. and Nanospot GmbH supported this work.
Materials
| Reagent | |||
| [2-(Trimethylammonium)ethyl] methanethiosulfonate |
Toronto Research Chemicals Inc. | T792900 | MTSET; hydrolized by water. Keep as dry pouder aliquot at -80 °C. Use immediately (30 minutes) after solubilization in buffer. |
| 1 ml gas-tight syringe | Hamilton | #1001 | |
| 10 ml round flask | Schott Duran | ||
| 2.7 mm glas beads | Roth | N032.1 | |
| 2-Propanole | Roth | 9866.5 | |
| 30 cm Luer-Lock Extension Tube | Sarstedt | 744304 | |
| Acetone | Roth | 5025.5 | |
| Bio-Beads SM-2 Adsorbent | Bio-Rad | 152-3920 | need to be activated before first use |
| CaCl2 Dihydrat | Roth | HN04.3 | |
| Calcein | Sigma | C0875 | store dark at -20 °C |
| Chloroform reagent grade | VWR Chemicals | 22711324 | |
| DOPEATTO390 | ATTO-TEC | AD 390-165 | store dark at -20 °C |
| Ethanol absolute | Sigma-Aldrich | 32205 | |
| Injekt Single-use syringe | Braun | 460 60 51V | |
| Injekt-F single-use syringe | Braun | 91 66 017V | |
| Keck clips | Schott | KC29 | |
| L-α-Phosphatidylcholine, 20% (Soy) | Avanti Polar Lipids | 5416016 | store under inert gas at -20 °C |
| NaCl 99.5% p.a. | Roth | 3957.2 | |
| Nanopore E100 wafer/chips | Micromotive (Mainz/Germany) | available on request | |
| Nucleopore Track-Etch Membrane 0.4 µm | Whatman | 800282 | |
| Oregon Green Dextran 488 (70 kDa) | life Technologies | D-7173 | store dark at -20 °C |
| Oy647 | Luminartis (Münster/Germany) | OY-647-T-1mg | store dark at -20 °C |
| Rotilabo-syringe filtration, unsterile, pore-size 0.22 µm | Roth | P 818.1 | |
| Sephadex G-50 | Sigma-Aldrich | G5080 | column material for size exclusion chromatography |
| Silastic MDX4-4210 | Dow Corning | curing agent for chip fixation onto cover glass support | |
| sticky-Slide 8-well | ibidi | 80828 | multi-well chamber for the mounting onto glass slides (chip holder) |
| Three-way stopcock blue | Sarstedt | 744410001 | |
| Tris Pufferan 99.9% Ultra Quality | Roth | 5429.2 | |
| Triton-X 100 | Roth | 6683.1 | |
| Whatman 0.2 µm cellulose nitrate membrane filter | Roth | NH69.1 | |
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| Büchi 461 water bath | Büchi | ||
| Büchi Rotavapor RE 111 | Büchi | ||
| Cary Eclipse Fluorescence Spectrophtometer | Varian | ||
| LiposoFast Mini Extruder | Avestin | ||
| Membrane pump | Vaccubrand | 15430 | |
| Nanosight Nanoparticle Tracking Microscope | Malvern / Nanosight | LM 14C | |
| NyONE microscope | Synentec | available on request | |
| Pump control | Vaccubrand | CVC 2II | |
| Sonicator bath Sonorex RK100H | Brandelin electronic | 31200001107477 | |
| Vaccum pump RC5 | Vaccubrand | 1805400204 | |
| Water bath W13 | Haake | 002-9910 | |
| Plasma Cleaner PDC-37G | Harrick Plasma | PDC-37G | |
| Name | Company | Catalog Number | Comments |
| Software | |||
| ImageJ | Open Source | http://imagej.nih.gov/ij/ | scientific image processing software |
| NanoCalcFX | Freeware | http://sourceforge.net/projects/nanocalc/ | data analysis/evaluation software for massive transport kinetic datasets |
| NTA 2.3 Analytical Software | Nanosight | data acquisition and analysis software for nanoparticle tracking microscope | |
| NTA 2.3 Temperature Comms | Nanosight | temperature controle software for nanoparticle tracking microscope |
References
- Yildirim, M. A., Goh, K. I., Cusick, M. E., Barabasi, A. L., Vidal, M. Drug-target network. Nat Biotechnol. 25, 1119 (2007).
- Hamill, O. P., Marty, A., Neher, E., Sakmann, B., Sigworth, F. Improved patch-clamp techniques for high-resolution current recording from cells and cell-free membrane patches. Pflugers Arch. 391, 85-100 (1981).
- Osaki, T., Suzuki, H., Le Pioufle, B., Takeuchi, S. Multichannel simultaneous measurements of single-molecule translocation in α-hemolysin nanopore array. Anal. Chem. 81, 9866-9870 (2009).
- Carrillo, L., et al. High-resolution membrane capacitance measurements for studying endocytosis and exocytosis in yeast. Traffic. , (2015).
- Giacomini, K. M., et al. Membrane transporters in drug development. Nat. Rev. Drug Discov. 9, 215-236 (2010).
- Castell, O. K., Berridge, J., Wallace, M. I. Quantification of membrane protein inhibition by optical ion flux in a droplet interface bilayer array. Angew. Chem. Int. Ed. 51, 3134-3138 (2012).
- Zollmann, T., et al. Single liposome analysis of peptide translocation by the ABC transporter TAPL. Proc. Natl. Acad. Sci. U.S.A. 112, 2046-2051 (2015).
- Tamm, L. K., McConnell, H. M. Supported phospholipid bilayers. Biophys. J. 47, 105-113 (1985).
- Sackmann, E. Supported membranes: scientific and practical applications. Science. 271, 43-48 (1996).
- Castellana, E. T., Cremer, P. S. Solid supported lipid bilayers: From biophysical studies to sensor design. Surf. Sci. Rep. 61, 429-444 (2006).
- Wagner, M. L., Tamm, L. K. Tethered polymer-supported planar lipid bilayers for reconstitution of integral membrane proteins: silane-polyethyleneglycol-lipid as a cushion and covalent linker. Biophys. J. 79, 1400-1414 (2000).
- Naumann, C. A., et al. The polymer-supported phospholipid bilayer: tethering as a new approach to substrate-membrane stabilization. Biomacromolecules. 3, 27-35 (2002).
- Weiskopf, D., Schmitt, E. K., Klühr, M. H., Dertinger, S. K., Steinem, C. Micro-BLMs on highly ordered porous silicon substrates: Rupture process and lateral mobility. Langmuir. 23, 9134-9139 (2007).
- Watanabe, R., et al. Arrayed lipid bilayer chambers allow single-molecule analysis of membrane transporter activity. Nat. Commun. 5, (2014).
- Stamou, D., Duschl, C., Delamarche, E., Vogel, H. Self-Assembled Microarrays of Attoliter Molecular Vessels. Angew. Chem. Int. Ed. 115, 5738-5741 (2003).
- Lohr, C., et al. Single Liposomes Used to Study the Activity of Individual Transporters. Biophysical Journal. 106, 229a (2014).
- Dunlop, J., Bowlby, M., Peri, R., Vasilyev, D., Arias, R. High-throughput electrophysiology: an emerging paradigm for ion-channel screening and physiology. Nat. Rev. Drug Discov. 7, 358-368 (2008).
- Milligan, C. J., et al. Robotic multiwell planar patch-clamp for native and primary mammalian cells. Nat. Protoc. 4, 244-255 (2009).
- Soga, N., Watanabe, R., Noji, H. Attolitre-sized lipid bilayer chamber array for rapid detection of single transporters. Sci. Rep. 5, (2015).
- Kleefen, A., et al. Multiplexed parallel single transport recordings on nanopore arrays. Nano Lett. 10, 5080-5087 (2010).
- Wei, R., Gatterdam, V., Wieneke, R., Tampé, R., Rant, U. Stochastic sensing of proteins with receptor-modified solid-state nanopores. Nat. Nanotechnol. 7, 257-263 (2012).
- Urban, M., et al. Highly parallel transport recordings on a membrane-on-nanopore chip at single molecule resolution. Nano Lett. 14, 1674-1680 (2014).
- Kusters, I., Van Oijen, A. M., Driessen, A. J. Membrane-on-a-Chip: Microstructured Silicon/Silicon-Dioxide Chips for High-Throughput Screening of Membrane Transport and Viral Membrane Fusion. ACS Nano. 8, 3380-3392 (2014).
- Hansen, J. S., Thompson, J. R., Hélix-Nielsen, C., Malmstadt, N. Lipid directed intrinsic membrane protein segregation. J. Am. Chem. Soc. 135, 17294-17297 (2013).
- Heinemann, F., Schwille, P. Preparation of Micrometer-Sized Free-Standing Membranes. ChemPhysChem. 12, 2568-2571 (2011).
- Lazzara, T. D., Carnarius, C., Kocun, M., Janshoff, A., Steinem, C. Separating attoliter-sized compartments using fluid pore-spanning lipid bilayers. ACS nano. 5, 6935-6944 (2011).
- Winterhalter, M. Black lipid membranes. Curr. Opin. Colloid Interface Sci. 5, 250-255 (2000).
- Koçer, A., Walko, M., Feringa, B. L. Synthesis and utilization of reversible and irreversible light-activated nanovalves derived from the channel protein MscL. Nat. Protoc. 2, 1426-1437 (2007).

