Membran Transport Tek Protein çözünürlükte Oldukça Paralel nanopore çip Sistemi Analiz Süreçleri
Summary
The presented protocol describes the analysis of membrane protein mediated transport on the single transporter level using pore-spanning solvent-free lipid bilayers. This is achieved by the creation of bulk produced nanopore array chips, combined with highly parallel data acquisition and analysis, enabling the future establishment of membrane protein effector screenings.
Abstract
transloke substrat olmayan electrogenic ise tek bir protein düzeyinde membran proteini taşıma hala detaylı analizler kaçıyor. Kaydadeğer çaba bu alanda yapılmış, ancak zar taşıyıcıları analizi için gerekli olan solvent içermeyen çift katlı lipid teknikleri ile birlikte otomatik yüksek verimli taşıma analizi sağlayan teknikler nadirdir. taşıyıcıların Bu sınıf, ancak hücre homeostazında önemli ve bu nedenle ilaç geliştirme ve yeni anlayışlar kazanmak için metodolojiler önemli bir hedef umutsuzca ihtiyaç olduğunu.
Burada sunulan el yazması tek taşıyıcı çözünürlükte membran proteini aracılı taşıma süreçlerinin analizi için yeni bir Biochip kurulmasını ve kullanımını açıklar. biyoçip tasarımında oldukça paraleldir ve endüstriyel cins ve miktarda üretilebilir nanopores çevrelediği, mikro boşlukların üst oluşmaktadır. Protein Barınma lipozomlar doğrudan uygulanabilirSSM-teknikleri kullanarak kendini monte gözenek kapsayan çift katlı lipit katmanını oluşturan yonga yüzeyi (katı lipid membranlar desteklenir). membran parçaları, müstakil içine veya gerçek zamanlı multi-spektral floresan okuma ile takip edilebilir kavite alanı, dışarı substrat translokasyonu için arayüz sağlıyor Gözenek kapsayan. standart operasyon prosedürleri (SOP) kurulması işlevsel yeniden olabilir hemen hemen her membran proteini çip yüzeyinde protein barındıran lipit tabakalarının kolay kurulmasını sağlar. tek ön şart olmayan electrogenic taşıma yüzeyler için bir floresan okuma-out sistemi kurulmasıdır.
Yüksek içerik tarama uygulamaları paralel olarak birden fazla fiş kayıt otomatik ters floresan mikroskop kullanılarak accomplishable vardır. Büyük veri kümeleri serbestçe kullanılabilir özel tasarlanmış bilgisayar programları kullanılarak analiz edilebilir. Üç renkli çoklu spektral floresanOkuması ayrıca yanlış pozitif sonuçlar ortadan kaldırarak, farklı olay sınıflarına tarafsız veri ayrımcılık sağlar.
Chip teknolojisi şu anda SiO 2 yüzeylere göre, ama altın kaplamalı yonga yüzeyleri kullanılarak daha fonksiyonlandırmalar da mümkündür edilir.
Introduction
membran proteinlerinin analizi son 20 yılda temel ve ilaç araştırmaları için artan ilgi haline gelmiştir. yeni ilaçların geliştirilmesi halen kısıtlayan faktörlerden biri olan tanımlanması ve yeni hedefler detaylı karakterizasyonu bağlıdır. Tüm ilaç hedefleri yaklaşık% 60 zar proteinleri 1 olması, tekniklerin geliştirilmesi fonksiyonları önemli aydınlatmak için yapar.
4 – Geçmişte, electrogenic kanalları ve taşıyıcıları çalışma teknikleri 2 sayıda geliştirilmiştir. Aksine olmayan electrogenic yüzeyler daha zorlu bir görev sunuyoruz. Onlar kaskadlar 5 sinyal kilit reseptörler olarak hücre zarı ve fonksiyonu üzerinde çözünen ve besin akışını kontrol gibi onlar, ancak ana ilaç hedefleri olarak özel bir öneme sahiptir.
Önemli çaba t gelişimi girmiştirtekniki membran transport proteinleri 6, 7 işlevini incelemek için. Birkaç isim katı desteklenen lipid bilayers, gergin bilayers 11, 12, microblack lipit membranlar 13, 14 ve yerli vezikül dizileri 15, 16 olmak üzere 10, – Katı destekli membranlar kullanılarak Sistemleri bu alanda 8 olarak en umut verici araçlar ortaya çıkmıştır. Bazıları ticari kurulumları 17, 18 hatta mevcuttur. Bazı örnekler son derece paralel bir şekilde 14, 19, tarama uygulamaları için bir ön koşul olarak, tek zar proteinleri çalışma yeteneği birleştiren yayınlanmıştır. Ancak, bu yöntemler nadiren endüstriyel ortamlarda temel araştırma köprü. güçlükler çoğu otomatikleştirilebilir için sistemin özelliği, yüksek maliyetli üretim ve / veya zahmetli hazırlanmasında uzanmaktadır. Bir yaklaşım oYukarıda açıklanan tüm engelleri aşılabilir nihai amaçtır.
22 – Burada sunulan teknik tek bir protein düzeyinde 20 kontrollü bir ortamda in vitro membran kanalları ve taşıyıcıları incelemek için geliştirilmiştir. 26 ya da siyah lipit membranlar 27 – luvs içine saflaştırılmış membran proteinlerinin Sulandırma daha GUVs 23 karşılaştırılabilir yaklaşımlara göre kurulmuştur. Direkt olarak iki katmanlı oluşum şeklinde kendini montaj işlemi ile gerçekleşiyor yonga yüzeyi, uygulanabilir. Nano-gözenekli çip (Şek. 1) ve cam alt tasarım sistemi basit otomasyon izin hava mikroskopi için izin verir. motorlu bir sahne ile birlikte çoklu cips analiz için mühürlü boşlukların binlerce içeren görüş her alanı ile aynı anda ölçülebilir.
<p class="jove_content" fo:keep-together.within-page= "1">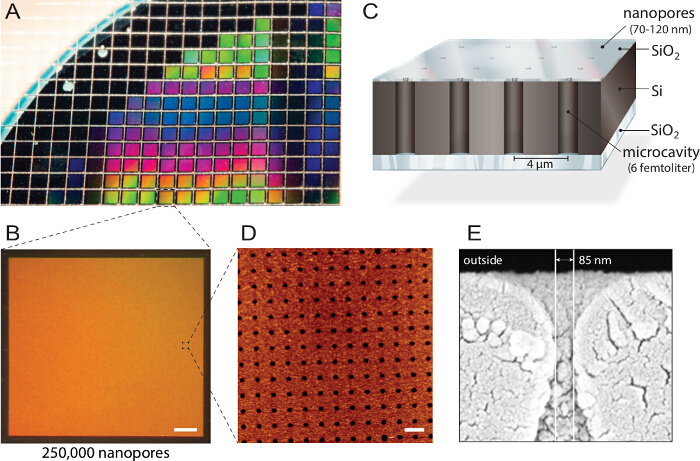
Şekil 1. Çoklanmış nanopore bioçipler tasarımı. A) silikon üzerinde yalıtkan (SOI) gofret reaktif iyon dağlama ile yapılandırılmıştır. Yaklaşık 1,150 ayrı cips aynı özellikleri ve kalite. B) Her çip nano delikler ile 250.000 bireysel, mikro içermektedir her gofret imal edilmektedir. Ölçek çubuğu:. 200 mikron C) Her oyuk multi-spektral floresan okuma-out üzerinden adreslenebilir. Ters floresan mikroskobu. D) Atomik kuvvet mikroskobu ile biyoçip uyumlu hale bir şeffaf üst tabaka blokları tampon hazneden floresan sinyalleri (AFM) görüntüleme eşit (gözenek açıklıkları ve 3.6 nm silikon dioksit tabakasının yüzey pürüzlülüğü düzenlenmiş ortaya koymaktadır n = vezikül füzyon 40) optimum. Ölçek çubuğu:. 5 mikron E) Taramalı elektron mikrofonfloroskop (SEM) görüntüsü silikon çip içinde femtoliter boşluklara erişim sağlayan nano-gözeneklere enine kesitini göstermektedir. Bu rakam 21 izni ile yeniden yapıldı. Bu rakamın büyük halini görmek için lütfen buraya tıklayınız.
Tüm veri analizi, son kullanıcılar için sınırsız erişim sağlamak için ücretsiz kullanarak gerçekleştirilir. Zaman serisi ücretsiz görüntü işleme yazılımları ve özel bir yapı eğrisi analiz yazılımı sağlayan toplu işleme ve eğrilerin çoklu floresan kanalları ve binlerce büyük veri kümelerinin basit korelasyon kullanılarak analiz edilmektedir.
Bu protokolde kullanılan model proteini E. türetilen büyük iletkenlik (MSCL) kanal proteinin mechanosensitive kanal E. coli. Doğada ozmotik şok serbest bırakmak için bir valf olarak davranır, fakat rasyonel SYNT tasarlanmış bir şekilde değiştirildiinfographie işlevleri kovalent kanal daralma tarafına monte edilebilir. Nano-valf oluşturarak kanal açmak için tetiklenir kovalent bağlı aktivatör (MTSET) şarj-itme Via. iyonları, su, küçük proteinler, aynı zamanda küçük fluorophores gibi küçük moleküller kanalından nüfuz edebilir. Burada, bir protein, protein aracılı translokasyon algılamak üzere yeteneğini göstermek için bir model olarak kullanılır.
Protocol
Representative Results
Discussion
Burada sunulan teknik membran proteini ulaşım son derece paralel analiz sağlar. Sulandırılmış membran proteini sistemleri doğrudan teorik her zar taşıyıcı uyarlanmasını yapmadan veya olası kanala, Biochip uygulanabilir. Taşıma analizi sadece doğrudan floresans değişimi (fluorophores translokasyon veya floresan etiketli substratlar) ya da dolaylı olarak floresans değişimi (pH'a duyarlı boyalar, sekonder enzimatik reaksiyonlar) yoluyla, bir flüoresan okuması sisteminin kurulması ile sınırl…
Disclosures
The authors have nothing to disclose.
Acknowledgements
We thank Barbara Windschiegl for her help in establishing SOPs; Dennis Remme for his work on the NanoCalcFX software and Alina Kollmannsperger, Markus Braner and Milan Gerovac for helpful suggestions on the manuscript. The German-Israeli Project Cooperation (DIP) provided by the DFG and the Federal Ministry of Education and Research to R.T., as well as the Federal Ministry of Economics and Technology (ZIM R&D Project) to R.T. and Nanospot GmbH supported this work.
Materials
| Reagent | |||
| [2-(Trimethylammonium)ethyl] methanethiosulfonate |
Toronto Research Chemicals Inc. | T792900 | MTSET; hydrolized by water. Keep as dry pouder aliquot at -80 °C. Use immediately (30 minutes) after solubilization in buffer. |
| 1 ml gas-tight syringe | Hamilton | #1001 | |
| 10 ml round flask | Schott Duran | ||
| 2.7 mm glas beads | Roth | N032.1 | |
| 2-Propanole | Roth | 9866.5 | |
| 30 cm Luer-Lock Extension Tube | Sarstedt | 744304 | |
| Acetone | Roth | 5025.5 | |
| Bio-Beads SM-2 Adsorbent | Bio-Rad | 152-3920 | need to be activated before first use |
| CaCl2 Dihydrat | Roth | HN04.3 | |
| Calcein | Sigma | C0875 | store dark at -20 °C |
| Chloroform reagent grade | VWR Chemicals | 22711324 | |
| DOPEATTO390 | ATTO-TEC | AD 390-165 | store dark at -20 °C |
| Ethanol absolute | Sigma-Aldrich | 32205 | |
| Injekt Single-use syringe | Braun | 460 60 51V | |
| Injekt-F single-use syringe | Braun | 91 66 017V | |
| Keck clips | Schott | KC29 | |
| L-α-Phosphatidylcholine, 20% (Soy) | Avanti Polar Lipids | 5416016 | store under inert gas at -20 °C |
| NaCl 99.5% p.a. | Roth | 3957.2 | |
| Nanopore E100 wafer/chips | Micromotive (Mainz/Germany) | available on request | |
| Nucleopore Track-Etch Membrane 0.4 µm | Whatman | 800282 | |
| Oregon Green Dextran 488 (70 kDa) | life Technologies | D-7173 | store dark at -20 °C |
| Oy647 | Luminartis (Münster/Germany) | OY-647-T-1mg | store dark at -20 °C |
| Rotilabo-syringe filtration, unsterile, pore-size 0.22 µm | Roth | P 818.1 | |
| Sephadex G-50 | Sigma-Aldrich | G5080 | column material for size exclusion chromatography |
| Silastic MDX4-4210 | Dow Corning | curing agent for chip fixation onto cover glass support | |
| sticky-Slide 8-well | ibidi | 80828 | multi-well chamber for the mounting onto glass slides (chip holder) |
| Three-way stopcock blue | Sarstedt | 744410001 | |
| Tris Pufferan 99.9% Ultra Quality | Roth | 5429.2 | |
| Triton-X 100 | Roth | 6683.1 | |
| Whatman 0.2 µm cellulose nitrate membrane filter | Roth | NH69.1 | |
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| Büchi 461 water bath | Büchi | ||
| Büchi Rotavapor RE 111 | Büchi | ||
| Cary Eclipse Fluorescence Spectrophtometer | Varian | ||
| LiposoFast Mini Extruder | Avestin | ||
| Membrane pump | Vaccubrand | 15430 | |
| Nanosight Nanoparticle Tracking Microscope | Malvern / Nanosight | LM 14C | |
| NyONE microscope | Synentec | available on request | |
| Pump control | Vaccubrand | CVC 2II | |
| Sonicator bath Sonorex RK100H | Brandelin electronic | 31200001107477 | |
| Vaccum pump RC5 | Vaccubrand | 1805400204 | |
| Water bath W13 | Haake | 002-9910 | |
| Plasma Cleaner PDC-37G | Harrick Plasma | PDC-37G | |
| Name | Company | Catalog Number | Comments |
| Software | |||
| ImageJ | Open Source | http://imagej.nih.gov/ij/ | scientific image processing software |
| NanoCalcFX | Freeware | http://sourceforge.net/projects/nanocalc/ | data analysis/evaluation software for massive transport kinetic datasets |
| NTA 2.3 Analytical Software | Nanosight | data acquisition and analysis software for nanoparticle tracking microscope | |
| NTA 2.3 Temperature Comms | Nanosight | temperature controle software for nanoparticle tracking microscope |
References
- Yildirim, M. A., Goh, K. I., Cusick, M. E., Barabasi, A. L., Vidal, M. Drug-target network. Nat Biotechnol. 25, 1119 (2007).
- Hamill, O. P., Marty, A., Neher, E., Sakmann, B., Sigworth, F. Improved patch-clamp techniques for high-resolution current recording from cells and cell-free membrane patches. Pflugers Arch. 391, 85-100 (1981).
- Osaki, T., Suzuki, H., Le Pioufle, B., Takeuchi, S. Multichannel simultaneous measurements of single-molecule translocation in α-hemolysin nanopore array. Anal. Chem. 81, 9866-9870 (2009).
- Carrillo, L., et al. High-resolution membrane capacitance measurements for studying endocytosis and exocytosis in yeast. Traffic. , (2015).
- Giacomini, K. M., et al. Membrane transporters in drug development. Nat. Rev. Drug Discov. 9, 215-236 (2010).
- Castell, O. K., Berridge, J., Wallace, M. I. Quantification of membrane protein inhibition by optical ion flux in a droplet interface bilayer array. Angew. Chem. Int. Ed. 51, 3134-3138 (2012).
- Zollmann, T., et al. Single liposome analysis of peptide translocation by the ABC transporter TAPL. Proc. Natl. Acad. Sci. U.S.A. 112, 2046-2051 (2015).
- Tamm, L. K., McConnell, H. M. Supported phospholipid bilayers. Biophys. J. 47, 105-113 (1985).
- Sackmann, E. Supported membranes: scientific and practical applications. Science. 271, 43-48 (1996).
- Castellana, E. T., Cremer, P. S. Solid supported lipid bilayers: From biophysical studies to sensor design. Surf. Sci. Rep. 61, 429-444 (2006).
- Wagner, M. L., Tamm, L. K. Tethered polymer-supported planar lipid bilayers for reconstitution of integral membrane proteins: silane-polyethyleneglycol-lipid as a cushion and covalent linker. Biophys. J. 79, 1400-1414 (2000).
- Naumann, C. A., et al. The polymer-supported phospholipid bilayer: tethering as a new approach to substrate-membrane stabilization. Biomacromolecules. 3, 27-35 (2002).
- Weiskopf, D., Schmitt, E. K., Klühr, M. H., Dertinger, S. K., Steinem, C. Micro-BLMs on highly ordered porous silicon substrates: Rupture process and lateral mobility. Langmuir. 23, 9134-9139 (2007).
- Watanabe, R., et al. Arrayed lipid bilayer chambers allow single-molecule analysis of membrane transporter activity. Nat. Commun. 5, (2014).
- Stamou, D., Duschl, C., Delamarche, E., Vogel, H. Self-Assembled Microarrays of Attoliter Molecular Vessels. Angew. Chem. Int. Ed. 115, 5738-5741 (2003).
- Lohr, C., et al. Single Liposomes Used to Study the Activity of Individual Transporters. Biophysical Journal. 106, 229a (2014).
- Dunlop, J., Bowlby, M., Peri, R., Vasilyev, D., Arias, R. High-throughput electrophysiology: an emerging paradigm for ion-channel screening and physiology. Nat. Rev. Drug Discov. 7, 358-368 (2008).
- Milligan, C. J., et al. Robotic multiwell planar patch-clamp for native and primary mammalian cells. Nat. Protoc. 4, 244-255 (2009).
- Soga, N., Watanabe, R., Noji, H. Attolitre-sized lipid bilayer chamber array for rapid detection of single transporters. Sci. Rep. 5, (2015).
- Kleefen, A., et al. Multiplexed parallel single transport recordings on nanopore arrays. Nano Lett. 10, 5080-5087 (2010).
- Wei, R., Gatterdam, V., Wieneke, R., Tampé, R., Rant, U. Stochastic sensing of proteins with receptor-modified solid-state nanopores. Nat. Nanotechnol. 7, 257-263 (2012).
- Urban, M., et al. Highly parallel transport recordings on a membrane-on-nanopore chip at single molecule resolution. Nano Lett. 14, 1674-1680 (2014).
- Kusters, I., Van Oijen, A. M., Driessen, A. J. Membrane-on-a-Chip: Microstructured Silicon/Silicon-Dioxide Chips for High-Throughput Screening of Membrane Transport and Viral Membrane Fusion. ACS Nano. 8, 3380-3392 (2014).
- Hansen, J. S., Thompson, J. R., Hélix-Nielsen, C., Malmstadt, N. Lipid directed intrinsic membrane protein segregation. J. Am. Chem. Soc. 135, 17294-17297 (2013).
- Heinemann, F., Schwille, P. Preparation of Micrometer-Sized Free-Standing Membranes. ChemPhysChem. 12, 2568-2571 (2011).
- Lazzara, T. D., Carnarius, C., Kocun, M., Janshoff, A., Steinem, C. Separating attoliter-sized compartments using fluid pore-spanning lipid bilayers. ACS nano. 5, 6935-6944 (2011).
- Winterhalter, M. Black lipid membranes. Curr. Opin. Colloid Interface Sci. 5, 250-255 (2000).
- Koçer, A., Walko, M., Feringa, B. L. Synthesis and utilization of reversible and irreversible light-activated nanovalves derived from the channel protein MscL. Nat. Protoc. 2, 1426-1437 (2007).

