Generazione CRISPR / Cas9 Mediated monoallelic eliminazioni per studiare la funzione Enhancer nel topo cellule staminali embrionali
Summary
Experimental validation of enhancer activity is best approached by loss-of-function analysis. Presented here is an efficient protocol that uses CRISPR/Cas9 mediated deletion to study allele-specific regulation of gene transcription in F1 ES cells which contain a hybrid genome (Mus musculus129 x Mus castaneus).
Abstract
Enhancers control cell identity by regulating tissue-specific gene expression in a position and orientation independent manner. These enhancers are often located distally from the regulated gene in intergenic regions or even within the body of another gene. The position independent nature of enhancer activity makes it difficult to match enhancers with the genes they regulate. Deletion of an enhancer region provides direct evidence for enhancer activity and is the gold standard to reveal an enhancer’s role in endogenous gene transcription. Conventional homologous recombination based deletion methods have been surpassed by recent advances in genome editing technology which enable rapid and precisely located changes to the genomes of numerous model organisms. CRISPR/Cas9 mediated genome editing can be used to manipulate the genome in many cell types and organisms rapidly and cost effectively, due to the ease with which Cas9 can be targeted to the genome by a guide RNA from a bespoke expression plasmid. Homozygous deletion of essential gene regulatory elements might lead to lethality or alter cellular phenotype whereas monoallelic deletion of transcriptional enhancers allows for the study of cis-regulation of gene expression without this confounding issue. Presented here is a protocol for CRISPR/Cas9 mediated deletion in F1 mouse embryonic stem (ES) cells (Mus musculus129 x Mus castaneus). Monoallelic deletion, screening and expression analysis is facilitated by single nucleotide polymorphisms (SNP) between the two alleles which occur on average every 125 bp in these cells.
Introduction
Elementi regolatori trascrizionali sono fondamentali per la messa a punto spazio-temporale dell'espressione genica durante lo sviluppo 1 e la modifica di questi elementi può portare a malattia a causa di espressione genica aberranti 2. Molte regioni associate alla malattia identificati da studi di associazione genome wide sono nelle regioni non codificanti e hanno caratteristiche di esaltatori trascrizionale 3-4. Identificare esaltatori e abbinandoli con i geni che regolano è complicato in quanto sono spesso trovano diversi kilobases lontano dai geni che regolano e possono essere attivati in maniera tessuto-specifica 5-6. Previsioni Enhancer sono comunemente basate su segni modificazione degli istoni, complessi mediatore-cohesin e vincolante di trascrizione specifici per tipo di cellula fattori 7-10. Convalida di esaltatori previsti è più spesso fatto attraverso un saggio basato vettore in cui l'enhancer attiva espressione di un gene reporter 11-12. Questi dati forniscono vinformazioni aluable circa il potenziale normativo di sequenze enhancer putativi, ma non rivelano la loro funzione nel loro contesto genomico endogena o identificare i geni che regolano. la modifica del genoma serve come un potente strumento per studiare la funzione di elementi regolatori trascrizionali nel loro contesto endogena di analisi perdita-di-funzione.
I recenti progressi nella modifica del genoma, cioè il / Cas9 sistema di editing genoma CRISPR, facilitano la ricerca della funzione del genoma. Il sistema / Cas9 CRISPR è facile da usare e adattabile per molti sistemi biologici. La proteina Cas9 si rivolge ad un sito specifico nel genoma da un RNA guida (gRNA) 13. Il complesso SpCas9 / gRNA esegue la scansione del genoma per il suo obiettivo di sequenza genomica che deve essere 5 'ad una sequenza protospacer motivo adiacente (PAM), NGG 14-15. appaiamento delle basi del gRNA al suo obiettivo, a 20 nucleotidi (nt) sequenza complementare al gRNA, attiva SpCas9 attività nucleasi con un conseguente Doublpausa e filamento (DSB) 3 bp a monte della sequenza PAM. Specificità è ottenuto attraverso l'accoppiamento base completa nella regione seme gRNA, il 6-12 nt adiacente al PAM; Al contrario, disallineamenti 5 'del seme di solito sono tollerati 16-17. La DSB introdotta può essere riparato entro la fine non omologa unirsi (NHEJ) riparazione del DNA o di omologia riparazione diretta (HDR) mechanisms.NHEJ riparazione del DNA spesso crea inserzione / delezione (indels) di pochi bp al sito di destinazione che possono disturbare l'open reading frame (ORF) di un gene. Per generare delezioni grandi nel genoma due gRNAs, che fiancheggiano la regione di interesse, può essere utilizzato 18-19. Questo approccio è particolarmente utile per lo studio di esaltatori di trascrizione raggruppati in regioni di controllo locus o super-stimolatori che sono più grandi di esaltatori convenzionali 9,18,20-22.
Eliminazioni monoallelic sono un modello valido per lo studio -Regolamento cis di trascrizione. Il chang osservatae nel livello di trascrizione dopo la cancellazione monoallelic di un potenziatore correlato al ruolo di detta enhancer nella regolazione genica senza gli effetti confondenti che possono verificarsi quando la trascrizione di entrambi gli alleli è influenzata potenzialmente influenzare il fitness cellulare. Valutare ridotta espressione è difficile ma senza la capacità di distinguere la eliminato dal tipo di allele selvatico. Inoltre, genotipizzazione delezioni in ogni allele senza la capacità di distinguere i due alleli è impegnativo, soprattutto per le grandi delezioni di> 10 kb a 1 Mb 23 in cui è difficile per amplificare l'intera regione di tipo selvatico mediante PCR. L'uso di cellule ES F1 generati dal passaggio a Mus musculus 129 con Mus castaneus permette ai due alleli per essere differenziati mediante PCR allele-specifica 18,24. Il genoma ibrido in queste cellule facilita allele specifico di screening cancellazione e analisi di espressione. In media c'è un SNP ogni 125 bp tra questi due genomi, Fornendo flessibilità nella progettazione di primer per l'espressione e la genotipizzazione analisi. La presenza di uno SNP può influenzare la temperatura di innesco di fusione (T m) e destinazione specificità amplificazione real-time PCR quantitativa (qPCR) consentendo la discriminazione dei due alleli 25. Inoltre un disadattamento all'interno all'estremità 3 'del primer influenza notevolmente la capacità della DNA polimerasi di estendere dal primer impedendo amplificazione del bersaglio indesiderato allele 26. Descritto nella seguente protocollo è l'uso di cellule ES F1 per l'allele specifico delezioni enhancer di maggiore di 1 KB e la successiva analisi di espressione utilizzando il / Cas9 sistema di editing genoma CRISPR (Figura 1).
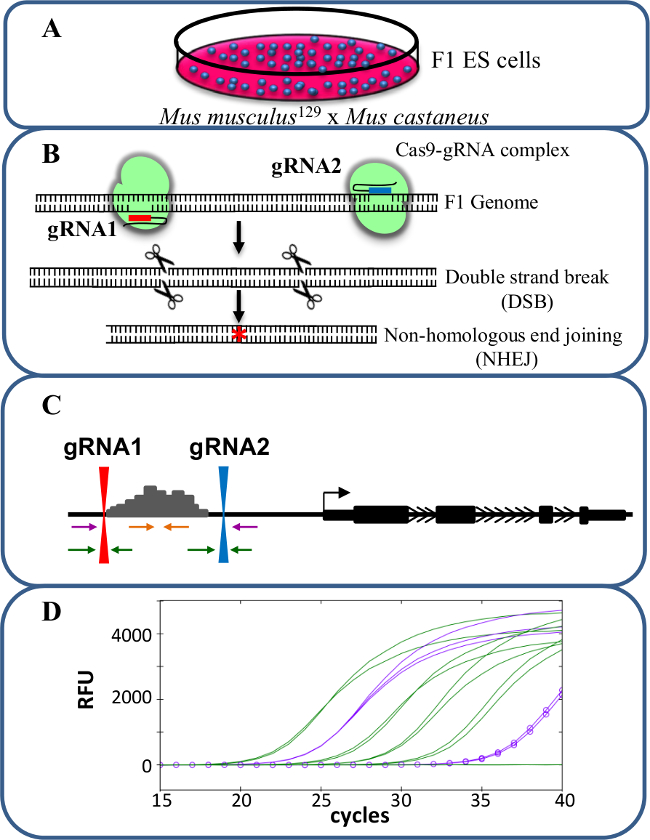
Figura 1. eliminazione Enhancer usando CRISPR / Cas9 per studiare cis -REGlamento di espressione genica. cellule (A) F1 ES generati da un incrocio tra Mus musculus 129 e Mus castaneus sono utilizzati per consentire l'eliminazione allele specifico. (B) Due RNA guida (gRNA) vengono utilizzati per indurre una grande delezione Cas9-mediata della regione enhancer. (C) Set di primer sono utilizzati per identificare grande mono e bi-delezioni alleliche. I primer di colore arancione sono i primer all'interno, i primer viola sono i primer esterni ei primer verde sono i primer fiancheggianti gRNA. (D) Le variazioni di espressione genica sono monitorati utilizzando allele-specifica qPCR. RFU denota unità di fluorescenza relativa. Clicca qui per vedere una versione più grande di questa figura.
Protocol
Representative Results
Discussion
tecnologia di editing genoma mediata CRISPR / Cas9 fornisce un metodo semplice, veloce e poco costoso per la modifica del genoma. Il metodo descritto qui per generare e analizzare l'eliminazione enhancer monoallelic per la caratterizzazione funzionale potenziatore sfrutta SNPs nelle cellule di topo F1. I vantaggi di questo tipo di approccio sono: 1) le eliminazioni enhancer monoallelic non producono effetti confondenti che si verificano quando un esaltatore di critica viene eliminato da entrambi gli alleli, cio?…
Disclosures
The authors have nothing to disclose.
Acknowledgements
We would like to thank all the members of the Mitchell lab for helpful discussions. This work was supported by the Canadian Institutes of Health Research, the Canada Foundation for Innovation and the Ontario Ministry of Research and Innovation (operating and infrastructure grants held by JAM).
Materials
| Phusion High-Fidelity DNA Polymerase | NEB | M0530S | high fidelity DNA polymerase used in gRNA assembly |
| Gibson Assembly Master Mix | NEB | E2611L | |
| gRNA_Cloning Vector | Addgene | 41824 | A target sequence is cloned into this vector to create the gRNA plasmid |
| pCas9_GFP | Addgene | 44719 | Codon-optimized SpCas9 and EGFP co-expression plasmid |
| AflII | NEB | R0520S | |
| EcoRI | NEB | R3101S | |
| Neon Transfection System 100 µL Kit | Life Technologies | MPK10096 | Microporator transfection technology |
| prepGEM | ZyGEM | PT10500 | genomic DNA extraction reagent |
| Nucleo Spin Gel & PCR Clean-up | Macherey-Nagel | 740609.5 | |
| High-Speed Plasmid Mini Kit | Geneaid | PD300 | |
| Maxi Plasmid Kit Endotoxin Free | Geneaid | PME25 | |
| SYBR select mix for CFX | Life Technologies | 4472942 | qPCR reagent |
| iScript cDNA synthesis kit | Bio-rad | 170-8891 | Reverse transcription reagent |
| 0.25% Trypsin with EDTA | Life Technologies | 25200072 | |
| PBS without Ca/Mg2+ | Sigma | D8537 | |
| 0.5M EDTA | Bioshop | EDT111.500 | |
| HBSS | Life Technologies | 14175095 | |
| 1M HEPES | Life Technologies | 13630080 | |
| BSA fraction V (7.5%) | Life Technologies | 15260037 | |
| Max Efficiency DH5α competent cells | Invitrogen | 18258012 | |
| FBS | ES cell qualified | FBS is subjected to a prior testing in mouse ES cells for pluripotency | |
| DMSO | Sigma | D2650 | |
| Glutamax | Invitrogen | 35050 | |
| DMEM | Life Technologies | 11960069 | |
| Pencillin/Streptomycin | Invitrogen | 15140 | |
| Sodium pyruvate | Invitrogen | 11360 | |
| Non-essential aminoacid | Invitrogen | 11140 | |
| β-mercaptoethanol | Sigma | M7522 | |
| 96-well plate | Sarstedt | 83.3924 | |
| Sealing tape | Sarstedt | 95.1994 | |
| CoolCell LX | Biocision | BCS-405 | alcohol-free cell freezing container |
| CHIR99021 | Biovision | 1748-5 | Inhibitor for F1 ES cell culture |
| PD0325901 | Invivogen | inh-pd32 | Inhibitor for F1 ES cell culture |
| LIF | Chemicon | ESG1107 | Inhibitor for F1 ES cell culture |
References
- Sagai, T., Hosoya, M., Mizushina, Y., Tamura, M., Shiroishi, T. Elimination of a long-range cis-regulatory module causes complete loss of limb-specific Shh expression and truncation of the mouse limb. Development. 132 (4), 797-803 (2005).
- Kleinjan, D. A., Lettice, L. A. Long-range gene control and genetic disease. Adv Genet. 61, 339-388 (2008).
- Visel, A., Rubin, E. M., Pennacchio, L. A. Genomic views of distant-acting enhancers. Nature. 461 (7261), 199-205 (2009).
- Maurano, M. T., et al. Systematic localization of common disease-associated variation in regulatory DNA. Science. 337 (6099), 1190-1195 (2012).
- Heintzman, N. D., et al. Histone modifications at human enhancers reflect global cell-type-specific gene expression. Nature. 459 (7243), 108-112 (2009).
- Shen, Y., et al. A map of the cis-regulatory sequences in the mouse genome. Nature. 488 (7409), 116-120 (2012).
- Johnson, D. S., Mortazavi, A., Myers, R. M., Wold, B. Genome-wide mapping of in vivo protein-DNA interactions. Science. 316 (5830), 1497-1502 (2007).
- Rhee, H. S., Pugh, B. F. Comprehensive genome-wide protein-DNA interactions detected at single-nucleotide resolution. Cell. 147 (6), 1408-1419 (2011).
- Whyte, W. A., et al. Master transcription factors and mediator establish super-enhancers at key cell identity genes. Cell. 153 (2), 307-319 (2013).
- Chen, C. Y., Morris, Q., Mitchell, J. A. Enhancer identification in mouse embryonic stem cells using integrative modeling of chromatin and genomic features. BMC Genomics. 13 (1), 152 (2012).
- Patwardhan, R. P., et al. Massively parallel functional dissection of mammalian enhancers in vivo. Nat Biotechnol. 30 (3), 265-270 (2012).
- Melnikov, A., et al. Systematic dissection and optimization of inducible enhancers in human cells using a massively parallel reporter assay. Nat Biotechnol. 30 (3), 271-277 (2012).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339 (6121), 819-823 (2013).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat Biotechnol. 31 (9), 827-832 (2013).
- Cho, S. W., et al. Analysis of off-target effects of CRISPR/Cas-derived RNA-guided endonucleases and nickases. Genome Res. 24 (1), 132-141 (2014).
- Zhou, H. Y., et al. A Sox2 distal enhancer cluster regulates embryonic stem cell differentiation potential. Genes Dev. 28 (24), 2699-2711 (2014).
- Fujii, W., Kawasaki, K., Sugiura, K., Naito, K. Efficient generation of large-scale genome-modified mice using gRNA and CAS9 endonuclease. Nucleic Acids Res. 41 (20), e187 (2013).
- Tuan, D. Y., Solomon, W. B., London, I. M., Lee, D. P. An erythroid-specific, developmental-stage-independent enhancer far upstream of the human ‘beta-like globin’ genes. Proc Natl Acad Sci U S A. 86 (8), 2554-2558 (1989).
- Amano, T., et al. Chromosomal dynamics at the Shh locus: limb bud-specific differential regulation of competence and active transcription. Dev Cell. 16 (1), 47-57 (2009).
- Li, Y., et al. CRISPR reveals a distal super-enhancer required for Sox2 expression in mouse embryonic stem cells. PLoS One. 9 (12), e114485 (2014).
- Canver, M. C., et al. Characterization of genomic deletion efficiency mediated by clustered regularly interspaced palindromic repeats (CRISPR)/Cas9 nuclease system in mammalian cells. J Biol Chem. 289 (31), 21312-21324 (2014).
- Mlynarczyk-Evans, S., et al. X chromosomes alternate between two states prior to random X-inactivation. PLoS Biol. 4 (6), e159 (2006).
- Lefever, S., Pattyn, F., Hellemans, J., Vandesompele, J. Single-nucleotide polymorphisms and other mismatches reduce performance of quantitative PCR assays. Clin Chem. 59 (10), 1470-1480 (2013).
- Huang, M. M., Arnheim, N., Goodman, M. F. Extension of base mispairs by Taq DNA polymerase: implications for single nucleotide discrimination in PCR. Nucleic Acids Res. 20 (17), 4567-4573 (1992).
- Keane, T. M., et al. Mouse genomic variation and its effect on phenotypes and gene regulation. Nature. 477 (7364), 289-294 (2011).
- Yalcin, B., et al. Sequence-based characterization of structural variation in the mouse genome. Nature. 477 (7364), 326-329 (2011).
- Gibson, D. G., et al. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nat Methods. 6 (5), 343-345 (2009).
- Gibson, D. G., Smith, H. O., Hutchison, C. A., Venter, J. C., Merryman, C. Chemical synthesis of the mouse mitochondrial genome. Nat Methods. 7 (11), 901-903 (2010).
- Ding, Q., et al. Enhanced efficiency of human pluripotent stem cell genome editing through replacing TALENs with CRISPRs. Cell Stem Cell. 12 (4), 393-394 (2013).
- Basu, S., Campbell, H. M., Dittel, B. N., Ray, A. Purification of specific cell population by fluorescence activated cell sorting (FACS). J Vis Exp. (41), (2010).
- Forlenza, M., Kaiser, T., Savelkoul, H. F., Wiegertjes, G. F. The use of real-time quantitative PCR for the analysis of cytokine mRNA levels. Methods Mol Biol. 820, 7-23 (2012).
- Wu, J. H., Hong, P. Y., Liu, W. T. Quantitative effects of position and type of single mismatch on single base primer extension. J Microbiol Methods. 77 (3), 267-275 (2009).
- Sanyal, A., Lajoie, B. R., Jain, G., Dekker, J. The long-range interaction landscape of gene promoters. Nature. 489 (7414), 109-113 (2012).

