La generación de CRISPR / Cas9 mediada monoallelic deleciones para estudiar la función del reforzador en las células madre embrionarias de ratón
Summary
Experimental validation of enhancer activity is best approached by loss-of-function analysis. Presented here is an efficient protocol that uses CRISPR/Cas9 mediated deletion to study allele-specific regulation of gene transcription in F1 ES cells which contain a hybrid genome (Mus musculus129 x Mus castaneus).
Abstract
Enhancers control cell identity by regulating tissue-specific gene expression in a position and orientation independent manner. These enhancers are often located distally from the regulated gene in intergenic regions or even within the body of another gene. The position independent nature of enhancer activity makes it difficult to match enhancers with the genes they regulate. Deletion of an enhancer region provides direct evidence for enhancer activity and is the gold standard to reveal an enhancer’s role in endogenous gene transcription. Conventional homologous recombination based deletion methods have been surpassed by recent advances in genome editing technology which enable rapid and precisely located changes to the genomes of numerous model organisms. CRISPR/Cas9 mediated genome editing can be used to manipulate the genome in many cell types and organisms rapidly and cost effectively, due to the ease with which Cas9 can be targeted to the genome by a guide RNA from a bespoke expression plasmid. Homozygous deletion of essential gene regulatory elements might lead to lethality or alter cellular phenotype whereas monoallelic deletion of transcriptional enhancers allows for the study of cis-regulation of gene expression without this confounding issue. Presented here is a protocol for CRISPR/Cas9 mediated deletion in F1 mouse embryonic stem (ES) cells (Mus musculus129 x Mus castaneus). Monoallelic deletion, screening and expression analysis is facilitated by single nucleotide polymorphisms (SNP) between the two alleles which occur on average every 125 bp in these cells.
Introduction
Elementos reguladores de la transcripción son críticos para la puesta a punto espacio-temporal de la expresión génica durante el desarrollo 1 y la modificación de estos elementos puede dar lugar a la enfermedad debido a la expresión aberrante de genes 2. Muchas regiones asociadas a la enfermedad identificados por el genoma estudios de asociación de todo se encuentran en las regiones no codificantes y tienen características de potenciadores de la transcripción 3-4. La identificación de los potenciadores y emparejarlos con los genes que regulan se complica ya que a menudo se encuentran varias kilobases lejos de los genes que regulan y pueden ser activados de una manera específica de tejido 5-6. Predicciones Enhancer se basan comúnmente en marcas de modificación de las histonas, complejos de cohesinas-mediador y la unión de células de transcripción de tipo específico factores de 7-10. Validación de potenciadores predichos a menudo se realiza a través de un ensayo basado en vector en el que el potenciador activa la expresión de un gen indicador 11-12. Estos datos proporcionan valuable información sobre el potencial regulador de las secuencias potenciadoras putativos, pero no revelan su función en su contexto genómico endógeno o identificar los genes que regulan. edición genoma sirve como una poderosa herramienta para estudiar la función de los elementos reguladores de la transcripción en su contexto endógena mediante análisis de la pérdida de función.
Los avances recientes en la edición del genoma, es decir, el sistema de edición de genoma / Cas9 CRISPR, facilitar la investigación de la función del genoma. El sistema / Cas9 CRISPR es fácil de usar y adaptable para muchos sistemas biológicos. La proteína Cas9 está dirigida a un sitio específico en el genoma por un ARN de guía (gRNA) 13. El complejo SpCas9 / gRNA escanea el genoma de su secuencia genómica diana que debe ser de 5 'a una secuencia protospacer motivo adyacente (PAM), NGG 14-15. el apareamiento de bases de la gRNA a su diana, un (nt) secuencia de 20 nucleótidos complementaria a la gRNA, activa la actividad nucleasa SpCas9 que resulta en una doubldescanso e hebra (DSB) 3 pb aguas arriba de la secuencia de PAM. La especificidad se logra a través de apareamiento de bases completa en la región de la semilla gRNA, el 6-12 nt adyacente a la PAM; por el contrario, no corresponde a 5 'de la semilla son generalmente tolerada 16-17. El OSD introducido se puede reparar, ya sea por el extremo no homóloga (NHEJ) de reparación del ADN o reparación dirigida homología de reparación del ADN (HDR) mechanisms.NHEJ menudo crea inserción / deleción (indeles) de unos pocos pares de bases en el sitio diana que puede interrumpir el marco de lectura abierto (ORF) de un gen. Para generar deleciones de mayor tamaño en el genoma de dos gRNAs, que flanquean la región de interés, se puede utilizar 18-19. Este enfoque es particularmente útil para el estudio de los potenciadores de la transcripción agrupadas en regiones de control de locus o super-potenciadores que son más grandes que los potenciadores convencionales 9,18,20-22.
Supresiones monoallelic son un modelo valioso para el estudio de -Reglamento cis de la transcripción. El chang observadoe en el nivel de transcripción después de la eliminación monoallelic de un potenciador se correlaciona con el papel de promotor de que en la regulación de genes, sin los efectos de confusión que pueden ocurrir cuando la transcripción de ambos alelos se ve afectada potencialmente influir en la aptitud celular. Evaluación de la reducción de expresión es difícil, sin embargo, sin la capacidad de distinguir el borrado de el alelo de tipo salvaje. Además, la genotipificación de deleciones en cada alelo sin la capacidad de distinguir los dos alelos es un reto, especialmente para las grandes deleciones de> 10 kb a 1 Mb 23 en el que es difícil para amplificar toda la región de tipo salvaje por PCR. El uso de células ES F1 generados por musculus cruce Mus 129 con Mus castaneus permite que los dos alelos que se diferencian por alelo-específico PCR 18,24. El genoma híbrido en estas células facilita alelo cribado deleción específica y análisis de la expresión. En promedio hay un SNP cada 125 pb entre estos dos genomas, Proporcionando flexibilidad en el diseño de cebadores para la expresión y genotipado analiza. La presencia de un SNP puede influir en la temperatura de fusión del cebador (T m) y apuntar a la especificidad en la amplificación cuantitativa en tiempo real PCR (qPCR) lo que permite la discriminación de los dos alelos 25. Además, una falta de coincidencia dentro de el extremo 3 'del cebador influye en gran medida la capacidad de la ADN polimerasa para extender el cebador de la prevención de la amplificación de la diana alelo no deseado 26. Describe en el siguiente protocolo es el uso de células madre embrionarias para la F1 deleciones específicas de alelo potenciador de más de 1 kb y el análisis posterior expresión que utilizan el sistema de CRISPR / Cas9 edición genoma (Figura 1).
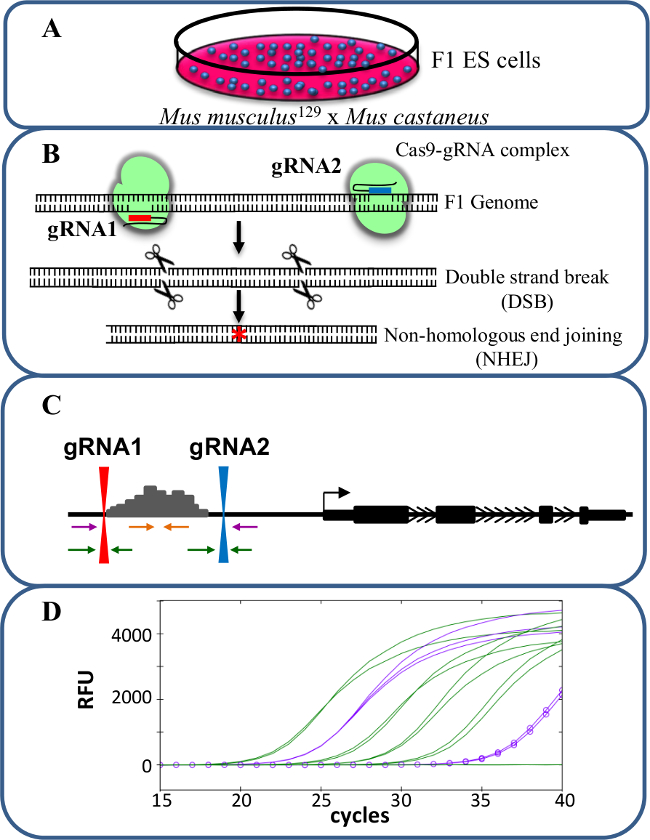
Figura 1. eliminación reforzador usando CRISPR / Cas9 para estudiar cis -regulación de la expresión génica. células F1 ES (A) generados por un cruce entre Mus musculus 129 y castaneus Mus se utilizan para permitir la supresión alelo específico. (B) dos ARN de guía (gRNA) se utilizan para inducir una gran deleción mediada por Cas9 de la región potenciadora. (C) Los conjuntos de cebadores se utilizan para identificar gran mono- y bi-deleciones alélicas. Los cebadores de color naranja son los iniciadores en el interior, los cebadores de color púrpura son los cebadores externos y los cebadores verdes son los cebadores que flanquean gRNA. (D) Los cambios en la expresión génica son monitoreados mediante qPCR específica de alelo. RFU denota unidades relativas de fluorescencia. Por favor, haga clic aquí para ver una versión más grande de esta figura.
Protocol
Representative Results
Discussion
CRISPR / Cas9 tecnología de edición genoma mediada proporciona un método sencillo, rápido y barato para la modificación del genoma. El método detallado aquí para generar y analizar eliminación potenciador monoallelic para la caracterización funcional potenciador se aprovecha de SNPs en células de ratón F1. Las ventajas de este tipo de enfoque son: 1) deleciones potenciador monoallelic no producen efectos de confusión que se producen cuando un promotor de crítico se elimina de ambos alelos, es decir,</em…
Disclosures
The authors have nothing to disclose.
Acknowledgements
We would like to thank all the members of the Mitchell lab for helpful discussions. This work was supported by the Canadian Institutes of Health Research, the Canada Foundation for Innovation and the Ontario Ministry of Research and Innovation (operating and infrastructure grants held by JAM).
Materials
| Phusion High-Fidelity DNA Polymerase | NEB | M0530S | high fidelity DNA polymerase used in gRNA assembly |
| Gibson Assembly Master Mix | NEB | E2611L | |
| gRNA_Cloning Vector | Addgene | 41824 | A target sequence is cloned into this vector to create the gRNA plasmid |
| pCas9_GFP | Addgene | 44719 | Codon-optimized SpCas9 and EGFP co-expression plasmid |
| AflII | NEB | R0520S | |
| EcoRI | NEB | R3101S | |
| Neon Transfection System 100 µL Kit | Life Technologies | MPK10096 | Microporator transfection technology |
| prepGEM | ZyGEM | PT10500 | genomic DNA extraction reagent |
| Nucleo Spin Gel & PCR Clean-up | Macherey-Nagel | 740609.5 | |
| High-Speed Plasmid Mini Kit | Geneaid | PD300 | |
| Maxi Plasmid Kit Endotoxin Free | Geneaid | PME25 | |
| SYBR select mix for CFX | Life Technologies | 4472942 | qPCR reagent |
| iScript cDNA synthesis kit | Bio-rad | 170-8891 | Reverse transcription reagent |
| 0.25% Trypsin with EDTA | Life Technologies | 25200072 | |
| PBS without Ca/Mg2+ | Sigma | D8537 | |
| 0.5M EDTA | Bioshop | EDT111.500 | |
| HBSS | Life Technologies | 14175095 | |
| 1M HEPES | Life Technologies | 13630080 | |
| BSA fraction V (7.5%) | Life Technologies | 15260037 | |
| Max Efficiency DH5α competent cells | Invitrogen | 18258012 | |
| FBS | ES cell qualified | FBS is subjected to a prior testing in mouse ES cells for pluripotency | |
| DMSO | Sigma | D2650 | |
| Glutamax | Invitrogen | 35050 | |
| DMEM | Life Technologies | 11960069 | |
| Pencillin/Streptomycin | Invitrogen | 15140 | |
| Sodium pyruvate | Invitrogen | 11360 | |
| Non-essential aminoacid | Invitrogen | 11140 | |
| β-mercaptoethanol | Sigma | M7522 | |
| 96-well plate | Sarstedt | 83.3924 | |
| Sealing tape | Sarstedt | 95.1994 | |
| CoolCell LX | Biocision | BCS-405 | alcohol-free cell freezing container |
| CHIR99021 | Biovision | 1748-5 | Inhibitor for F1 ES cell culture |
| PD0325901 | Invivogen | inh-pd32 | Inhibitor for F1 ES cell culture |
| LIF | Chemicon | ESG1107 | Inhibitor for F1 ES cell culture |
References
- Sagai, T., Hosoya, M., Mizushina, Y., Tamura, M., Shiroishi, T. Elimination of a long-range cis-regulatory module causes complete loss of limb-specific Shh expression and truncation of the mouse limb. Development. 132 (4), 797-803 (2005).
- Kleinjan, D. A., Lettice, L. A. Long-range gene control and genetic disease. Adv Genet. 61, 339-388 (2008).
- Visel, A., Rubin, E. M., Pennacchio, L. A. Genomic views of distant-acting enhancers. Nature. 461 (7261), 199-205 (2009).
- Maurano, M. T., et al. Systematic localization of common disease-associated variation in regulatory DNA. Science. 337 (6099), 1190-1195 (2012).
- Heintzman, N. D., et al. Histone modifications at human enhancers reflect global cell-type-specific gene expression. Nature. 459 (7243), 108-112 (2009).
- Shen, Y., et al. A map of the cis-regulatory sequences in the mouse genome. Nature. 488 (7409), 116-120 (2012).
- Johnson, D. S., Mortazavi, A., Myers, R. M., Wold, B. Genome-wide mapping of in vivo protein-DNA interactions. Science. 316 (5830), 1497-1502 (2007).
- Rhee, H. S., Pugh, B. F. Comprehensive genome-wide protein-DNA interactions detected at single-nucleotide resolution. Cell. 147 (6), 1408-1419 (2011).
- Whyte, W. A., et al. Master transcription factors and mediator establish super-enhancers at key cell identity genes. Cell. 153 (2), 307-319 (2013).
- Chen, C. Y., Morris, Q., Mitchell, J. A. Enhancer identification in mouse embryonic stem cells using integrative modeling of chromatin and genomic features. BMC Genomics. 13 (1), 152 (2012).
- Patwardhan, R. P., et al. Massively parallel functional dissection of mammalian enhancers in vivo. Nat Biotechnol. 30 (3), 265-270 (2012).
- Melnikov, A., et al. Systematic dissection and optimization of inducible enhancers in human cells using a massively parallel reporter assay. Nat Biotechnol. 30 (3), 271-277 (2012).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339 (6121), 819-823 (2013).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat Biotechnol. 31 (9), 827-832 (2013).
- Cho, S. W., et al. Analysis of off-target effects of CRISPR/Cas-derived RNA-guided endonucleases and nickases. Genome Res. 24 (1), 132-141 (2014).
- Zhou, H. Y., et al. A Sox2 distal enhancer cluster regulates embryonic stem cell differentiation potential. Genes Dev. 28 (24), 2699-2711 (2014).
- Fujii, W., Kawasaki, K., Sugiura, K., Naito, K. Efficient generation of large-scale genome-modified mice using gRNA and CAS9 endonuclease. Nucleic Acids Res. 41 (20), e187 (2013).
- Tuan, D. Y., Solomon, W. B., London, I. M., Lee, D. P. An erythroid-specific, developmental-stage-independent enhancer far upstream of the human ‘beta-like globin’ genes. Proc Natl Acad Sci U S A. 86 (8), 2554-2558 (1989).
- Amano, T., et al. Chromosomal dynamics at the Shh locus: limb bud-specific differential regulation of competence and active transcription. Dev Cell. 16 (1), 47-57 (2009).
- Li, Y., et al. CRISPR reveals a distal super-enhancer required for Sox2 expression in mouse embryonic stem cells. PLoS One. 9 (12), e114485 (2014).
- Canver, M. C., et al. Characterization of genomic deletion efficiency mediated by clustered regularly interspaced palindromic repeats (CRISPR)/Cas9 nuclease system in mammalian cells. J Biol Chem. 289 (31), 21312-21324 (2014).
- Mlynarczyk-Evans, S., et al. X chromosomes alternate between two states prior to random X-inactivation. PLoS Biol. 4 (6), e159 (2006).
- Lefever, S., Pattyn, F., Hellemans, J., Vandesompele, J. Single-nucleotide polymorphisms and other mismatches reduce performance of quantitative PCR assays. Clin Chem. 59 (10), 1470-1480 (2013).
- Huang, M. M., Arnheim, N., Goodman, M. F. Extension of base mispairs by Taq DNA polymerase: implications for single nucleotide discrimination in PCR. Nucleic Acids Res. 20 (17), 4567-4573 (1992).
- Keane, T. M., et al. Mouse genomic variation and its effect on phenotypes and gene regulation. Nature. 477 (7364), 289-294 (2011).
- Yalcin, B., et al. Sequence-based characterization of structural variation in the mouse genome. Nature. 477 (7364), 326-329 (2011).
- Gibson, D. G., et al. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nat Methods. 6 (5), 343-345 (2009).
- Gibson, D. G., Smith, H. O., Hutchison, C. A., Venter, J. C., Merryman, C. Chemical synthesis of the mouse mitochondrial genome. Nat Methods. 7 (11), 901-903 (2010).
- Ding, Q., et al. Enhanced efficiency of human pluripotent stem cell genome editing through replacing TALENs with CRISPRs. Cell Stem Cell. 12 (4), 393-394 (2013).
- Basu, S., Campbell, H. M., Dittel, B. N., Ray, A. Purification of specific cell population by fluorescence activated cell sorting (FACS). J Vis Exp. (41), (2010).
- Forlenza, M., Kaiser, T., Savelkoul, H. F., Wiegertjes, G. F. The use of real-time quantitative PCR for the analysis of cytokine mRNA levels. Methods Mol Biol. 820, 7-23 (2012).
- Wu, J. H., Hong, P. Y., Liu, W. T. Quantitative effects of position and type of single mismatch on single base primer extension. J Microbiol Methods. 77 (3), 267-275 (2009).
- Sanyal, A., Lajoie, B. R., Jain, G., Dekker, J. The long-range interaction landscape of gene promoters. Nature. 489 (7414), 109-113 (2012).

