CRISPR Génération / cas9 Mediated monoallélique Suppressions pour étudier la fonction Enhancer dans souris cellules souches embryonnaires
Summary
Experimental validation of enhancer activity is best approached by loss-of-function analysis. Presented here is an efficient protocol that uses CRISPR/Cas9 mediated deletion to study allele-specific regulation of gene transcription in F1 ES cells which contain a hybrid genome (Mus musculus129 x Mus castaneus).
Abstract
Enhancers control cell identity by regulating tissue-specific gene expression in a position and orientation independent manner. These enhancers are often located distally from the regulated gene in intergenic regions or even within the body of another gene. The position independent nature of enhancer activity makes it difficult to match enhancers with the genes they regulate. Deletion of an enhancer region provides direct evidence for enhancer activity and is the gold standard to reveal an enhancer’s role in endogenous gene transcription. Conventional homologous recombination based deletion methods have been surpassed by recent advances in genome editing technology which enable rapid and precisely located changes to the genomes of numerous model organisms. CRISPR/Cas9 mediated genome editing can be used to manipulate the genome in many cell types and organisms rapidly and cost effectively, due to the ease with which Cas9 can be targeted to the genome by a guide RNA from a bespoke expression plasmid. Homozygous deletion of essential gene regulatory elements might lead to lethality or alter cellular phenotype whereas monoallelic deletion of transcriptional enhancers allows for the study of cis-regulation of gene expression without this confounding issue. Presented here is a protocol for CRISPR/Cas9 mediated deletion in F1 mouse embryonic stem (ES) cells (Mus musculus129 x Mus castaneus). Monoallelic deletion, screening and expression analysis is facilitated by single nucleotide polymorphisms (SNP) between the two alleles which occur on average every 125 bp in these cells.
Introduction
Des éléments de régulation transcriptionnelle sont critiques pour réglage fin spatio-temporelle de l' expression des gènes au cours du développement 1 et la modification de ces éléments peut entraîner une maladie due à l' expression génique aberrante 2. De nombreuses régions associées à des maladies identifiées par le génome des études d'association sont larges dans les régions non codantes et présentent des caractéristiques d'amplificateurs de transcription 3-4. L' identification des amplificateurs et les harmoniser avec les gènes qu'ils régulent est compliqué car ils sont souvent situés à plusieurs kilobases à l' écart des gènes qu'ils régulent et peuvent être activées d'une manière spécifique au tissu 5-6. Prévisions Enhancer sont généralement basées sur des marques de modification des histones, des complexes de médiateur-cohesin et la liaison de la cellule transcription spécifique de type facteurs 7-10. La validation des amplificateurs de prédits est le plus souvent réalisé par un dosage à base de vecteur dans lequel l'activateur active l' expression d'un gène rapporteur 11-12. Ces données fournissent vinformations ressources précieuses sur le potentiel de régulation des séquences amplificatrices putatives, mais ne révèlent pas leur fonction dans leur contexte génomique endogène ou d'identifier les gènes qu'ils régulent. édition du génome est un outil puissant pour étudier la fonction des éléments de régulation transcriptionnelle dans leur contexte endogène par l'analyse de perte de fonction.
Les progrès récents dans l'édition du génome, à savoir le / cas9 système de montage du génome CRISPR, faciliter l'enquête de la fonction du génome. Le système / cas9 CRISPR est facile à utiliser et adaptable à de nombreux systèmes biologiques. La protéine cas9 est ciblé à un site spécifique par une ARN de guidage (ARNg) 13 dans le génome. Le complexe SpCas9 / gARN analyse du génome pour sa séquence génomique cible qui doit être 5 'à une séquence protospacer motif adjacent (PAM), NGG 14-15. Appariement de bases de l'ARNg à sa cible, un 20 nucleotides (nt) la séquence complémentaire de l'ARNg, active une activité nuclease SpCas9 résultant en un doublpause e brin (DSB) de 3 pb en amont de la séquence PAM. La spécificité est obtenue par appariement complet de la base dans la région de la graine gARN, le 12.06 nt adjacent à l'APM; à l' inverse, mal assortit 5 'de la graine sont généralement tolérés 16-17. L'ORD a introduit peut être réparé soit par l'extrémité non homologue de jonction (NHEJ) réparation de l'ADN ou d'homologie de réparation dirigée (HDR) mechanisms.NHEJ réparation de l'ADN crée souvent insertion / délétion (indels) de quelques pb au niveau du site cible qui peut perturber le cadre ouvert de lecture (ORF) d'un gène. Pour générer de plus grandes deletions dans le génome de deux ARNg, qui flanquent la région d'intérêt, peuvent être utilisés 18 à 19. Cette approche est particulièrement utile pour l'étude des amplificateurs de transcription regroupés en régions de contrôle de locus ou super-amplificateurs qui sont plus grands que des amplificateurs conventionnels 9,18,20-22.
Suppressions monoallélique sont un modèle précieux pour l' étude cis – régulation de la transcription. Le chang observée au niveau de la transcription après la suppression monoallélique d'un activateur est en corrélation avec le rôle de cet activateur dans la régulation des gènes sans les effets de confusion qui peuvent se produire lorsque la transcription des deux allèles est affecté potentiellement influencer remise en forme cellulaire. L'évaluation de l'expression réduite est difficile sans toutefois la capacité de distinguer la suppression de l'allèle de type sauvage. En outre, le génotypage délétions à chaque allèle sans la capacité de distinguer les deux allèles est difficile, surtout pour les grandes suppressions de> 10 kb à 1 Mb 23 , dans lequel il est difficile d'amplifier toute la région de type sauvage par PCR. L'utilisation de cellules ES F1 générées par croisement avec 129 Mus musculus Mus castaneus permet aux deux allèles d'être différenciés par PCR spécifique d'allèle 18,24. Le génome hybride dans ces cellules facilite le dépistage de la suppression spécifique de l'allèle et l'analyse d'expression. En moyenne, il y a un SNP tous les 125 pb entre ces deux génomes, Offrant une flexibilité dans la conception de l'amorce d'expression et de génotypage analyse. La présence d'un SNP peut influer sur la température d' amorce de fusion (Tm) et la spécificité de cible en temps réel par PCR quantitative (qPCR) d' amplification permettant une discrimination des deux allèles 25. En outre , un mésappariement à l'intérieur de l' extrémité 3 'de l'amorce influence grandement la capacité de l' ADN polymérase pour s'étendre à partir de l'amorce empêchant l' amplification de l'allèle cible indésirable 26. Décrite dans le protocole suivant est l'utilisation de cellules ES F1 pour allèle spécifique enhancer suppressions de plus de 1 kb et analyse de l' expression suivante en utilisant le / cas9 système de montage du génome CRISPR (figure 1).
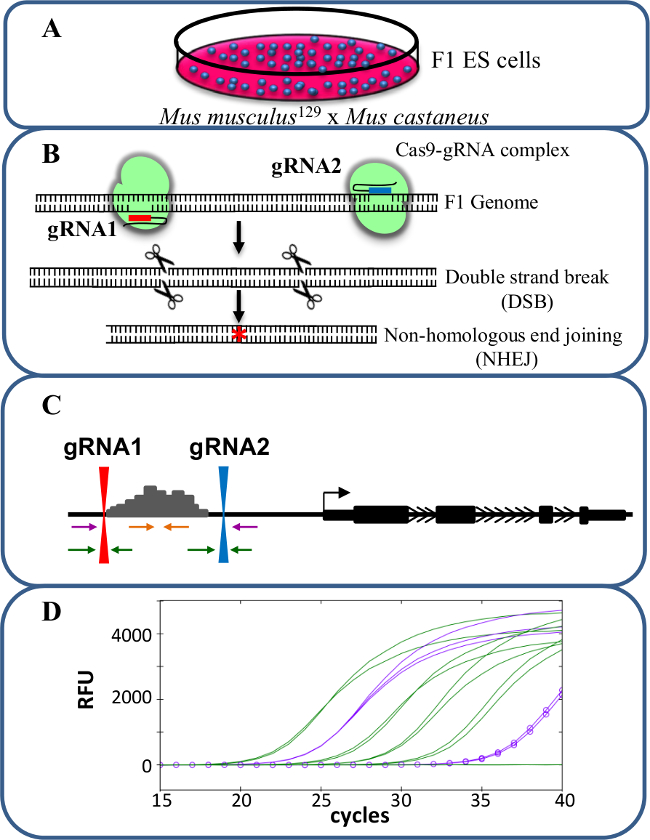
Figure 1. suppression Enhancer utilisant CRISPR / cas9 pour étudier cis -regulation de l' expression génique. (A) Les cellules ES F1 générées par un croisement entre Mus musculus 129 et Mus castaneus sont utilisés pour permettre la suppression allèle spécifique. (B) Deux ARN de guidage (ARNg) sont utilisés pour induire une grande deletion à médiation par cas9 de la région amplificatrice. (C) Les amorces sont utilisées pour identifier les grandes mono- et délétions bi-alléliques. Les amorces orange sont les amorces à l'intérieur, les amorces violet sont les amorces externes et les amorces vertes sont les amorces flanquant gARN. (D) Les changements dans l' expression des gènes sont surveillés à l' aide spécifique d'allèle qPCR. RFU désigne des unités de fluorescence relative. S'il vous plaît cliquez ici pour voir une version plus grande de cette figure.
Protocol
Representative Results
Discussion
CRISPR / cas9 technologie d'édition du génome médiation fournit une méthode simple, rapide et peu coûteuse pour la modification du génome. La méthode détaillée ici pour générer et analyser monoallélique suppression d'activateur pour la caractérisation fonctionnelle enhancer tire profit de SNP dans les cellules de souris F1. Les avantages de ce type d'approche sont: 1) les suppressions d'activateurs monoalléliques ne produisent pas des effets de confusion qui se produisent quand un activateu…
Disclosures
The authors have nothing to disclose.
Acknowledgements
We would like to thank all the members of the Mitchell lab for helpful discussions. This work was supported by the Canadian Institutes of Health Research, the Canada Foundation for Innovation and the Ontario Ministry of Research and Innovation (operating and infrastructure grants held by JAM).
Materials
| Phusion High-Fidelity DNA Polymerase | NEB | M0530S | high fidelity DNA polymerase used in gRNA assembly |
| Gibson Assembly Master Mix | NEB | E2611L | |
| gRNA_Cloning Vector | Addgene | 41824 | A target sequence is cloned into this vector to create the gRNA plasmid |
| pCas9_GFP | Addgene | 44719 | Codon-optimized SpCas9 and EGFP co-expression plasmid |
| AflII | NEB | R0520S | |
| EcoRI | NEB | R3101S | |
| Neon Transfection System 100 µL Kit | Life Technologies | MPK10096 | Microporator transfection technology |
| prepGEM | ZyGEM | PT10500 | genomic DNA extraction reagent |
| Nucleo Spin Gel & PCR Clean-up | Macherey-Nagel | 740609.5 | |
| High-Speed Plasmid Mini Kit | Geneaid | PD300 | |
| Maxi Plasmid Kit Endotoxin Free | Geneaid | PME25 | |
| SYBR select mix for CFX | Life Technologies | 4472942 | qPCR reagent |
| iScript cDNA synthesis kit | Bio-rad | 170-8891 | Reverse transcription reagent |
| 0.25% Trypsin with EDTA | Life Technologies | 25200072 | |
| PBS without Ca/Mg2+ | Sigma | D8537 | |
| 0.5M EDTA | Bioshop | EDT111.500 | |
| HBSS | Life Technologies | 14175095 | |
| 1M HEPES | Life Technologies | 13630080 | |
| BSA fraction V (7.5%) | Life Technologies | 15260037 | |
| Max Efficiency DH5α competent cells | Invitrogen | 18258012 | |
| FBS | ES cell qualified | FBS is subjected to a prior testing in mouse ES cells for pluripotency | |
| DMSO | Sigma | D2650 | |
| Glutamax | Invitrogen | 35050 | |
| DMEM | Life Technologies | 11960069 | |
| Pencillin/Streptomycin | Invitrogen | 15140 | |
| Sodium pyruvate | Invitrogen | 11360 | |
| Non-essential aminoacid | Invitrogen | 11140 | |
| β-mercaptoethanol | Sigma | M7522 | |
| 96-well plate | Sarstedt | 83.3924 | |
| Sealing tape | Sarstedt | 95.1994 | |
| CoolCell LX | Biocision | BCS-405 | alcohol-free cell freezing container |
| CHIR99021 | Biovision | 1748-5 | Inhibitor for F1 ES cell culture |
| PD0325901 | Invivogen | inh-pd32 | Inhibitor for F1 ES cell culture |
| LIF | Chemicon | ESG1107 | Inhibitor for F1 ES cell culture |
References
- Sagai, T., Hosoya, M., Mizushina, Y., Tamura, M., Shiroishi, T. Elimination of a long-range cis-regulatory module causes complete loss of limb-specific Shh expression and truncation of the mouse limb. Development. 132 (4), 797-803 (2005).
- Kleinjan, D. A., Lettice, L. A. Long-range gene control and genetic disease. Adv Genet. 61, 339-388 (2008).
- Visel, A., Rubin, E. M., Pennacchio, L. A. Genomic views of distant-acting enhancers. Nature. 461 (7261), 199-205 (2009).
- Maurano, M. T., et al. Systematic localization of common disease-associated variation in regulatory DNA. Science. 337 (6099), 1190-1195 (2012).
- Heintzman, N. D., et al. Histone modifications at human enhancers reflect global cell-type-specific gene expression. Nature. 459 (7243), 108-112 (2009).
- Shen, Y., et al. A map of the cis-regulatory sequences in the mouse genome. Nature. 488 (7409), 116-120 (2012).
- Johnson, D. S., Mortazavi, A., Myers, R. M., Wold, B. Genome-wide mapping of in vivo protein-DNA interactions. Science. 316 (5830), 1497-1502 (2007).
- Rhee, H. S., Pugh, B. F. Comprehensive genome-wide protein-DNA interactions detected at single-nucleotide resolution. Cell. 147 (6), 1408-1419 (2011).
- Whyte, W. A., et al. Master transcription factors and mediator establish super-enhancers at key cell identity genes. Cell. 153 (2), 307-319 (2013).
- Chen, C. Y., Morris, Q., Mitchell, J. A. Enhancer identification in mouse embryonic stem cells using integrative modeling of chromatin and genomic features. BMC Genomics. 13 (1), 152 (2012).
- Patwardhan, R. P., et al. Massively parallel functional dissection of mammalian enhancers in vivo. Nat Biotechnol. 30 (3), 265-270 (2012).
- Melnikov, A., et al. Systematic dissection and optimization of inducible enhancers in human cells using a massively parallel reporter assay. Nat Biotechnol. 30 (3), 271-277 (2012).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339 (6121), 819-823 (2013).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat Biotechnol. 31 (9), 827-832 (2013).
- Cho, S. W., et al. Analysis of off-target effects of CRISPR/Cas-derived RNA-guided endonucleases and nickases. Genome Res. 24 (1), 132-141 (2014).
- Zhou, H. Y., et al. A Sox2 distal enhancer cluster regulates embryonic stem cell differentiation potential. Genes Dev. 28 (24), 2699-2711 (2014).
- Fujii, W., Kawasaki, K., Sugiura, K., Naito, K. Efficient generation of large-scale genome-modified mice using gRNA and CAS9 endonuclease. Nucleic Acids Res. 41 (20), e187 (2013).
- Tuan, D. Y., Solomon, W. B., London, I. M., Lee, D. P. An erythroid-specific, developmental-stage-independent enhancer far upstream of the human ‘beta-like globin’ genes. Proc Natl Acad Sci U S A. 86 (8), 2554-2558 (1989).
- Amano, T., et al. Chromosomal dynamics at the Shh locus: limb bud-specific differential regulation of competence and active transcription. Dev Cell. 16 (1), 47-57 (2009).
- Li, Y., et al. CRISPR reveals a distal super-enhancer required for Sox2 expression in mouse embryonic stem cells. PLoS One. 9 (12), e114485 (2014).
- Canver, M. C., et al. Characterization of genomic deletion efficiency mediated by clustered regularly interspaced palindromic repeats (CRISPR)/Cas9 nuclease system in mammalian cells. J Biol Chem. 289 (31), 21312-21324 (2014).
- Mlynarczyk-Evans, S., et al. X chromosomes alternate between two states prior to random X-inactivation. PLoS Biol. 4 (6), e159 (2006).
- Lefever, S., Pattyn, F., Hellemans, J., Vandesompele, J. Single-nucleotide polymorphisms and other mismatches reduce performance of quantitative PCR assays. Clin Chem. 59 (10), 1470-1480 (2013).
- Huang, M. M., Arnheim, N., Goodman, M. F. Extension of base mispairs by Taq DNA polymerase: implications for single nucleotide discrimination in PCR. Nucleic Acids Res. 20 (17), 4567-4573 (1992).
- Keane, T. M., et al. Mouse genomic variation and its effect on phenotypes and gene regulation. Nature. 477 (7364), 289-294 (2011).
- Yalcin, B., et al. Sequence-based characterization of structural variation in the mouse genome. Nature. 477 (7364), 326-329 (2011).
- Gibson, D. G., et al. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nat Methods. 6 (5), 343-345 (2009).
- Gibson, D. G., Smith, H. O., Hutchison, C. A., Venter, J. C., Merryman, C. Chemical synthesis of the mouse mitochondrial genome. Nat Methods. 7 (11), 901-903 (2010).
- Ding, Q., et al. Enhanced efficiency of human pluripotent stem cell genome editing through replacing TALENs with CRISPRs. Cell Stem Cell. 12 (4), 393-394 (2013).
- Basu, S., Campbell, H. M., Dittel, B. N., Ray, A. Purification of specific cell population by fluorescence activated cell sorting (FACS). J Vis Exp. (41), (2010).
- Forlenza, M., Kaiser, T., Savelkoul, H. F., Wiegertjes, G. F. The use of real-time quantitative PCR for the analysis of cytokine mRNA levels. Methods Mol Biol. 820, 7-23 (2012).
- Wu, J. H., Hong, P. Y., Liu, W. T. Quantitative effects of position and type of single mismatch on single base primer extension. J Microbiol Methods. 77 (3), 267-275 (2009).
- Sanyal, A., Lajoie, B. R., Jain, G., Dekker, J. The long-range interaction landscape of gene promoters. Nature. 489 (7414), 109-113 (2012).

