ヒト単球由来マクロファージにおける炎症性カスパーゼ誘導近接の可視化
Summary
このプロトコルは、ヒト血液サンプルから単球由来マクロファージ(MDM)を取得するワークフロー、細胞の生存率と挙動を損なうことなく炎症性カスパーゼ二分子蛍光相補(BiFC)レポーターをヒトMDMに効率的に導入する簡単な方法、および生細胞における炎症性カスパーゼ活性化を測定するためのイメージングベースのアプローチを記述しています。
Abstract
炎症性カスパーゼは、カスパーゼ−1、−4、−5、−11、および−12を含み、開始カスパーゼのサブグループに属する。カスパーゼ-1は、炎症シグナル伝達の正しい調節を確実にするために必要であり、インフラマソームへの動員に続く近接誘導二量体化によって活性化される。カスパーゼ-1は単球系細胞系譜に豊富であり、炎症誘発性サイトカインインターロイキン(IL)-1βおよびIL-18の成熟を活性分泌分子に誘導する。他の炎症性カスパーゼでは、カスパーゼ−4および−5(ならびにそれらのマウスホモログカスパーゼ−11)は、ピロトーシスを誘導することによってIL−1β放出を促進する。カスパーゼ二分子蛍光相補(BiFC)は、カスパーゼ活性化の読み出しとして炎症性カスパーゼ誘導近接を測定するために使用されるツールです。インフラマソームに結合する領域を含むカスパーゼ-1、-4、または-5プロドメインは、黄色蛍光タンパク質Venus(Venus-N [VN]またはVenus-C [VC])の非蛍光断片に融合され、カスパーゼが誘導近接したときに蛍光金星複合体を改質する。このプロトコルは、ヌクレオフェクションを使用してこれらのレポーターを初代ヒト単球由来マクロファージ(MDM)に導入する方法、炎症性カスパーゼ活性化を誘導するために細胞を治療する方法、および蛍光および共焦点顕微鏡を用いてカスパーゼ活性化を測定する方法を記述している。このアプローチの利点は、生細胞における炎症性カスパーゼ活性化複合体の成分、要件、および局在を同定するために使用することができることである。しかし、細胞の生存率と挙動を損なわないように、慎重な制御を考慮する必要があります。この技術は、インフラマソームレベルでの動的カスパーゼ相互作用の分析、ならびにヒト血液サンプル由来の生きたMDMおよび単球における炎症シグナル伝達カスケードの尋問のための強力なツールである。
Introduction
カスパーゼは、システインアスパラギン酸プロテアーゼのファミリーであり、イニシエーターカスパーゼおよび死刑執行人カスパーゼにグループ化することができる。死刑執行人カスパーゼは、カスパーゼ-3、-6および-7を含む。それらは二量体として細胞内に自然に見出され、アポトーシス1を実行するために開始カスパーゼによって切断される。開始カスパーゼには、ヒトカスパーゼ−1、−2、−4、−5、−8、−9、−10および−12が含まれる。それらは、近接誘導二量体化によって活性化され、自己タンパク質分解切断によって安定化される不活性ザイモゲン(プロカスパーゼ)として見出される2,3。炎症性カスパーゼは、開始カスパーゼ2のサブセットであり、ヒトにおけるカスパーゼ−1、−4、−5、および−12、ならびにマウス4、5におけるカスパーゼ−1、−11、および−12を包含する。アポトーシスの役割ではなく、炎症において中心的な役割を果たします。それらは、病原性侵入者8,9に応答して放出される最初のサイトカインであるプロインターロイキン(IL)−1βおよびプロIL−18 6,7のタンパク質分解的プロセシングおよび分泌を媒介する。カスパーゼ-1は、その活性化プラットフォームへの募集時に活性化される。インフラマソームと呼ばれる大きな分子量のタンパク質複合体(図1A)10。カスパーゼ−4、−5、および−11の二量体化は、非正準インフラマソーム経路11、12を介してこれらのプラットフォームとは独立して起こる。
正準インフラマソームは、インフラマソームセンサータンパク質、アダプタータンパク質ASC(CARDを含むアポトーシス関連スペック様タンパク質)、およびエフェクタータンパク質カスパーゼ-110からなる細胞質ゾル多量体タンパク質複合体である。最もよく研究された正準インフラマソームは、ピリンドメイン(NLRP)、NLRP1およびNLRP3を含むNOD様受容体ファミリー、CARD(NLRC)、NLRC4を含むNLRファミリー、および黒色腫2に存在しない(AIM2)である。それらはそれぞれ、ピリンドメイン、CARD、またはその両方のドメインを含む。CARD ドメインは、CARD を含むカスパーゼとそのアップストリーム アクティベーター間の相互作用を仲介します。したがって、N末端ピリンドメイン(PYD)とC末端CARDモチーフ13,14とからなる足場分子ASCは、NLRP110、NLRP3 15、およびAIM216インフラマソームへのカスパーゼ-1のリクルートに必要である。
各インフラマソームは、明確な炎症促進性刺激を認識する独自のセンサータンパク質にちなんで名付けられました(図1B)。この経路の活性化因子は、正準刺激と呼ばれる。インフラマソームは、微生物成分および組織ストレスのセンサとして機能し、炎症性カスパーゼ17の活性化を介して堅牢な炎症反応を誘発するように組み立てられる。インフラマソームアセンブリは、カスパーゼ−1活性化を開始し、その主要基質であるプロ−IL−1βおよびプロIL−18の成熟および分泌を媒介する。このプロセスは、2段階のメカニズムを介して行われます。第1に、プライミング刺激は、NF−κB経路の活性化を介して、ある種のインフラマソームタンパク質およびプロIL−1βの発現をアップレギュレートする。第二に、細胞内(正準)刺激は、プロカスパーゼ-1 6,7のインフラマソーム集合および動員を誘導する。
カスパーゼ−4およびカスパーゼ−5は、マウスカスパーゼ−11 11のヒトオルソログである。それらは、グラム陰性菌18、19、20の外膜に見られる分子である細胞内リポ多糖(LPS)および赤血球溶血21の産物である細胞外ヘムによって、インフラマソーム非依存的に活性化される。LPSがこれらのタンパク質のCARDモチーフに直接結合し、それらのオリゴマー化を誘導することが提案されている20。カスパーゼ-4またはカスパーゼ-5の活性化は、孔形成タンパク質ガスデルミンD(GSDMD)の切断を介してピロトーシスと呼ばれる炎症型の細胞死を誘導することによってIL-1β放出を促進する18,19。さらに、カスパーゼ−4およびGSDMD媒介性パイロプトーシス死から生じるカリウムイオンの流出は、NLRP3インフラマソームの活性化およびその後のカスパーゼ−1の活性化を誘導する22,23。したがって、カスパーゼ−4、−5、および−11は、特定の刺激に応答してパイロプトーシスおよびカスパーゼ−1活性化を誘導することができるLPSの細胞内センサと考えられる11、24。
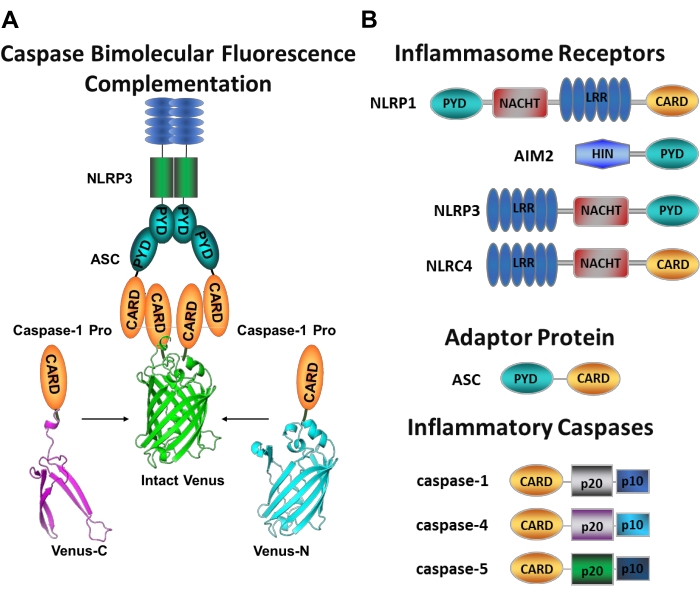
図1:炎症性カスパーゼおよびカスパーゼ-二分子蛍光相補(BiFC)アッセイ。(A)カスパーゼ-BiFC系を示す図では、金星の各非蛍光断片(Venus-CまたはVenus-N)に連結された2つのカスパーゼ-1プロドメイン(C1-pro)がNLRP3活性化プラットフォームにリクルートされ、金星にリフォールドと蛍光が強制されます。この複合体は、顕微鏡下で緑色の斑点として現れ、開始カスパーゼ活性化の最初のステップである炎症性カスパーゼ誘発近接の読み出しとして機能する。(b)インフラマソーム成分および炎症性カスパーゼのドメイン組織を示す模式図。この図の拡大版を表示するには、ここをクリックしてください。
特定の開始剤カスパーゼ活性化を測定することは困難であり、イメージングアプローチによってこれを行うために利用可能な方法は多くない。カスパーゼ二分子蛍光相補(BiFC)は、生細胞における炎症性カスパーゼ活性化を直接可視化するために使用できます(図1A)25。この技術は、最近、ヒト単球由来マクロファージ(MDM)21における使用に適応されている。カスパーゼBiFCは、炎症性カスパーゼ活性化における第1ステップを測定し、二量体化を促進するために近接を誘導する。CARD含有カスパーゼプロドメインをコードするプラスミドの発現は、光線上黄色蛍光タンパク質Venusの非蛍光断片に融合(Venus−C[VC])およびVenus−N[VN])が使用される。2つのカスパーゼプロドメインが活性化プラットフォームにリクルートされるか、または誘導された近接を受けると、金星の2つの半分が近接し、リフォールドおよび蛍光を発することを余儀なくされる( 図1A、B参照)。これは、特定の炎症性カスパーゼ活性化のリアルタイム読み出しを提供する。
ヒトMDMは、危険シグナルや病原体産物を同定するインフラマソーム遺伝子やパターン認識受容体を豊富に発現しています。これは、炎症性カスパーゼ経路の尋問に理想的な細胞型を提供する。さらに、それらは末梢血から、さらには患者サンプルから誘導して、特定の疾患状態における炎症性カスパーゼ活性化を評価することができる。このプロトコルでは、ヌクレオフェクションを使用してBiFCカスパーゼレポーターをMDMに導入する方法、エレクトロポレーションベースのトランスフェクション法、炎症性カスパーゼ活性化を誘導するために細胞を治療する方法、および顕微鏡アプローチを使用して活性カスパーゼ複合体を視覚化する方法について説明しています。さらに、この方法論は、これらの複合体の分子組成、細胞内局在化、動態、およびこれらの高度に秩序付けられた構造のサイズを決定するために適合させることができる25、26、27。
Protocol
Representative Results
Discussion
このプロトコルは、ヒト血液サンプルから単離された単球からマクロファージを得るワークフローと、細胞の生存率および挙動を損なうことなく炎症性カスパーゼBiFCレポーターをヒトMDMに効率的に導入する方法を記述している。
このプロトコルは、BiFC技術35 を利用して、カスパーゼリクルートメントドメイン(CARD)における炎症性カスパーゼを、分割蛍…
Disclosures
The authors have nothing to disclose.
Acknowledgements
この技術の開発に貢献したLBHの過去と現在の研究室のメンバーに感謝します。このラボは、NIH/NIDDK T32DK060445 (BEB)、NIH/NIDDK F32DK121479 (BEB)、NIH/NIGMS R01GM121389 (LBH) によってサポートされています。図2は、Biorenderソフトウェアを使用して描画されました。
Materials
| 48 well tissue culture2:34 plates | Genesee Scientific | 25-108 | |
| 10 cm Tissue Culture Dishes | VWR | 25382-166 | |
| 2 Mercaptoethanol 1000x | Thermo Fisher Scientific | 21985023 | |
| 8 well chambered coverglass with 1.5 HP coverglass | Cellvis | c8-1.5H-N | |
| AutoMACS columns | Miltenyi (Biotec) | 130-021-101 | For automated separation using AutoMACS Pro Separator only |
| AutoMACS Pro Separator | Miltenyi (Biotec) | 130-092-545 | For automated separation using AutoMACS Pro Separator only |
| AutoMACS Pro Washing Solution | Miltenyi (Biotec) | 130-092-987 | For automated separation using AutoMACS Pro Separator only |
| AutoMACS Rinsing Solution | Miltenyi (Biotec) | 130-091-222 | For automated separation using AutoMACS Pro Separator only |
| AutoMacs running buffer | Miltenyi (Biotec) | 130-091-221 | For manual or automated separation using QuadroMACS or AutoMACS pro Separator |
| Axio Observer Z1 motorized inverted microscope equipped with a CSU-X1A 5000 spinning disk unit | Zeiss | Any confocal microscope equipped with a laser module fitted with laser lines of 568 nm (RFP) and 488 or 512 nm (GFP or YFP) wavelengths can be used | |
| AxioObserver A1, Research Grade Inverted Microscope | Zeiss | Any epifluorescence microscope with fluorescence filters capable of exciting 568 nm (RFP) and 488 or 512 nm (GFP or YFP) wavelengths can be used | |
| CD14+ MICROBEADS | Miltenyi (Biotec) | 130-050-201 | For manual or automated separation using QuadroMACS or AutoMACS pro Separator |
| DPBS without calcium chloride and magnesium chloride | Sigma | D8537-6x500ML | |
| DsRed mito plasmid | Clontech | 632421 | Similar plasmids that can be used as fluorescent reporters can be found on Addgene |
| Fetal Bovine Serum | Thermo Fisher Scientific | 10437028 | |
| Ficoll-Paque PLUS 6 x 100 mL | Sigma | GE17-1440-02 | |
| GlutaMAX Supplement (100x) | Thermo Fisher Scientific | 35050079 | |
| GM-CSF | Thermo Fisher Scientific | PHC2011 | |
| Hemin BioXtra, from Porcine, ≥96.0% (HPLC) | Sigma | 51280-1G | |
| HEPES | Thermo Fisher Scientific | 15630106 | |
| Inflammatory caspase BiFC plasmids | Available by request from LBH lab | ||
| LPS-EB Ultrapure | Invivogen | TLRL-3PELPS | |
| LS Columns | Miltenyi (Biotec) | 130-042-401 | For manual separation using QuadroMACS Separator only |
| MACS 15 mL Tube Rack | Miltenyi (Biotec) | 130-091-052 | For manual separation using QuadroMACS Separator only |
| MACS MultiStand | Miltenyi (Biotec) | 130-042-303 | For manual separation using QuadroMACS Separator only |
| mCherry plasmid | Yungpeng Wang Lab | Similar plasmids that can be used as fluorescent reporters can be found on Addgene | |
| Neon Transfection System | Thermo Fisher Scientific | MPK5000 | Includes Neon electroporation device, pipette and pipette station |
| Neon Transfection System 10 µL Kit | Thermo Fisher Scientific | MPK1096 | Includes resuspension buffer R, resuspension buffer T, electrolytic buffer E, 96 x 10 µL Neon tips and Neon electroporation tubes |
| Nigericin sodium salt, ready made solution | Sigma | SML1779-1ML | |
| Penicillin-Streptomycin (10,000 U/mL) | Thermo Fisher Scientific | 15140122 | |
| Poly-D-Lysine Hydrobromide | Sigma | P7280-5mg | |
| QuadroMACS Separator | Miltenyi (Biotec) | 130-090-976 | For manual separation using QuadroMACS Separator only |
| qVD-OPh | Fisher (ApexBio) | 50-101-3172 | |
| RPMI 1640 Medium | Thermo Fisher Scientific | 11875119 | |
| Trypsin-EDTA (0.25%), phenol red | Thermo Fisher Scientific | 25200072 | |
| UltraPure 0.5 M EDTA, pH 8.0 | Thermo Fisher Scientific | 15575020 | |
| Zeiss Zen 2.6 (blue edition) software | Zeiss | Any software used to operate the confocal microscope of choice |
References
- Boatright, K. M., et al. A unified model for apical caspase activation. Molecular Cell. 11 (2), 529-541 (2003).
- Pop, C., Salvesen, G. S. Human caspases: activation, specificity, and regulation. Journal of Biological Chemistry. 284 (33), 21777-21781 (2009).
- Boice, A., Bouchier-Hayes, L. Targeting apoptotic caspases in cancer. Biochimica et Biophysica Acta (BBA) – Molecular Cell Research. 1867 (6), 118688 (2020).
- Bolívar, B. E., Vogel, T. P., Bouchier-Hayes, L. Inflammatory caspase regulation: maintaining balance between inflammation and cell death in health and disease. The FEBS Journal. 286 (14), 2628-2644 (2019).
- Martinon, F., Tschopp, J. Inflammatory caspases: linking an intracellular innate immune system to autoinflammatory diseases. Cell. 117 (5), 561-574 (2004).
- Lamkanfi, M., Vishva, M. D. Mechanisms and functions of inflammasomes. Cell. 157 (5), 1013-1022 (2014).
- Viganò, E., et al. Human caspase-4 and caspase-5 regulate the one-step noncanonical inflammasome activation in monocytes. Nature Communications. 6, 8761 (2015).
- Cerretti, D. P., et al. Molecular cloning of the interleukin-1 beta converting enzyme. Science. 256 (5053), 97 (1992).
- van de Veerdonk, F. L., Netea, M. G., Dinarello, C. A., Joosten, L. A. Inflammasome activation and IL-1β and IL-18 processing during infection. Trends in Immunology. 32 (3), 110-116 (2011).
- Martinon, F., Burns, K., Tschopp, J. The inflammasome: a molecular platform triggering activation of inflammatory caspases and processing of proIL-beta. Molecular Cell. 10 (2), 417-426 (2002).
- Kayagaki, N., et al. Noncanonical inflammasome activation targets caspase-11. Nature. 479 (7371), 117-121 (2011).
- Kayagaki, N., et al. Noncanonical inflammasome activation by intracellular LPS independent of TLR4. Science. 341 (6151), 1246-1249 (2013).
- Masumoto, J., et al. ASC, a novel 22-kDa protein, aggregates during apoptosis of human promyelocytic leukemia HL-60 cells. The Journal of Biological Chemistry. 274 (48), 33835-33838 (1999).
- Bertin, J., DiStefano, P. S. The PYRIN domain: a novel motif found in apoptosis and inflammation proteins. Cell Death and Differentiation. 7 (12), 1273-1274 (2000).
- Agostini, L., et al. NALP3 forms an IL-1beta-processing inflammasome with increased activity in Muckle-Wells autoinflammatory disorder. Immunity. 20 (3), 319-325 (2004).
- Bürckstümmer, T., et al. An orthogonal proteomic-genomic screen identifies AIM2 as a cytoplasmic DNA sensor for the inflammasome. Nature Immunology. 10 (3), 266-272 (2009).
- Latz, E., Xiao, T. S., Stutz, A. Activation and regulation of the inflammasomes. Nature Reviews Immunology. 13 (6), 397-411 (2013).
- Kayagaki, N., et al. Caspase-11 cleaves gasdermin D for noncanonical inflammasome signalling. Nature. 526 (7575), 666-671 (2015).
- Shi, J., et al. Cleavage of GSDMD by inflammatory caspases determines pyroptotic cell death. Nature. 526 (7575), 660-665 (2015).
- Shi, J., et al. Inflammatory caspases are innate immune receptors for intracellular LPS. Nature. 514 (7521), 187-192 (2014).
- Bolívar, B. E., et al. Noncanonical Roles of Caspase-4 and Caspase-5 in Heme-Driven IL-1β Release and Cell Death. The Journal of Immunology. 206 (8), 1878-1889 (2021).
- Schmid-Burgk, J. L., et al. Caspase-4 mediates noncanonical activation of the NLRP3 inflammasome in human myeloid cells. European Journal of Immunology. 45 (10), 2911-2917 (2015).
- Baker, P. J., et al. NLRP3 inflammasome activation downstream of cytoplasmic LPS recognition by both caspase-4 and caspase-5. European Journal of Immunology. 45 (10), 2918-2926 (2015).
- Cullen, S. P., Kearney, C. J., Clancy, D. M., Martin, S. J. Diverse activators of the NLRP3 inflammasome promote IL-1β secretion by triggering necrosis. Cell Reports. 11 (10), 1535-1548 (2015).
- Sanders, M. G., et al. Single-cell imaging of inflammatory caspase dimerization reveals differential recruitment to inflammasomes. Cell Death & Disease. 6, 1813 (2015).
- Charendoff, C. I., Bouchier-Hayes, L. Lighting up the pathways to caspase activation using bimolecular fluorescence complementation. Journal of Visualized Experiments: JoVE. (133), e57316 (2018).
- Bouchier-Hayes, L., et al. Characterization of cytoplasmic caspase-2 activation by induced proximity. Molecular Cell. 35 (6), 830-840 (2009).
- Riedhammer, C., Halbritter, D., Weissert, R. Peripheral blood mononuclear cells: Isolation, Freezing, thawing, and culture. Methods in Molecular Biology. 1304, 53-61 (2016).
- Betsou, F., Gaignaux, A., Ammerlaan, W., Norris, P. J., Stone, M. Biospecimen science of blood for peripheral blood mononuclear cell (PBMC) functional applications. Current Pathobiology Reports. 7 (2), 17-27 (2019).
- Perregaux, D., et al. IL-1 beta maturation: evidence that mature cytokine formation can be induced specifically by nigericin. The Journal of Immunology. 149 (4), 1294-1303 (1992).
- Cheneval, D., et al. Increased mature interleukin-1β (IL-1β) secretion from THP-1 cells induced by nigericin is a result of activation of p45 IL-1β-converting enzyme processing. Journal of Biological Chemistry. 273 (28), 17846-17851 (1998).
- Fernandes-Alnemri, T., et al. The pyroptosome: a supramolecular assembly of ASC dimers mediating inflammatory cell death via caspase-1 activation. Cell Death and Differentiation. 14 (9), 1590-1604 (2007).
- Lu, A., et al. Unified polymerization mechanism for the assembly of ASC-dependent inflammasomes. Cell. 156 (6), 1193-1206 (2014).
- Muller-Eberhard, U., Javid, J., Liem, H. H., Hanstein, A., Hanna, M. Brief report: Plasma concentrations of hemopexin, haptoglobin and heme in patients with various hemolytic diseases. Blood. 32 (5), 811-815 (1968).
- Shyu, Y. J., Liu, H., Deng, X., Hu, C. D. Identification of new fluorescent protein fragments for bimolecular fluorescence complementation analysis under physiological conditions. Biotechniques. 40 (1), 61-66 (2006).
- Thornberry, N. A., et al. A novel heterodimeric cysteine protease is required for interleukin-1βprocessing in monocytes. Nature. 356 (6372), 768-774 (1992).
- Jensen, K., Anderson, J. A., Glass, E. J. Comparison of small interfering RNA (siRNA) delivery into bovine monocyte-derived macrophages by transfection and electroporation. Veterinary Immunology and Immunopathology. 158 (3-4), 224-232 (2014).
- Tada, Y., Sakamoto, M., Fujimura, T. Efficient gene introduction into rice by electroporation and analysis of transgenic plants: use of electroporation buffer lacking chloride ions. Theoretical and Applied Genetics. 80 (4), 475-480 (1990).
- Bhowmik, P., et al. Targeted mutagenesis in wheat microspores using CRISPR/Cas9. Scientific Reports. 8 (1), 6502 (2018).
- Sansom, D. M., Manzotti, C. N., Zheng, Y. What’s the difference between CD80 and CD86. Trends in Immunology. 24 (6), 314-319 (2003).
- Kurokawa, M., Kornbluth, S. Caspases and kinases in a death grip. Cell. 138 (5), 838-854 (2009).

