Erfassung der Chromosomenkonformation über Längenskalen hinweg
Summary
Hi-C 3.0 ist ein verbessertes Hi-C-Protokoll, das Formaldehyd- und Disuccinimidylglutarat-Vernetzer mit einem Cocktail aus DpnII- und DdeI-Restriktionsenzymen kombiniert, um das Signal-Rausch-Verhältnis und die Auflösung der Chromatin-Interaktionsdetektion zu erhöhen.
Abstract
Chromosomenkonformationserfassung (3C) wird verwendet, um dreidimensionale Chromatin-Wechselwirkungen zu erkennen. Typischerweise wird chemische Vernetzung mit Formaldehyd (FA) verwendet, um Chromatinwechselwirkungen zu fixieren. Dann wandelt der Chromatinaufschluss mit einem Restriktionsenzym und die anschließende Religation der Fragmentenden dreidimensionale (3D) Nähe in einzigartige Ligationsprodukte um. Schließlich wird die DNA nach der Umkehrung der Vernetzungen, der Proteinentfernung und der DNA-Isolierung geschert und für die Hochdurchsatzsequenzierung vorbereitet. Die Häufigkeit der Annäherungsligation von Loci-Paaren ist ein Maß für die Häufigkeit ihrer Kolokalisation im dreidimensionalen Raum in einer Zellpopulation.
Eine sequenzierte Hi-C-Bibliothek liefert genomweite Informationen über Interaktionsfrequenzen zwischen allen Loci-Paaren. Die Auflösung und Präzision von Hi-C beruht auf einer effizienten Vernetzung, die Chromatinkontakte und eine häufige und gleichmäßige Fragmentierung des Chromatins aufrechterhält. Dieser Artikel beschreibt ein verbessertes In-situ-Hi-C-Protokoll, Hi-C 3.0, das die Effizienz der Vernetzung durch Kombination von zwei Vernetzern (Formaldehyd [FA] und Disuccinimidylglutarat [DSG]) erhöht, gefolgt von einem feineren Aufschluss mit zwei Restriktionsenzymen (DpnII und DdeI). Hi-C 3.0 ist ein einziges Protokoll für die genaue Quantifizierung von Genomfaltungsmerkmalen auf kleineren Skalen wie Schleifen und topologisch assoziierenden Domänen (TADs) sowie von Merkmalen auf größeren kernweiten Skalen wie Kompartimenten.
Introduction
Chromosomenkonformationserfassung wird seit 2002 verwendet1. Grundsätzlich beruht jede Konformationserfassungsvariante auf der Fixierung von DNA-Protein- und Protein-Protein-Wechselwirkungen, um die 3D-Chromatinorganisation zu erhalten. Es folgt eine DNA-Fragmentierung, in der Regel durch Restriktionsverdauung, und schließlich endet die Religation der nahe gelegenen DNA, um räumlich proximale Loci in einzigartige kovalente DNA-Sequenzen umzuwandeln. Anfängliche 3C-Protokolle verwendeten PCR, um spezifische “Eins-zu-Eins”-Interaktionen abzutasten. Nachfolgende 4C-Assays ermöglichten den Nachweis von “One-to-all”-Interaktionen2, während 5C “many-to-many”-Interaktionen3 detektierten. Die Erfassung von Chromosomenkonformationen kam nach der Implementierung der Hochdurchsatzsequenzierung (NGS) der nächsten Generation, die den Nachweis von “all-to-all” genomischen Interaktionen mit genomweitem Hi-C4 und vergleichbaren Techniken wie 3C-seq5, TCC6 und Micro-C7,8 ermöglichte, vollständig zum Tragen (siehe auch Review von Denker und De Laat9).
In Hi-C werden biotinylierte Nukleotide verwendet, um 5′-Überhänge nach dem Verdauung und vor der Ligation zu markieren (Abbildung 1). Dies ermöglicht die Auswahl von richtig verdauten und religierten Fragmenten mit Streptavidin-beschichteten Perlen und unterscheidet sie von GCC10. Eine wichtige Aktualisierung des Hi-C-Protokolls wurde von Rao et al.11 implementiert, die die Verdauung und Religation in intakten Kernen (d.h. in situ) durchführten, um falsche Ligaturprodukte zu reduzieren. Darüber hinaus reduzierte der Ersatz des HindIII-Aufschlusses durch den MboI- (oder DpnII-) Aufschluss die Fragmentgröße und erhöhte das Auflösungspotenzial von Hi-C. Dieser Anstieg ermöglichte den Nachweis relativ kleiner Strukturen und eine genauere genomische Lokalisierung von Kontaktpunkten, wie z.B. DNA-Schleifen zwischen kleinen cis-Elementen, z.B. Schleifen zwischen CTCF-gebundenen Stellen, die durch Schleifenextrusionerzeugt werden 11,12. Dieses Potenzial hat jedoch seinen Preis. Erstens erfordert eine zweifache Erhöhung der Auflösung eine vierfache (22) Erhöhung der Sequenzierungslesevorgänge13. Zweitens erhöhen die kleinen Fragmentgrößen die Möglichkeit, unverdaute benachbarte Fragmente mit verdauten und religierten Fragmenten zu verwechseln14. Wie bereits erwähnt, unterscheiden sich bei Hi-C verdaute und religierte Fragmente von unverdauten Fragmenten durch die Anwesenheit von Biotin an der Ligationsverbindung. Es ist jedoch eine ordnungsgemäße Biotinentfernung von unligierten Enden erforderlich, um sicherzustellen, dass nur Ligationsverbindungen nach unten gezogenwerden 14,15.
Mit den sinkenden Kosten von NGS wird es möglich, die Chromosomenfaltung genauer zu untersuchen. Um die Größe von DNA-Fragmenten zu verringern und dadurch die Auflösung zu erhöhen, kann das Hi-C-Protokoll angepasst werden, um häufiger schneidende Restriktionsenzyme16 oder Kombinationen von Restriktionsenzymen 17,18,19 zu verwenden. Alternativ können MNase 7,8 in Micro-C und DNase in DNase Hi-C20 titriert werden, um eine optimale Verdauung zu erreichen.
Eine kürzlich durchgeführte systematische Auswertung der Grundlagen von 3C-Methoden zeigte, dass sich die Detektion von Chromosomenfaltungsmerkmalen auf jeder Längenskala mit sequentieller Vernetzung mit 1% FA gefolgt von 3 mM DSG17 stark verbesserte. Darüber hinaus war Hi-C mit HindIII-Aufschluss die beste Option zum Nachweis großräumiger Faltungsmerkmale wie Kompartimente, und Micro-C war bei der Erkennung kleiner Faltungsmerkmale wie DNA-Schleifen überlegen. Diese Ergebnisse führten zur Entwicklung einer einzigen, hochauflösenden “Hi-C 3.0”-Strategie, die die Kombination von FA- und DSG-Vernetzern gefolgt von einem Doppelaufschluss mit DpnII- und DdeI-Endonukleasen verwendet21. Hi-C 3.0 bietet eine effektive Strategie für den allgemeinen Gebrauch, da es Faltmerkmale über alle Längenskalen hinweg genau erkennt17. Der experimentelle Teil des Hi-C 3.0-Protokolls wird hier detailliert beschrieben und typische Ergebnisse, die nach der Sequenzierung zu erwarten sind, werden gezeigt.
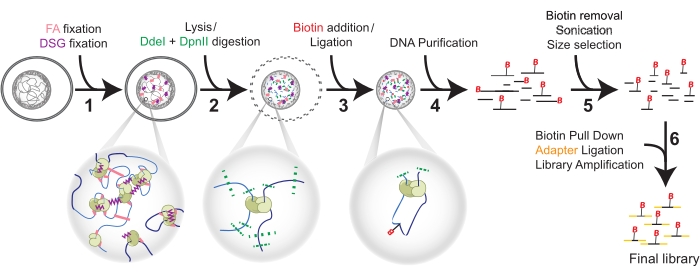
Abbildung 1: Hi-C-Verfahren in sechs Schritten. Die Zellen werden zuerst mit FA und dann mit DSG fixiert (1). Dann geht die Lyse einer Doppelverdauung mit DdeI und DpnII voraus (2). Biotin wird durch Überhangfüllung hinzugefügt und proximal abgestumpfte Enden werden vor der DNA-Reinigung ligiert (3). Biotin wird vor der Beschallung und Größenauswahl von unligierten Enden entfernt (5). Schließlich ermöglicht das Pull-down von Biotin eine Adapterligation und Bibliotheksamplifikation durch PCR (6). Abkürzungen: FA = Formaldehyd; DSG = Disuccinimidylglutarat; B = Biotin. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Protocol
Representative Results
Discussion
Kritische Schritte für das Zellhandling
Obwohl es möglich ist, eine geringere Anzahl von Eingangszellen zu verwenden, wurde dieses Protokoll für ~5 × 106 Zellen pro Sequenzierungsspur (~400 M Lesevorgänge) optimiert, um eine angemessene Komplexität nach der tiefen Sequenzierung zu gewährleisten. Zellen werden am besten vor der Fixierung gezählt. Für die Generierung von Ultradeep-Bibliotheken multiplizieren wir in der Regel die Anzahl der Lanes (und Zellen), bis die gewünschte Lesetiefe erreicht ist. Für eine optimale Fixierung sollte das serumhaltige Medium vor der FA-Fixierung durch PBS ersetzt werden, und Fixierlösungen sollten sofort und ohne Konzentrationsgradienten15,22 hinzugefügt werden. Für die Zellernte wird das Schaben der Trypsinisierung vorgezogen, da der Übergang von einer flacheren zu einer kugelförmigen Form nach der Trypsinisierung die Kernkonformation beeinflussen könnte. Nach der Zugabe von DSG gehen lose und klumpige Zellpellets leicht verloren. Seien Sie vorsichtig beim Umgang mit Zellen in diesem Stadium und fügen Sie bis zu 0,05% BSA hinzu, um die Verklumpung zu verringern.
Änderungen an der Methode
Dieses Protokoll wurde unter Verwendung menschlicher Zellenentwickelt 17. Basierend auf Erfahrungen mit der Erfassung von Chromosomen sollte dieses Protokoll jedoch für die meisten eukaryotischen Zellen funktionieren. Für einen signifikant geringeren Input (~1 × 106 Zellen) empfehlen wir, die Hälfte der Volumina für die Lyse- und Exterieurerfassungsverfahren zu verwenden [Schritte 2.1-2.4]. Dies würde auch die DNA-Isolierung [Schritt 2.5] in einer Tischzentrifuge mit 1,7-ml-Röhrchen ermöglichen, was die Pelletierung bei niedrigen DNA-Konzentrationen verbessern könnte. Die Quantifizierung der DNA (Schritt 2.6.6) zeigt das weitere Vorgehen an. Für geringe Mengen isolierter DNA (1-5 μg) empfehlen wir, die Größenauswahl (Schritt 3.3) zu überspringen und mit der Biotinentfernung fortzufahren, nachdem das Volumen mit einer KBE von 130 μL auf ~45 μL reduziert wurde.
Dieses Protokoll wurde speziell entwickelt, um qualitativ hochwertige Daten nach anschließender Vernetzung mit FA und DSG und Aufschluss mit DpnII und DdeI zu gewährleisten. Alternative Vernetzungsstrategien wie FA gefolgt von EGS (Ethylenglykolbis(succinimidylsuccinat)), das auch in ChIP-seq23 und ChIA-PET24 verwendet wird, könnten jedoch ebenso gut funktionieren17. Ebenso können verschiedene Enzymkombinationen wie DpnII und HinfI18 oder MboI, MseI und NlaIII19 für die Verdauung verwendet werden. Achten Sie bei der Anpassung von Enzymkombinationen darauf, biotinylierte Nukleotide zu verwenden, die die spezifischen 5′-Überhänge ausfüllen können, und verwenden Sie die optimalen Puffer für jeden Cocktail. DpnII wird mit einem eigenen Puffer geliefert und der Enzymhersteller empfiehlt einen speziellen Puffer für die DdeI-Verdauung. Für die Doppelverdauung mit DpnII und DdeI in diesem Protokoll wird jedoch Restriction Buffer empfohlen, da es für beide Enzyme mit 100% Aktivität bewertet wird.
Fehlerbehebung bei der Konformationserfassung
Die drei wichtigsten Schritte bei der Erfassung der Chromosomenkonformation: Crosslinking, Verdauung und Religation wurden alle durchgeführt, bevor die Ergebnisse auf Gel visualisiert werden können. Um die Qualität jedes dieser drei Schritte zu bestimmen und zu erkennen, wo Probleme aufgetreten sein könnten, werden Aliquots vor (CI) und nach dem Aufschluss (DC) entnommen und zusammen mit der ligierten Hi-C-Probe auf das Gel geladen (Abbildung 2). Dieses Gel wird verwendet, um die Qualität der Hi-C-Probe zu bestimmen und ob es sich lohnt, das Protokoll fortzusetzen. Ohne CI und DC ist es schwierig, potenzielle suboptimale Schritte zu lokalisieren. Es ist erwähnenswert, dass eine suboptimale Ligatur auf ein Problem in der Ligatur selbst, das Ausfüllen oder ein Problem mit der Vernetzung zurückzuführen sein kann. Um Probleme bei der Vernetzung zu beheben, achten Sie darauf, nicht mehr als 1 × 107 Zellen pro Bibliothek zu verwenden, und beginnen Sie mit frischen Vernetzungsreagenzien und sauberen Zellen (d. h. mit PBS gespült). Stellen Sie für die Ligatur sicher, dass Zellen und Ligationsmischung auf Eis gehalten werden. T4-DNA-Ligase kurz vor der 4-stündigen Inkubation bei 16 °C hinzufügen und gut mischen.
Problembehandlung bei der Bibliotheksvorbereitung
Wenn mehr als 10 PCR-Zyklen erforderlich sind oder nach der PCR-Titration kein PCR-Produkt auf Gel zu sehen ist (Abbildung 4), gibt es einige Möglichkeiten, die Hi-C-Probe zu speichern. Nach der PCR-Titration besteht die erste Möglichkeit darin, die PCR erneut zu versuchen. Wenn immer noch nicht genügend Produkt vorhanden ist, ist es möglich, nach zweimaligem Waschen der Perlen mit 1x TLE-Puffer eine weitere Runde A-Tailing und Adapterligation (Schritt 3.6) zu versuchen. Nach dieser zusätzlichen A-Tailing und Adapterligatur kann man wie bisher zur PCR-Titration übergehen. Wenn immer noch kein Produkt vorhanden ist, besteht die letzte Option darin, den 0,8x-Anteil aus Schritt 3.3 zu wiederholen und von dort aus fortzufahren.
Einschränkungen und Vorteile von Hi-C3.0
Es ist wichtig zu wissen, dass Hi-C eine populationsbasierte Methode ist, die die durchschnittliche Häufigkeit von Interaktionen zwischen Loci-Paaren in der Zellpopulation erfasst. Einige Computeranalysen sind darauf ausgelegt, Kombinationen von Konformationen aus einer Population25 zu entwirren, aber im Prinzip ist Hi-C blind für Unterschiede zwischen Zellen. Obwohl es möglich ist, Einzelzell-Hi-C26,27 durchzuführen und rechnerische Schlussfolgerungen28 zu ziehen, ist Einzelzell-Hi-C nicht geeignet, ultrahochauflösende 3C-Informationen zu erhalten. Eine weitere Einschränkung von Hi-C besteht darin, dass es nur paarweise Wechselwirkungen erkennt. Um Multikontakt-Interaktionen zu erkennen, kann man entweder häufige Cutter in Kombination mit Short-Read-Sequenzierung (Illumina)16 verwenden oder Multicontact 3C29 oder 4C30 mit Long-Read-Sequenzierung von PacBio- oder Oxford Nanopore-Plattformen durchführen. Hi-C-Derivate zur spezifischen Detektion von Kontakten zwischen und entlang von Schwesterchromatiden wurden ebenfalls entwickelt31,32.
Obwohl Hi-C19 und Micro-C33 verwendet werden können, um Kontaktkarten mit Subkilobasenauflösungen zu erstellen, erfordern beide eine große Menge an Sequenzierungslesevorgängen, was zu einem kostspieligen Unterfangen werden kann. Um ohne Kosten eine ähnliche oder sogar höhere Auflösung zu erreichen, kann eine Anreicherung für bestimmte genomische Regionen (capture-C 34) oder spezifische Proteininteraktionen (ChiA-PET 35, PLAC-seq36, Hi-ChIP37) angewendet werden. Die Stärke und der Nachteil dieser Anreicherungsanwendungen besteht darin, dass nur eine begrenzte Anzahl von Interaktionen untersucht wird. Mit solchen Anreicherungen geht der globale Aspekt von Hi-C (und die Option der globalen Normalisierung) verloren.
Bedeutung und Einsatzmöglichkeiten von Hi-C3.0
Dieses Protokoll wurde entwickelt, um hochauflösendes, ultratiefes 3C zu ermöglichen und gleichzeitig großflächige Faltmerkmale wie TADs und Fächer17 zu erkennen (Abbildung 6). Dieses Protokoll beginnt mit 5 × 106 Zellen pro Röhre für jede Hi-C-Bibliothek, was mehr als genug Material sein sollte, um eine oder zwei Bahnen auf einer Flusszelle zu sequenzieren, um bis zu 1 Milliarde Paired-End-Lesevorgänge zu erhalten. Für die ultratiefe Sequenzierung sollten mehrere Röhrchen mit 5 × 106 Zellen vorbereitet werden, abhängig von der Anzahl der kartierten Reads und PCR-Duplikate. Bei der höchsten Auflösung (<1 kb) werden Looping-Interaktionen meist zwischen CTCF-Stellen gefunden, aber auch Promotor-Enhancer-Interaktionen können nachgewiesen werden. Leser können sich auf Akgol Oksuz et al.17 für eine detaillierte Beschreibung der Datenanalyse beziehen.
Disclosures
The authors have nothing to disclose.
Acknowledgements
Wir danken Denis Lafontaine für die Protokollentwicklung und Sergey Venev für die bioinformatische Unterstützung. Diese Arbeit wurde durch einen Zuschuss des National Institutes of Health Common Fund 4D Nucleome Program an J.D. (U54-DK107980, UM1-HG011536) unterstützt. J.D. ist ein Forscher des Howard Hughes Medical Institute.
Dieser Artikel unterliegt der HHMI-Richtlinie Open Access to Publications. HHMI-Laborleiter haben HHMI in ihren Forschungsartikeln zuvor eine nicht-exklusive CC BY 4.0-Lizenz und HHMI eine unterlizenzierbare Lizenz gewährt. Gemäß diesen Lizenzen kann das vom Autor akzeptierte Manuskript dieses Artikels unmittelbar nach der Veröffentlichung unter einer CC BY 4.0-Lizenz frei verfügbar gemacht werden.
Materials
| 1 kb Ladder | New England Biolabs | N3232L | |
| Agarose | Invitrogen | 16500100 | |
| Agencourt AMPure XP magnetic beads , 60 mL | Beckman Coulter | A63881 | |
| Amicon Ultra-0.5 Centrifugal Filter Unit (CFU) | EMD Millipore | UFC500396 | |
| Annealing Buffer (5x) | See recipe in supplemental materials | ||
| ATP 10 mM | ThermoFisher | R0441 | |
| Avanti J-25i High Speed Refrigerated ultra-centrifuge | Beckman Coulter | ||
| beckman ultracentrifuge tube 35 mL | Beckman Coulter | 357002 | |
| Binding Buffer (2x) | See recipe in supplemental materials | ||
| biotin-14-dATP 0.4 mM | Invitrogen | 19524-016 | |
| BSA 10 mg/mL | New England Biolabs | B9000S | dilute from 20 mg/mL |
| Cell scraper | Falcon | 353089 | |
| Cell scraper | Corning | 3008 | |
| Conical polypropylene tubes 50 mL | Denville | C1062-P | |
| Conical tube 15 mL | Denville | C1017-P | |
| Covaris micro tube AFA fiber with snap-cap 130 µL | Covaris | 520045/520077 | |
| Covaris Sonicator | Covaris | E220/E220evolution/M220 | |
| Culture flask 175 cm2 | Falcon | 353112 | |
| Culture plates 150 mm x 25 mm | Corning | 430599 | |
| dATP 1 mM | Invitrogen | 56172 | |
| dATP 10 mM | Invitrogen | 56172 | |
| dCTP 10 mM | Invitrogen | 56173 | |
| DdeI | New England Biolabs | R0175L | |
| dGTP 10 mM | Invitrogen | 56174 | |
| DMSO | Sigma | D2650-5x10ML | |
| dNTP mix 25 mM | Invitrogen | 10297117 | |
| Dounce homogenizer | DWK Life Sciences | 8853010002/8853030002 | |
| DPBS | Gibco | 14190-144 | |
| DpnII | New England Biolabs | R0543M | |
| DSG | ThermoScientific | 20593 | |
| dTTP 10 mM | Invitrogen | 56175 | |
| Ethanol 70% | Fisher | A409-4 | Diluted from 100% |
| Ethidium Bromide | Fisher | BP1302-10 | |
| Formaldehyde (37%) | Fisher | BP531-500 | |
| Gel loading dye (6x ) | New England Biolabs | B7024S | |
| Glycine in ultrapure water 2.5 M | Sigma | G8898-1KG | |
| HBSS | Gibco | 14025-092 | |
| Igepal CA-630 detergent | MP Biomedicals | 198596 | |
| Klenow DNA polymerase 5 U/µL | New England Biolabs | M0210L | |
| Klenow Fragment 3–>5’ exo-, 5 U/µL | New England Biolabs | M0212L | |
| ligation buffer (10x) | New England Biolabs | B7203S | |
| Liquid nitrogen | |||
| LoBind microcentrifuge tube 1.7 mL | Eppendorf | 22431021 | |
| Low Molecular Weight DNA Ladder | New England Biolabs | N3233L | |
| Lysis buffer | See recipe in supplemental materials | ||
| Magnetic Particle separator | ThermoFisher | 12321D | |
| Microfuge tubes 1.7 mL | Axygen | MCT-175-C | |
| MyOne Streptavidin C1 beads | Invitrogen | 65001 | |
| NEBuffer 2.1 (10x) | New England Biolabs | B7002S | |
| NEBuffer 3.1 (10x) | New England Biolabs | B7203S | |
| PBS | Gibco | 70013-032 | |
| PCR (strip) tubes | Biorad | TBS0201/ TCS0803 | |
| PCR thermocycler | Biorad | T100 | |
| Pfu Ultra II Buffer (10x) | Agilent | Comes with Pfu Ultra | |
| PfuUltra II Fusion HS DNA Polymerase | Agilent | 600674 | |
| Phase lock tube 15 mL | Qiagen | 129065 | |
| Phase lock tubes 2 mL | Qiagen | 129056 | |
| Phenol:chloroform:isoamyl alcohol | Invitrogen | 15593-049 | |
| Protease inhibitor cocktail | ThermoFisher | 78440 | |
| Proteinase K in ultrapure water 10 mg/mL | Invitrogen | 25530-031 | |
| Refrigerated Centrifuge | Eppendorf | 5810R | |
| RNase A, DNase and protease-free 10 mg/mL | Thermo Scientific | EN0531 | |
| Rotator | Argos technologies | EW-04397-40 or rocking platform | |
| SDS 1% | Fisher | BP13111 | |
| Sodium acetate pH = 5.2, 3 M | Sigma | ||
| Sub-Cell GT Horizontal Electrophoresis System | Biorad | 1704401 | |
| T4 DNA ligase 1 U/µL | Invitrogen | 100004817 | |
| T4 DNA polymerase | New England Biolabs | M0203L | |
| T4 DNA polymerase | New England Biolabs | M0203L | |
| T4 DNA polymerase 3 U/µL | New England Biolabs | M0203L | |
| T4 ligation buffer (5x) | Invitrogen | Y90001 | |
| T4 polynucleotide kinase 10 U/µL | New England Biolabs | M0201L | |
| Tabletop centrifuge | Eppendorf | 5425 | |
| TBE buffer | See recipe in supplemental materials | ||
| Tris Low EDTA Buffer (TLE) | See recipe in supplemental materials | ||
| Triton X-100 (10%) | Sigma | 93443 | |
| Truseq adapter oligos | Integrated DNA Technologies (IDT)) | https://www.idtdna.com/site/order/oligoentry | 250 nmole and HPLC purified |
| Tween 20 detergent | Fisher | 9005-64-5 | |
| Tween Wash Buffer | See recipe in supplemental materials | ||
| Vortex | Scientific Industries | (G560)SI-0236 |
References
- Dekker, J., Rippe, K., Dekker, M., Kleckner, N. Capturing chromosome conformation. Science. 295 (5558), 1306-1311 (2002).
- Simonis, M., et al. Nuclear organization of active and inactive chromatin domains uncovered by chromosome conformation capture-on-chip (4C). Nature Genetics. 38 (11), 1348-1354 (2006).
- Dostie, J., et al. Chromosome Conformation Capture Carbon Copy (5C): a massively parallel solution for mapping interactions between genomic elements. Genome Research. 16 (10), 1299-1309 (2006).
- Lieberman-Aiden, E., et al. Comprehensive mapping of long-range interactions reveals folding principles of the human genome. Science. 326 (5950), 289-293 (2009).
- Stadhouders, R., et al. Multiplexed chromosome conformation capture sequencing for rapid genome-scale high-resolution detection of long-range chromatin interactions. Nature Protocols. 8 (3), 509-524 (2013).
- Kalhor, R., Tjong, H., Jayathilaka, N., Alber, F., Chen, L. Genome architectures revealed by tethered chromosome conformation capture and population-based modeling. Nature Biotechnology. 30 (1), 90-98 (2012).
- Hsieh, T. H., et al. Mapping nucleosome resolution chromosome folding in yeast by micro-C. Cell. 162 (1), 108-119 (2015).
- Hsieh, T. -. H. S., Fudenberg, G., Goloborodko, A., Rando, O. J. Micro-C XL: assaying chromosome conformation from the nucleosome to the entire genome. Nature Methods. 13 (12), 1009-1011 (2016).
- Denker, A., de Laat, W. The second decade of 3C technologies: detailed insights into nuclear organization. Genes & Development. 30 (12), 1357-1382 (2016).
- Rodley, C. D., Bertels, F., Jones, B., O’Sullivan, J. M. Global identification of yeast chromosome interactions using Genome conformation capture. Fungal Genetics and Biology. 46 (11), 879-886 (2009).
- Rao, S. S., et al. A 3D map of the human genome at kilobase resolution reveals principles of chromatin looping. Cell. 159 (7), 1665-1680 (2014).
- Alipour, E., Marko, J. F. Self-organization of domain structures by DNA-loop-extruding enzymes. Nucleic Acids Research. 40 (22), 11202-11212 (2012).
- Lajoie, B. R., Dekker, J., Kaplan, N. The Hitchhiker’s guide to Hi-C analysis: Practical guidelines. Methods. 72, 65-75 (2015).
- Belaghzal, H., Dekker, J., Gibcus, J. H. Hi-C 2.0: An optimized Hi-C procedure for high-resolution genome-wide mapping of chromosome conformation. Methods. 123, 56-65 (2017).
- Golloshi, R., Sanders, J. T., McCord, R. P. Iteratively improving Hi-C experiments one step at a time. Methods. 142, 47-58 (2018).
- Darrow, E. M., et al. Deletion of DXZ4 on the human inactive X chromosome alters higher-order genome architecture. Proceedings of the National Academy of Sciences of the United States of America. 113 (31), 4504-4512 (2016).
- Akgol Oksuz, B., et al. Systematic evaluation of chromosome conformation capture assays. Nature Methods. 18 (9), 1046-1055 (2021).
- Ghuryeid, J., et al. Integrating Hi-C links with assembly graphs for chromosome-scale assembly. PLoS Computational Biology. 15 (8), 1007273 (2019).
- Gu, H., et al. Fine-mapping of nuclear compartments using ultra-deep Hi-C shows that active promoter and enhancer elements localize in the active A compartment even when adjacent sequences do not. bioRxiv. , (2021).
- Ramani, V., et al. Mapping 3D genome architecture through in situ DNase Hi-C. Nature Protocols. 11 (11), 2104-2121 (2016).
- Lafontaine, D. L., Yang, L., Dekker, J., Gibcus, J. H. Hi-C 3.0: Improved Protocol for Genome-Wide Chromosome Conformation Capture. Current Protocols. 1 (7), 198 (2021).
- Belton, J. -. M. M., et al. Hi-C: A comprehensive technique to capture the conformation of genomes. Methods. 58 (3), 268-276 (2012).
- Truch, J., Telenius, J., Higgs, D. R., Gibbons, R. J. How to tackle challenging ChIP-Seq, with long-range cross-linking, Using ATRX as an example. Methods in Molecular Biology. 1832, 105-130 (2018).
- Wang, P., et al. In situ chromatin interaction analysis using paired-end tag sequencing. Current Protocols. 1 (8), 174 (2021).
- Tjong, H., et al. Population-based 3D genome structure analysis reveals driving forces in spatial genome organization. Proceedings of the National Academy of Sciences of the United States of America. 113 (12), 1663-1672 (2016).
- Nagano, T., et al. Cell-cycle dynamics of chromosomal organization at single-cell resolution. Nature. 547 (7661), 61-67 (2017).
- Ramani, V., et al. Massively multiplex single-cell Hi-C. Nature Methods. 14 (3), 263-266 (2017).
- Meng, L., Wang, C., Shi, Y., Luo, Q. Si-C is a method for inferring super-resolution intact genome structure from single-cell Hi-C data. Nature Communications. 12 (1), 4369 (2021).
- Tavares-Cadete, F., Norouzi, D., Dekker, B., Liu, Y., Dekker, J. Multi-contact 3C reveals that the human genome during interphase is largely not entangled. Nature Structural & Molecular Biology. 27 (12), 1105-1114 (2020).
- Vermeulen, C., et al. Multi-contact 4C: long-molecule sequencing of complex proximity ligation products to uncover local cooperative and competitive chromatin topologies. Nature Protocols. 15 (2), 364-397 (2020).
- Oomen, M. E., Hedger, A. K., Watts, J. K., Dekker, J. Detecting chromatin interactions between and along sister chromatids with SisterC. Nature Methods. 17 (10), 1002-1009 (2020).
- Mitter, M., et al. Conformation of sister chromatids in the replicated human genome. Nature. 586 (7827), 139-144 (2020).
- Krietenstein, N., et al. Ultrastructural Details of Mammalian Chromosome Architecture. Molecular Cell. 78 (3), 554-565 (2020).
- Hughes, J. R., et al. Analysis of hundreds of cis-regulatory landscapes at high resolution in a single, high-throughput experiment. Nature Genetics. 46 (2), 205-212 (2014).
- Fullwood, M. J., et al. An oestrogen-receptor-alpha-bound human chromatin interactome. Nature. 462 (7269), 58-64 (2009).
- Fang, R., et al. Mapping of long-range chromatin interactions by proximity ligation-assisted ChIP-seq. Cell Research. 26 (12), 1345-1348 (2016).
- Mumbach, M. R., et al. HiChIP: efficient and sensitive analysis of protein-directed genome architecture. Nature Methods. 13 (11), 919-922 (2016).

