Захват конформации хромосом по длинным шкалам
Summary
Hi-C 3.0 представляет собой улучшенный протокол Hi-C, который сочетает в себе формальдегидные и дисукцинимидиллутаратные сшивки с коктейлем ферментов dpnII и DdeI для увеличения отношения сигнал/шум и разрешения обнаружения взаимодействия хроматина.
Abstract
Захват конформации хромосом (3C) используется для обнаружения трехмерных взаимодействий хроматина. Как правило, химическое сшивание с формальдегидом (ФА) используется для фиксации взаимодействий хроматина. Затем переваривание хроматина ферментом рестрикции и последующее религирование концов фрагмента превращает трехмерную (3D) близость в уникальные продукты лигирования. Наконец, после разворота сшивок, удаления белка и выделения ДНК ДНК срезается и подготавливается к высокопроизводительному секвенированию. Частота бесконтактного лигирования пар локусов является мерой частоты их колокализации в трехмерном пространстве в клеточной популяции.
Секвенированная библиотека Hi-C предоставляет общегеномную информацию о частотах взаимодействия между всеми парами локусов. Разрешение и точность Hi-C зависит от эффективного сшивания, которое поддерживает контакты хроматина и частую и равномерную фрагментацию хроматина. В этой статье описывается улучшенный in situ протокол Hi-C, Hi-C 3.0, который повышает эффективность сшивки путем объединения двух сшивающих (формальдегид [FA] и дисукцинимидиллутарат [DSG]), с последующим более тонким пищеварением с использованием двух ферментов рестрикции (DpnII и DdeI). Hi-C 3.0 – это единый протокол для точной количественной оценки признаков складывания генома в меньших масштабах, таких как петли и топологически ассоциирующие домены (TADs), а также особенностей в более крупных масштабах ядра, таких как компартменты.
Introduction
Захват конформации хромосом используется с 2002 года1. По сути, каждый вариант захвата конформации зависит от фиксации взаимодействий ДНК-белок и белок-белок для сохранения 3D-организации хроматина. За этим следует фрагментация ДНК, обычно путем рестрикции пищеварения, и, наконец, религирование близлежащих концов ДНК для преобразования пространственно проксимальных локусов в уникальные ковалентные последовательности ДНК. Первоначальные протоколы 3C использовали ПЦР для выборки конкретных взаимодействий «один к одному». Последующие анализы 4C позволили обнаружить взаимодействия «один ко всем»2, в то время как 5C обнаружил взаимодействия «многие ко многим»3. Захват конформации хромосом полностью осуществился после внедрения высокопроизводительного секвенирования следующего поколения (NGS), которое позволило обнаружить геномные взаимодействия «все ко всем» с использованием геномных Hi-C4 и сопоставимых методов, таких как 3C-seq5, TCC6 и Micro-C 7,8 (см. также обзор Denker и De Laat9).
В Hi-C биотинилированные нуклеотиды используются для обозначения 5′-свесов после пищеварения и перед лигированием (рисунок 1). Это позволяет выбирать правильно переваренные и религированные фрагменты с использованием бусин, покрытых стрептавидином, что отличает их от GCC10. Важное обновление протокола Hi-C было реализовано Rao et al.11, которые выполнили переваривание и религирование в интактных ядрах (т.е. in situ) для уменьшения ложных продуктов лигирования. Кроме того, замена пищеварения HindIII перевариванием MboI (или DpnII) уменьшала размер фрагмента и увеличивала потенциал разрешения Hi-C. Это увеличение позволило обнаружить относительно небольшие структуры и более точную геномную локализацию точек контакта, таких как петли ДНК между мелкими цис-элементами, например, петли между CTCF-связанными сайтами, генерируемыми экструзией петли11,12. Однако этот потенциал имеет свою цену. Во-первых, двукратное увеличение разрешения требует четырехкратного (22) увеличения последовательности чтения13. Во-вторых, небольшие размеры фрагментов увеличивают возможность ошибочного принятия непереваренных соседних фрагментов за переваренные и лигированные фрагменты14. Как уже упоминалось, в Hi-C переваренные и религированные фрагменты отличаются от непереваренных фрагментов наличием биотина в лигативном соединении. Тем не менее, требуется правильное удаление биотина с нелигированных концов, чтобы гарантировать, что только лигационные соединения вытягиваются вниз14,15.
С уменьшением стоимости NGS становится возможным более детально изучить сворачивание хромосом. Чтобы уменьшить размер фрагментов ДНК и тем самым увеличить разрешение, протокол Hi-C может быть адаптирован для более частого использования редукционных ферментов16 или для использования комбинаций ферментоврестрикции 17,18,19. Альтернативно,MNase 7,8 в Micro-C и DNase в DNase Hi-C20 могут быть титрованы для достижения оптимального пищеварения.
Недавняя систематическая оценка основ методов 3C показала, что обнаружение особенностей сворачивания хромосом на каждой шкале длины значительно улучшилось благодаря последовательному сшиванию с 1% FA, за которым следует 3 мМ DSG17. Кроме того, Hi-C с пищеварением HindIII был лучшим вариантом для обнаружения крупномасштабных складных функций, таких как компартменты, и что Micro-C превосходил в обнаружении мелкомасштабных складных функций, таких как петли ДНК. Эти результаты привели к разработке единой стратегии высокого разрешения «Hi-C 3.0», которая использует комбинацию сшивающих устройств FA и DSG с последующим двойным перевариванием с эндонуклеазами DpnII и DdeI21. Hi-C 3.0 обеспечивает эффективную стратегию для общего использования, поскольку он точно обнаруживает складные элементы на всех масштабах длины17. Экспериментальная часть протокола Hi-C 3.0 подробно описана здесь и показаны типичные результаты, которые можно ожидать после секвенирования.
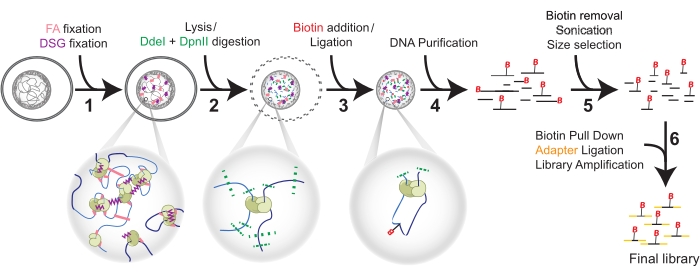
Рисунок 1: Процедура Hi-C в шесть этапов. Клетки фиксируются сначала ФА, а затем DSG (1). Затем лизис предшествует двойному пищеварению с DdeI и DpnII (2). Биотин добавляют путем заполнения свеса, а проксимальные притупленные концы перевязывают (3) перед очисткой ДНК (4). Биотин удаляется с нелигированных концов перед обработкой ультразвуком и подбором размера (5). Наконец, вытягивание биотина позволяет осуществлять лигирование адаптера и библиотечное усиление с помощью ПЦР (6). Сокращения: FA = формальдегид; DSG = дисукцинимидиллутарат; B = Биотин. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Protocol
Representative Results
Discussion
Критические шаги для обработки ячеек
Хотя можно использовать меньшее количество входных ячеек, этот протокол был оптимизирован для ~ 5 × 106 ячеек на линию секвенирования (~ 400 M чтения), чтобы обеспечить надлежащую сложность после глубокого секвенирования. Клетки лучше всего подсчитывать перед фиксацией. Для генерации сверхглубоких библиотек мы обычно умножаем количество полос (и ячеек) до тех пор, пока не будет достигнута желаемая глубина чтения. Для оптимальной фиксации сывороточную среду следует заменить ПБС до фиксации ФА, а фиксирующие растворы добавлять немедленно и без градиентов концентрации 15,22. Для сбора клеток соскоб предпочтительнее трипсинизации, потому что переход от более плоской к сферической форме после трипсинизации может повлиять на ядерную конформацию. После добавления DSG гранулы сыпучих и комковатых клеток легко теряются. Будьте осторожны при работе с клетками на этом этапе и добавьте до 0,05% BSA, чтобы уменьшить слипание.
Изменения в методе
Этот протокол был разработан с использованием клеток человека17. Тем не менее, основываясь на опыте захвата конформации хромосом, этот протокол должен работать для большинства эукариотических клеток. Для значительно более низкого ввода (~1 × 106 ячеек) мы советуем использовать половину объемов для процедур лизиса и конформационного захвата [этапы 2.1-2.4]. Это также позволит выполнить изоляцию ДНК [этап 2.5] в настольной центрифуге с пробирками объемом 1,7 мл, что может улучшить гранулирование для низких концентраций ДНК. Количественная оценка ДНК (этап 2.6.6) укажет, как действовать дальше. Для небольших количеств выделенной ДНК (1-5 мкг) мы предлагаем пропустить выбор размера (этап 3.3) и приступить к удалению биотина после уменьшения объема со 130 мкл до ~45 мкл с помощью КОЕ.
Этот протокол был разработан специально для обеспечения высокого качества данных после последующего сшивания с FA и DSG и пищеварения с DpnII и DdeI. Однако альтернативные стратегии сшивания, такие как FA, за которым следует EGS (этиленгликоль-бис (сукцинимидилсукцинат)), который также используется в ChIP-seq23 и ChIA-PET24, могут работать одинаково хорошо17. Аналогичным образом, различные комбинации ферментов, такие как DpnII и HinfI18 или MboI, MseI и NlaIII19 , могут быть использованы для пищеварения. При адаптации комбинаций ферментов обязательно используйте биотинилированные нуклеотиды, которые могут заполнять определенные 5′-свесы и использовать наиболее оптимальные буферы для каждого коктейля. DpnII поставляется с собственным буфером, и производитель ферментов рекомендует специальный буфер для пищеварения DdeI. Тем не менее, для двойного пищеварения с DpnII и DdeI в этом протоколе рекомендуется рестрикционный буфер, потому что он рассчитан на 100% активность для обоих ферментов.
Поиск и устранение неисправностей при захвате соответствия
Три ключевых шага в захвате конформации хромосом: сшивание, пищеварение и религирование были выполнены до того, как результаты могут быть визуализированы на геле. Чтобы определить качество каждого из этих трех этапов и определить, где могли возникнуть проблемы, аликвоты до (CI) и после пищеварения (DC) берутся и загружаются на гель вместе с лигированным образцом Hi-C (рисунок 2). Этот гель используется для определения качества образца Hi-C и того, стоит ли продолжать протокол. Без CI и DC трудно точно определить потенциальные неоптимальные шаги. Стоит отметить, что неоптимальная перевязка может быть вызвана проблемой в самой лигации, заполнении или проблемой с сшивки. Для устранения неполадок сшивки убедитесь, что вы не используете более 1 × 107 ячеек на библиотеку и начните со свежих реагентов для сшивания и чистых ячеек (т. Е. Промытых PBS). Для перевязки убедитесь, что клетки и лигационная смесь хранятся на льду. Добавьте Т4 ДНК-лигазу непосредственно перед 4-часовой инкубацией при 16 °C и хорошо перемешайте.
Устранение неполадок при подготовке библиотеки
Если требуется более 10 циклов ПЦР или продукт ПЦР не виден на геле после титрования ПЦР (рисунок 4), есть несколько вариантов сохранения образца Hi-C. Возвращаясь к титрованию ПЦР, первый вариант заключается в том, чтобы попробовать ПЦР еще раз. Если продукта все еще недостаточно, можно попробовать еще один раунд перевязки A-хвоста и адаптера (шаг 3.6) после промывки бусин дважды с буфером 1x TLE. После этого дополнительного А-хвоста и перевязки адаптера можно приступать к титрованию ПЦР, как и раньше. Если продукта по-прежнему нет, последний вариант состоит в том, чтобы повторно обработать фракцию 0,8x с шага 3.3 и продолжить оттуда.
Ограничения и преимущества Hi-C3.0
Важно понимать, что Hi-C является популяционным методом, который фиксирует среднюю частоту взаимодействий между парами локусов в клеточной популяции. Некоторые вычислительные анализы предназначены для разделения комбинаций конформаций из популяции25, но в принципе Hi-C слеп к различиям между клетками. Хотя можно выполнить одноэлементный Hi-C26,27 и вычислительные выводы могут быть сделаны28, одноэлементный Hi-C не подходит для получения информации 3C сверхвысокого разрешения. Дополнительным ограничением Hi-C является то, что он обнаруживает только парные взаимодействия. Для обнаружения многоконтактных взаимодействий можно либо использовать частые фрезы в сочетании с секвенированием короткого считывания (Illumina)16, либо выполнять многоконтактные 3C29 или 4C30, используя секвенирование с длинным чтением с платформ PacBio или Oxford Nanopore. Производные Hi-C для конкретного обнаружения контактов между и вдоль сестринских хроматид также были разработаны31,32.
Хотя Hi-C19 и Micro-C33 могут использоваться для создания карт контактов с субкилобазовыми разрешениями, оба требуют большого количества последовательных считываний, и это может стать дорогостоящим мероприятием. Чтобы получить аналогичное или даже более высокое разрешение без затрат, может быть применено обогащение для конкретных геномных областей (захват-C34) или специфических белковых взаимодействий (ChiA-PET35, PLAC-seq36, Hi-ChIP37). Сила и недостаток этих применений обогащения заключается в том, что отбирается только ограниченное количество взаимодействий. При таких обогащениях теряется глобальный аспект Hi-C (и возможность глобальной нормализации).
Важность и потенциальные возможности применения Hi-C3.0
Этот протокол был разработан для обеспечения сверхглубокого 3C с высоким разрешением при одновременном обнаружении крупномасштабных складных функций, таких как TAD и отсеки17 (рисунок 6). Этот протокол начинается с 5 × 106 ячеек на трубку для каждой библиотеки Hi-C, что должно быть более чем достаточным материалом для секвенирования одной или двух полос на проточной ячейке для получения до 1 миллиарда парных считываний. Для сверхглубокого секвенирования следует подготовить несколько пробирок по 5 × 106 клеток, в зависимости от количества отображаемых считываний и дубликатов ПЦР. При самом высоком разрешении (<1 кб) зацикленные взаимодействия в основном обнаруживаются между сайтами CTCF, но также могут быть обнаружены взаимодействия промотор-энхансер. Читатели могут обратиться к Akgol Oksuz et al.17 для подробного описания анализа данных.
Disclosures
The authors have nothing to disclose.
Acknowledgements
Мы хотели бы поблагодарить Дениса Лафонтена за разработку протокола и Сергея Венева за биоинформационную помощь. Эта работа была поддержана грантом Национального института здравоохранения Common Fund 4D Nucleome Program для J.D. (U54-DK107980, UM1-HG011536). Джей Ди является исследователем Медицинского института Говарда Хьюза.
Эта статья подпадает под действие политики открытого доступа к публикациям HHMI. Руководители лабораторий HHMI ранее предоставили неисключительную лицензию CC BY 4.0 для общественности и сублицензируемую лицензию HHMI в своих исследовательских статьях. В соответствии с этими лицензиями принятая автором рукопись этой статьи может быть предоставлена в свободный доступ по лицензии CC BY 4.0 сразу после публикации.
Materials
| 1 kb Ladder | New England Biolabs | N3232L | |
| Agarose | Invitrogen | 16500100 | |
| Agencourt AMPure XP magnetic beads , 60 mL | Beckman Coulter | A63881 | |
| Amicon Ultra-0.5 Centrifugal Filter Unit (CFU) | EMD Millipore | UFC500396 | |
| Annealing Buffer (5x) | See recipe in supplemental materials | ||
| ATP 10 mM | ThermoFisher | R0441 | |
| Avanti J-25i High Speed Refrigerated ultra-centrifuge | Beckman Coulter | ||
| beckman ultracentrifuge tube 35 mL | Beckman Coulter | 357002 | |
| Binding Buffer (2x) | See recipe in supplemental materials | ||
| biotin-14-dATP 0.4 mM | Invitrogen | 19524-016 | |
| BSA 10 mg/mL | New England Biolabs | B9000S | dilute from 20 mg/mL |
| Cell scraper | Falcon | 353089 | |
| Cell scraper | Corning | 3008 | |
| Conical polypropylene tubes 50 mL | Denville | C1062-P | |
| Conical tube 15 mL | Denville | C1017-P | |
| Covaris micro tube AFA fiber with snap-cap 130 µL | Covaris | 520045/520077 | |
| Covaris Sonicator | Covaris | E220/E220evolution/M220 | |
| Culture flask 175 cm2 | Falcon | 353112 | |
| Culture plates 150 mm x 25 mm | Corning | 430599 | |
| dATP 1 mM | Invitrogen | 56172 | |
| dATP 10 mM | Invitrogen | 56172 | |
| dCTP 10 mM | Invitrogen | 56173 | |
| DdeI | New England Biolabs | R0175L | |
| dGTP 10 mM | Invitrogen | 56174 | |
| DMSO | Sigma | D2650-5x10ML | |
| dNTP mix 25 mM | Invitrogen | 10297117 | |
| Dounce homogenizer | DWK Life Sciences | 8853010002/8853030002 | |
| DPBS | Gibco | 14190-144 | |
| DpnII | New England Biolabs | R0543M | |
| DSG | ThermoScientific | 20593 | |
| dTTP 10 mM | Invitrogen | 56175 | |
| Ethanol 70% | Fisher | A409-4 | Diluted from 100% |
| Ethidium Bromide | Fisher | BP1302-10 | |
| Formaldehyde (37%) | Fisher | BP531-500 | |
| Gel loading dye (6x ) | New England Biolabs | B7024S | |
| Glycine in ultrapure water 2.5 M | Sigma | G8898-1KG | |
| HBSS | Gibco | 14025-092 | |
| Igepal CA-630 detergent | MP Biomedicals | 198596 | |
| Klenow DNA polymerase 5 U/µL | New England Biolabs | M0210L | |
| Klenow Fragment 3–>5’ exo-, 5 U/µL | New England Biolabs | M0212L | |
| ligation buffer (10x) | New England Biolabs | B7203S | |
| Liquid nitrogen | |||
| LoBind microcentrifuge tube 1.7 mL | Eppendorf | 22431021 | |
| Low Molecular Weight DNA Ladder | New England Biolabs | N3233L | |
| Lysis buffer | See recipe in supplemental materials | ||
| Magnetic Particle separator | ThermoFisher | 12321D | |
| Microfuge tubes 1.7 mL | Axygen | MCT-175-C | |
| MyOne Streptavidin C1 beads | Invitrogen | 65001 | |
| NEBuffer 2.1 (10x) | New England Biolabs | B7002S | |
| NEBuffer 3.1 (10x) | New England Biolabs | B7203S | |
| PBS | Gibco | 70013-032 | |
| PCR (strip) tubes | Biorad | TBS0201/ TCS0803 | |
| PCR thermocycler | Biorad | T100 | |
| Pfu Ultra II Buffer (10x) | Agilent | Comes with Pfu Ultra | |
| PfuUltra II Fusion HS DNA Polymerase | Agilent | 600674 | |
| Phase lock tube 15 mL | Qiagen | 129065 | |
| Phase lock tubes 2 mL | Qiagen | 129056 | |
| Phenol:chloroform:isoamyl alcohol | Invitrogen | 15593-049 | |
| Protease inhibitor cocktail | ThermoFisher | 78440 | |
| Proteinase K in ultrapure water 10 mg/mL | Invitrogen | 25530-031 | |
| Refrigerated Centrifuge | Eppendorf | 5810R | |
| RNase A, DNase and protease-free 10 mg/mL | Thermo Scientific | EN0531 | |
| Rotator | Argos technologies | EW-04397-40 or rocking platform | |
| SDS 1% | Fisher | BP13111 | |
| Sodium acetate pH = 5.2, 3 M | Sigma | ||
| Sub-Cell GT Horizontal Electrophoresis System | Biorad | 1704401 | |
| T4 DNA ligase 1 U/µL | Invitrogen | 100004817 | |
| T4 DNA polymerase | New England Biolabs | M0203L | |
| T4 DNA polymerase | New England Biolabs | M0203L | |
| T4 DNA polymerase 3 U/µL | New England Biolabs | M0203L | |
| T4 ligation buffer (5x) | Invitrogen | Y90001 | |
| T4 polynucleotide kinase 10 U/µL | New England Biolabs | M0201L | |
| Tabletop centrifuge | Eppendorf | 5425 | |
| TBE buffer | See recipe in supplemental materials | ||
| Tris Low EDTA Buffer (TLE) | See recipe in supplemental materials | ||
| Triton X-100 (10%) | Sigma | 93443 | |
| Truseq adapter oligos | Integrated DNA Technologies (IDT)) | https://www.idtdna.com/site/order/oligoentry | 250 nmole and HPLC purified |
| Tween 20 detergent | Fisher | 9005-64-5 | |
| Tween Wash Buffer | See recipe in supplemental materials | ||
| Vortex | Scientific Industries | (G560)SI-0236 |
References
- Dekker, J., Rippe, K., Dekker, M., Kleckner, N. Capturing chromosome conformation. Science. 295 (5558), 1306-1311 (2002).
- Simonis, M., et al. Nuclear organization of active and inactive chromatin domains uncovered by chromosome conformation capture-on-chip (4C). Nature Genetics. 38 (11), 1348-1354 (2006).
- Dostie, J., et al. Chromosome Conformation Capture Carbon Copy (5C): a massively parallel solution for mapping interactions between genomic elements. Genome Research. 16 (10), 1299-1309 (2006).
- Lieberman-Aiden, E., et al. Comprehensive mapping of long-range interactions reveals folding principles of the human genome. Science. 326 (5950), 289-293 (2009).
- Stadhouders, R., et al. Multiplexed chromosome conformation capture sequencing for rapid genome-scale high-resolution detection of long-range chromatin interactions. Nature Protocols. 8 (3), 509-524 (2013).
- Kalhor, R., Tjong, H., Jayathilaka, N., Alber, F., Chen, L. Genome architectures revealed by tethered chromosome conformation capture and population-based modeling. Nature Biotechnology. 30 (1), 90-98 (2012).
- Hsieh, T. H., et al. Mapping nucleosome resolution chromosome folding in yeast by micro-C. Cell. 162 (1), 108-119 (2015).
- Hsieh, T. -. H. S., Fudenberg, G., Goloborodko, A., Rando, O. J. Micro-C XL: assaying chromosome conformation from the nucleosome to the entire genome. Nature Methods. 13 (12), 1009-1011 (2016).
- Denker, A., de Laat, W. The second decade of 3C technologies: detailed insights into nuclear organization. Genes & Development. 30 (12), 1357-1382 (2016).
- Rodley, C. D., Bertels, F., Jones, B., O’Sullivan, J. M. Global identification of yeast chromosome interactions using Genome conformation capture. Fungal Genetics and Biology. 46 (11), 879-886 (2009).
- Rao, S. S., et al. A 3D map of the human genome at kilobase resolution reveals principles of chromatin looping. Cell. 159 (7), 1665-1680 (2014).
- Alipour, E., Marko, J. F. Self-organization of domain structures by DNA-loop-extruding enzymes. Nucleic Acids Research. 40 (22), 11202-11212 (2012).
- Lajoie, B. R., Dekker, J., Kaplan, N. The Hitchhiker’s guide to Hi-C analysis: Practical guidelines. Methods. 72, 65-75 (2015).
- Belaghzal, H., Dekker, J., Gibcus, J. H. Hi-C 2.0: An optimized Hi-C procedure for high-resolution genome-wide mapping of chromosome conformation. Methods. 123, 56-65 (2017).
- Golloshi, R., Sanders, J. T., McCord, R. P. Iteratively improving Hi-C experiments one step at a time. Methods. 142, 47-58 (2018).
- Darrow, E. M., et al. Deletion of DXZ4 on the human inactive X chromosome alters higher-order genome architecture. Proceedings of the National Academy of Sciences of the United States of America. 113 (31), 4504-4512 (2016).
- Akgol Oksuz, B., et al. Systematic evaluation of chromosome conformation capture assays. Nature Methods. 18 (9), 1046-1055 (2021).
- Ghuryeid, J., et al. Integrating Hi-C links with assembly graphs for chromosome-scale assembly. PLoS Computational Biology. 15 (8), 1007273 (2019).
- Gu, H., et al. Fine-mapping of nuclear compartments using ultra-deep Hi-C shows that active promoter and enhancer elements localize in the active A compartment even when adjacent sequences do not. bioRxiv. , (2021).
- Ramani, V., et al. Mapping 3D genome architecture through in situ DNase Hi-C. Nature Protocols. 11 (11), 2104-2121 (2016).
- Lafontaine, D. L., Yang, L., Dekker, J., Gibcus, J. H. Hi-C 3.0: Improved Protocol for Genome-Wide Chromosome Conformation Capture. Current Protocols. 1 (7), 198 (2021).
- Belton, J. -. M. M., et al. Hi-C: A comprehensive technique to capture the conformation of genomes. Methods. 58 (3), 268-276 (2012).
- Truch, J., Telenius, J., Higgs, D. R., Gibbons, R. J. How to tackle challenging ChIP-Seq, with long-range cross-linking, Using ATRX as an example. Methods in Molecular Biology. 1832, 105-130 (2018).
- Wang, P., et al. In situ chromatin interaction analysis using paired-end tag sequencing. Current Protocols. 1 (8), 174 (2021).
- Tjong, H., et al. Population-based 3D genome structure analysis reveals driving forces in spatial genome organization. Proceedings of the National Academy of Sciences of the United States of America. 113 (12), 1663-1672 (2016).
- Nagano, T., et al. Cell-cycle dynamics of chromosomal organization at single-cell resolution. Nature. 547 (7661), 61-67 (2017).
- Ramani, V., et al. Massively multiplex single-cell Hi-C. Nature Methods. 14 (3), 263-266 (2017).
- Meng, L., Wang, C., Shi, Y., Luo, Q. Si-C is a method for inferring super-resolution intact genome structure from single-cell Hi-C data. Nature Communications. 12 (1), 4369 (2021).
- Tavares-Cadete, F., Norouzi, D., Dekker, B., Liu, Y., Dekker, J. Multi-contact 3C reveals that the human genome during interphase is largely not entangled. Nature Structural & Molecular Biology. 27 (12), 1105-1114 (2020).
- Vermeulen, C., et al. Multi-contact 4C: long-molecule sequencing of complex proximity ligation products to uncover local cooperative and competitive chromatin topologies. Nature Protocols. 15 (2), 364-397 (2020).
- Oomen, M. E., Hedger, A. K., Watts, J. K., Dekker, J. Detecting chromatin interactions between and along sister chromatids with SisterC. Nature Methods. 17 (10), 1002-1009 (2020).
- Mitter, M., et al. Conformation of sister chromatids in the replicated human genome. Nature. 586 (7827), 139-144 (2020).
- Krietenstein, N., et al. Ultrastructural Details of Mammalian Chromosome Architecture. Molecular Cell. 78 (3), 554-565 (2020).
- Hughes, J. R., et al. Analysis of hundreds of cis-regulatory landscapes at high resolution in a single, high-throughput experiment. Nature Genetics. 46 (2), 205-212 (2014).
- Fullwood, M. J., et al. An oestrogen-receptor-alpha-bound human chromatin interactome. Nature. 462 (7269), 58-64 (2009).
- Fang, R., et al. Mapping of long-range chromatin interactions by proximity ligation-assisted ChIP-seq. Cell Research. 26 (12), 1345-1348 (2016).
- Mumbach, M. R., et al. HiChIP: efficient and sensitive analysis of protein-directed genome architecture. Nature Methods. 13 (11), 919-922 (2016).

