התגבשות חלבון בתפוקה גבוהה באמצעות מיקרודיאליזה
Summary
הפרוטוקול המוצג מתאר גישה פשוטה לסינון תנאי התגבשות חלבונים וגדילת גבישים באמצעות צלחת דיאליזה בעלת תפוקה גבוהה של 96 בארות. השימוש בשפופרות דיאלייזר לגידול בקנה מידה גדול של מיקרו-גבישים מודגם גם עבור קריסטלוגרפיה טורית ויישומי MicroED.
Abstract
הבנת יחסי מבנה-פונקציה של מקרומולקולות, כגון חלבונים, ברמה המולקולרית חיונית לביו-רפואה ולגילוי תרופות מודרניות. עד כה, קריסטלוגרפיה בקרני רנטגן נותרה השיטה המוצלחת ביותר לפתרון מבנים חלבוניים תלת מימדיים ברזולוציה אטומית. עם ההתקדמות האחרונה בקריסטלוגרפיה טורית, בין אם באמצעות לייזר אלקטרונים ללא קרני רנטגן (XFELs) או מקורות אור סינכרוטרון, קריסטלוגרפיה של חלבונים התקדמה לחזית הבאה, שם היכולת להשיג נתונים שנפתרו בזמן מספקת תובנות מכניסטיות חשובות על התנהגותן של מולקולות ביולוגיות בטמפרטורת החדר. פרוטוקול זה מתאר תהליך עבודה פשוט בתפוקה גבוהה (HTP) לסינון תנאי התגבשות באמצעות שימוש בלוח דיאליזה של 96 בארות. לוחות אלה עומדים בתקן האגודה לסינון ביומולקולרי (SBS) וניתן להתקין אותם בקלות באמצעות כל מעבדת התגבשות סטנדרטית. לאחר זיהוי התנאים האופטימליים, ניתן לייצר כמויות גדולות של גבישים (מאות מיקרו-גבישים) באמצעות הדיאליזר. כדי לאמת את החוסן והרבגוניות של גישה זו, התגבשו ארבעה חלבונים שונים, כולל שני חלבוני ממברנה.
Introduction
במהלך המאה האחרונה, קריסטלוגרפיה של קרני רנטגן הייתה קריטית בהבהרה ובהבנה של פרדיגמת המבנה-פונקציה של מקרומולקולות ביולוגיות. נכון להיום, היא ממשיכה להיות אחת השיטות המוצלחות ביותר בהבהרת מבני רזולוציה אטומית של חלבונים רבים וייחודיים החיוניים להבנה הבסיסית של ביוכימיה של התא, רפואה, וגילוי מוקדם של תרופות 1,2. עם זאת, התגבשות חלבונים נותרה צוואר בקבוק בחקר מטרות חלבוניות רבות, במיוחד חלבוני ממברנה וקומפלקסים חלבוניים גדולים3. כתוצאה מכך, התגבשות חלבונים נחשבת כמעט תמיד לאמנות בשל גישות הניסוי והטעייה עתירות העבודה המופעלות 4,5,6.
בדרך כלל מוסיפים חומר מזרז לתמיסת חלבון בריכוז גבוה כדי ליצור סידור סריג מסודר, סדיר וחוזר של מולקולות חלבון, הידועות בשם גבישים. בתנאים נוחים, כגון טמפרטורה, pH, ריכוז וחומר מזרז, נוצרת בסופו של דבר תמיסה סופר-רוויה, ואחריה גרעין גבישיוצמיחה 7,8. למרות שהיו התפתחויות רבות במערכי ניסויי התגבשות, בעיקר עם הפיתוח של מערכות רובוטיות בעלות תפוקה גבוהה והזמינות של מסכי “מטריצה דלילה” מוכנים, הגישות הכלליות להתגבשות חלבונים נותרו במידה רבה ללא שינוי לאורך השנים. טכניקות ניסיוניות נפוצות להתגבשות חלבונים כוללות דיפוזיית אדים (טיפה תלויה וטיפה יושבת)9, מיקרו-אצווה (תחת שמן)10,11, דיפוזיה בממשק חופשי (התקנים מיקרופלואידים)12, ודיאליזה (באמצעות כפתורים וטכניקות אחרות)13,14,15. עם זאת, קיימים גם מערכים מיוחדים יותר, כגון גישות מזופאזות לגיבוש חלבוני ממברנה16,17. בעוד שרוב מבני החלבונים בקרני רנטגן שהופקדו בבנק נתוני החלבונים נפתרו עד כה באמצעות התגבשות בשיטות דיפוזיה של אדים 6,18, גישות אחרות, כגון התגבשות על ידי דיאליזה, נראות לא מנוצלות, ככל הנראה בשל ההיבטים המעשיים הקשורים למערך הניסוי שלהן.
התגבשות באמצעות דיאליזה פשוט מסתמכת על דיפוזיה איטית של מומסים (מזרזים, יונים, תוספים וחוצצים) דרך קרום חדיר למחצה המונע בו זמנית ממולקולות חלבון לזרום. בדרך זו, תמיסת החלבון מובאת לאט לאט לשיווי משקל, כאשר המשקע מגיע לריכוז הדרוש כדי להתגבש. הקינטיקה של המערכת תלויה בטמפרטורה, בריכוז המשקע ובחיתוך המשקל המולקולרי של קרום התאית (MWCO)19. עד כה, מערך ההתגבשות הפופולרי ביותר בדיאליזה היה באמצעות כפתורי מיקרודיאליזה העשויים מיריעות אקריליק שקופות. אלה שקועים בדרך כלל במאגרים (בעיקר באמצעות לוחות טיפה תלויים בדיפוזיה של אדים) המכילים את תמיסות ההתגבשות. אולם שיטה זו בעלת תפוקה נמוכה יותר דורשת גם הרכבה ספציפית כדי לאטום את תמיסת החלבון בתוך קרום הדיאליזה הממוקם מעל תא הכפתור, כפי שמודגם באיור 1. יתר על כן, בועות אוויר הכלואות בין קרום הדיאליזה לבין תמיסת החלבון הן בעיה שכיחה הפוגעת בצמיחת הגביש. אילוץ נוסף של השיטה הוא דרישות הדגימה, לפיהן יש צורך בריכוזים ונפחים גבוהים בהרבה בהשוואה לשיטות דיפוזיה של אדים, כדי להתאים לכפתורי הדיאליזה. לכן, התגבשות באמצעות לחצני מיקרודיאליזה נתפסה כשיטה לא מושכת, במיוחד עבור מטרות קשות כמו חלבוני ממברנה, שתפוקת הטיהור שלהם נמוכה באופן מתסכל. לאחרונה פותחו מכשירים מיקרופלואידים המאפשרים התגבשות חלבונים על ידי דיאליזה15. שבבים אלה תוכננו גם להיות בעלי שקיפות קרני רנטגן גבוהה עם רקע נמוך, מה שמאפשר להשתמש בשבבים לאיסוף נתונים באתרם בטמפרטורת החדר, ובכך לבטל את אי הנוחות של קציר וקירור קריסטלים בהקפאה. למרות התקדמות זו, הגישה היא עדיין תפוקה נמוכה מאוד ויקרה.
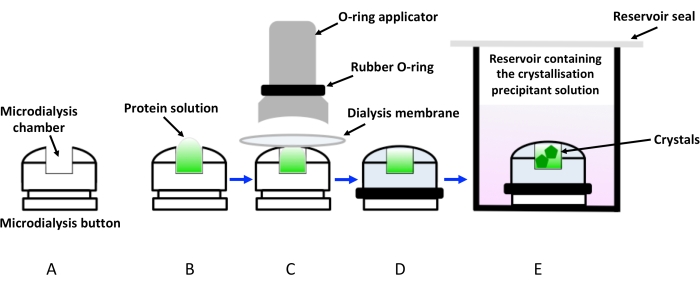
איור 1: ייצוג סכמטי של התגבשות באמצעות דיאליזה באמצעות לחצני דיאליזה. (A) ייצוג סכמטי של לחצן דיאליזה מתגבש. (B) תמיסת החלבון מתווספת לתא לחצן המיקרודיאליזה. (C) קרום הדיאליזה מוצמד ללחצן המיקרודיאליזה בעזרת טבעת גומי (O-ring) המופעלת באמצעות מוליך. (D) לחצן הדיאליזה מוכן לטבילה במאגר המכיל את תמיסת ההתגבשות (תמיסת דיאליזה), כמוצג ב-(E). יש לאטום את הבקבוקון המכיל את לחצן הדיאליזה השקוע כדי למנוע אידוי. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
כאן, מוצג פרוטוקול פשוט לבדיקת תנאי התגבשות חלבונים וגדילת גבישים באמצעות צלחת דיאליזה בעלת תפוקה גבוהה של 96 בארות. לוחות חד-פעמיים אלה מתוכננים לשימוש דומה ללוחות התגבשות דיפוזיה של אדים (פיפטה ואז חותם), כפי שמוצג באיור 2. הצלחות יכולות להכיל עד 3.2 מיקרוליטר חלבון ו-350 מיקרוליטר תמיסת דיאליזה. כל באר כוללת קרום תאית מתחדש נפרד למניעת זיהום צולב בין הבארות. ההתקנה אורכת כ-10 דקות ואינה דורשת ציוד מיוחד מלבד מה שניתן למצוא בכל מעבדות ההתגבשות הסטנדרטיות. ארבעה חלבונים שונים, כולל שני חלבוני ממברנה, משמשים כדי להדגים ולאמת גישה זו כשיטה יעילה לקריסטלוגרפיה של חלבונים בעלי תפוקה גבוהה (HTP).
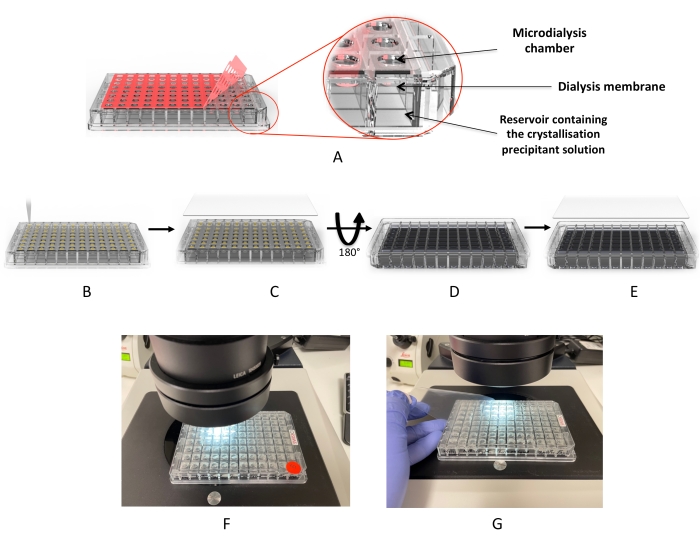
איור 2: תהליך התגבשות באמצעות לוח מיקרודיאליזה . (A) הסרת “סרט הכיסוי” של הדבק האדום. (B) ניפוק טיפות החלבון לכל אחת מבארות הטיפה. (C) הבארות מכוסות ב”סרט כיסוי” UV. (D) הצלחת הפוכה כדי להוסיף את תמיסות הדיאליזה (או מסך ההתגבשות). (ה) הצלחת אטומה ומודגרת. (ו,ז) בדיקת מיקרוסקופ של הטיפות. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
השימוש בהתגבשות זו באמצעות פרוטוקול דיאליזה הודגם באמצעות שפופרת דיאלייזר בנפח 0.5 מ”ל (איור 3) לייצור בקנה מידה גדול (מאות עד אלפים) של מיקרו-גבישים, המתאים לשיטות איסוף נתונים מתקדמות כגון קריסטלוגרפיה טורית בשני מתקני XFEL 20,21,22,23,24 וסינכרון25,26,27 כמו גם עבור גישות MicroED28,29,30.,
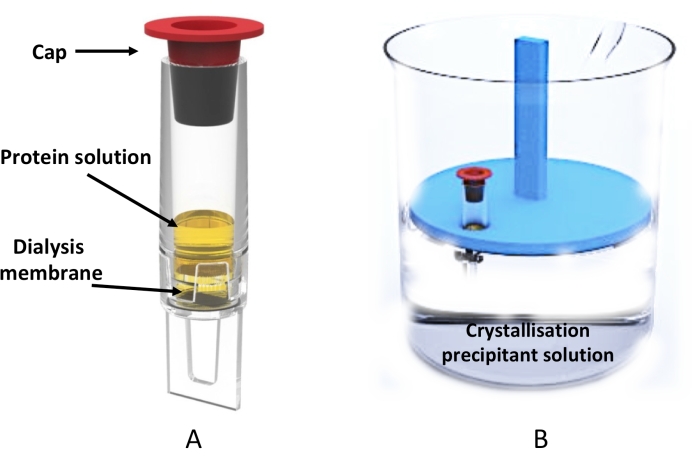
איור 3: התגבשות מיקרודיאליזה בקנה מידה גדול באמצעות צינור הדיאליזר . (A) ייצוג סכמטי של צינור הניב 0.5 מ”ל. (B) מבט מהצד על המכילה את תמיסת ההתגבשות ועל מתלה הצינור הצף המחזיק צינור דיאלייזר. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
Protocol
Representative Results
Discussion
כיום, התגבשות באמצעות דיאליזה היא שיטת ההתגבשות הפחות מנוצלת, כלומר בשל התפוקה הנמוכה והמייגעת של הגישות הקיימות, כגון מיקרודיאליזה עם כפתורים. כאן, פרוטוקול פשוט אך חזק עוקב אחר זרימת עבודה של HTP לגידול גבישי חלבונים באמצעות דיאליזה דרך צלחת מיקרודיאליזה וצינור דיאליזר הזמינים מסחרית. בהתאם לחלבון היעד, צלחת המיקרודיאליזה וצינור הדיאליזר המשמשים בהליך מגיעים עם בחירה של קרום דיאליזה עם MWCO 3, 5, 10 או 30 kDa MWCO. ניתן להגדיר את הפרוטוקול בקלות בכל מתקן התגבשות סטנדרטי ויש לו את היתרון הגדול בכך שהוא ישים הן לחלבונים מסיסים והן לחלבונים ממברנה. עם זאת, קומפלקסים של חלבון-חלבון וחלבון-חומצות גרעין לא נבדקו במהלך פרוטוקול זה.
כמו בכל גישת התגבשות בדיאליזה, יחס הנפח בין דגימת החלבון לתמיסת הדיאליזה הוא קריטי. בפרוטוקול זה מומלץ יחס של 1:100 בין הדגימה לתמיסת הדיאליזה, אך מכיוון שפלטת המיקרודיאליזה מאפשרת קיבולת מקסימלית של 350 מיקרוליטר של תמיסת דיאליזה, ניתן לחקור יחסים אלה כדי לקבל פגיעות גבישיות. נפח של 1-2 μL של חלבון משמש בפרוטוקול המוצג בעת הגדרת לוחות התגבשות. זאת על מנת להבטיח כי טיפות להגדיר במדויק עם פיפטה רב ערוצית ידנית. על ידי שימוש בפיפטות אלקטרוניות (פיפטות ניפוק רב ערוציות או חוזרות) או רובוטים לניפוק נוזל HTP, ניתן להשיג טיפות בנפחים נמוכים יותר, ובכך להפחית את כמות החלבון הנדרשת. יתר על כן, בשל הנפחים הנמוכים יחסית של חיץ הדיאליזה הנדרש על ידי לוחית המיקרודיאליזה (בניגוד לשיטות דיאליזה קונבנציונליות אחרות), ניתן (ללא שימוש נרחב במשאבים) לחקור חללים כימיים גדולים, לא רק באמצעות מסכי התגבשות הזמינים מסחרית אלא גם באמצעות מסכי אופטימיזציה (שתוכננו סביב מצב פגיעת ההתגבשות הראשונית).
שלב קריטי בהליך HTP המוצג הוא יישום בזמן של כמויות חלבון קטנות (0.50-3.2 μL לכל מצב) על צלחת הדיאליזה כדי להגביל התייבשות ואובדן דגימה. ניתן להקל על כך בקלות באמצעות פיפטה רב ערוצית, פיפטה חוזרת או מערכת התגבשות רובוטית. זמן הדגירה הארוך, כגון יותר משבועיים, של הצלחות ב -20 מעלות צלזיוס עלול להוביל להתייבשות טיפות החלבון או נזק לגבישים שנוצרו לאחרונה. שמירה על לוחיות הדיאליזה בתוך תא לחות או שקית אטומה יכולה להקל על השפעה זו. בנוסף, מומלץ להשתמש בחומרים סטריליים וטכניקות כדי למנוע צמיחת חיידקים.
כפי שהוזכר במבוא, לאחרונה, עם הצורך הגובר להבין דינמיקה מבנית של חלבונים עבור מנגנוני מחלה, אינטראקציות קישור חלבון-ליגנד ואינטראקציות חלבון-חלבון, עבר תחום הקריסטלוגרפיה של קרני רנטגן של חלבונים מהפכה באמצעות פיתוח טכניקות התגבשות חדשות וקיימות, גישות מודרניות להעברת דגימות גביש, דורות חדשים של מקורות קרני רנטגן, ושיטות מתוחכמות חדשות לרכישת נתונים ועיבודם36 ,37,38. לכן, הופעתה של מיקרו-קריסטלוגרפיה טורית בטמפרטורת החדר, המבוצעת באמצעות XFELs או מקורות אור סינכרוטרון, התפתחה ככלי יוצא דופן בביולוגיה מבנית, במיוחד בתחום חלבוני הממברנה39. עם זאת, אלפי מיקרו-גבישים נדרשים כדי לייצר מספיק נתונים לפתרון מבנה חזק, משימה שאינה קלה (לפחות בשיטות התגבשות קונבנציונליות). שיטת התגבשות הדיאליזה המתוארת כאן מאפשרת ייצור של מספר רב של מיקרו-גבישים. לאחר שתנאי ההתגבשות לייצור מיקרו-גבישים (1-10 מיקרומטר) נקבע באמצעות שימוש בלוח מיקרודיאליזה, ניתן לייצר כמויות גדולות של מיקרו-גבישים בצפיפות גבוהה באמצעות מכשיר דיאלייזר בנפח 0.5 מ”ל (איור 5). גבישים אלה מתאימים באופן אידיאלי לאיסוף נתונים באמצעות מטרות קבועות או מערכות העברת דגימות סילון נוזלי 27,40. גבישים המתקבלים בשיטה זו יכולים להתאים גם ליישומי MicroED. עם זאת, ייתכן שיהיה צורך לטחון אותם לגודל ועובי מתאימים ליישום ספציפי זה, מכיוון שאלקטרונים מתקשרים הרבה יותר חזק עם גבישים מאשר פוטונים של קרני רנטגן41.
לסיכום, גישת ההתגבשות באמצעות דיאליזה המתוארת כאן מוסיפה לאסטרטגיות המתפתחות בהתגבשות חלבונים לקביעת מבנה ומרחיבה את טווח המאמצים שניתן להשתמש בהם כדי לקבוע מטרות חלבוניות חדשות שלא הצליחו בעבר בשיטות קונבנציונליות אחרות.
Disclosures
The authors have nothing to disclose.
Acknowledgements
אנו מכירים במימון מהמחלקה לעסקים, אנרגיה ואסטרטגיה תעשייתית (BEIS) של בריטניה. אנו מודים לאלכס ר. ג’ונס ולמייק שו מהמעבדה הלאומית לפיזיקה על המשוב שלהם על כתב היד.
Materials
| 0.2 mL tubes | Thermo Scientific | AB0620 | For aliquoting protein solutions. |
| 0.2 µm syringe filter | Sartorius | 17823———-K | Surfactant-free cellulose acetate filters. For filtering dialysis solutions. |
| 0.22 µm membrane filters | Millipore | GSTF04700 | Membrane filters for filtering large volumes of buffers |
| 12-channel, variable 0.5 – 10 µL Research plus pipette | Eppendorf | 3125000028 | For dispensing protein drops onto the Diaplate. |
| 12-channel, variable 30 – 300 µL Eppendorf Research plus pipette | Eppendorf | 3125000060 | For dispensing dialysis solutions on the Diaplate reservoirs. |
| 20 mL syringe | Fisherbrand | 15889152 | For use with syringe filters. |
| 96 well 2.2 mL deep-well plates | Thermo Scientific | AB0788 | Polypropylene deep-well storage plates; for preparing screens using the Hamilton Microlab STARlet. |
| Centrifuge 5425 | Eppendorf | 5405000565 | With rotor FA-24×2 with a maximum g-force of 21,300 x g. |
| Diacon dialyser | SWISSCI | W72010 | Dialyzer tubes with a regenerated cellulose membrane with a molecular weight cut-off of 10 kDa. Ideal for protein solutions of up to 0.5 mL. |
| Diaplate 96-well plate | SWISSCI | W82010 | Microdialysis plate. The Diaplate consists of two sides with a regenerated cellulose membrane in-between with a molecular weight cut-off of 10 kDa. |
| Falcon 50 mL High Clarity PP Centrifuge Tube | Corning | 352070 | For holding dialysis solutions. |
| Floating rack | SWISSCI | n/a | Included in the Diacon kit |
| Floor-standing vibration-free incubator | Molecular Dimensions | MD5-01 | 400 L temperature-controlled incubator set to 20 °C. |
| Leica M205 C stereo microscope | Leica | Planapo 1.0x objective, 7.8x – 160x zoom range with DMC 4500 camera | |
| Lysozyme from chicken egg white | Sigma Aldrich | 62971 | Lyophilized protein |
| Memgold2 | Molecular Dimensions | MD1-64 | Sparse-matrix screen |
| Microlab STARlet | Hamilton | n/a | Liquid handler system. |
| Reservoir cover film | SWISSCI | n/a | Included in the Diaplate kit |
| Reusable bottle top filter | Thermo Scientific | DS0320-5045 | For fitering large volumes of buffers, for use with 0.22 µm membrane filters |
| Sealing paddle | SWISSCI | n/a | Included in the Diaplate kit |
| Thaumatin from Thaumatococcus daniellii | Sigma Aldrich | T7638 | Lyophilized protein |
| UV cover film | SWISSCI | n/a | Included in the Diaplate kit |
References
- Brooks-Bartlett, J. C., Garman, E. F. The nobel science: One hundred years of crystallography. Interdisciplinary Science Reviews. 40 (3), 244-264 (2015).
- Maveyraud, L., Mourey, L. Protein X-ray crystallography and drug discovery. Molecules. 25 (5), (2020).
- Kwan, T. O. C., Axford, D., Moraes, I. Membrane protein crystallography in the era of modern structural biology. Biochemical Society Transactions. 48 (6), 2505-2524 (2020).
- Birch, J., et al. The fine art of integral membrane protein crystallisation. Methods. 147, 150-162 (2018).
- Gorrec, F., Löwe, J. Automated protocols for macromolecular crystallization at the MRC laboratory of molecular biology. Journal of Visualized Experiments. 131 (131), (2018).
- Govada, L., Chayen, N. E. Choosing the method of crystallization to obtain optimal results. Crystals. 9 (2), 106 (2019).
- Chayen, N. E. Turning protein crystallisation from an art into science. Current Opinion in Structural Biology. 14 (5), 577-583 (2004).
- McPherson, A., Gavira, J. A. Introduction to protein crystallization. Acta Crystallographica Section F: Structural Biology Communications. 70 (1), 2-20 (2014).
- Gulbis, J. Protein crystallography: methods and protocols. Crystallography Reviews. 24 (2), 136-143 (2018).
- D’Arcy, A., Bergfors, T., Cowan-Jacob, S. W., Marsh, M. Microseed matrix screening for optimization in protein crystallization: What have we learned. Acta Crystallographica Section:F Structural Biology Communications. 70 (9), 1117-1126 (2014).
- Shaw Stewart, ., D, P., Kolek, S. A., Briggs, R. A., Chayen, N. E., Baldock, P. F. Random microseeding: a theoretical and practical exploration of seed stability and seeding techniques for successful protein crystallization. Crystal Growth & Design. 11 (8), 3432-3441 (2011).
- Junius, N., et al. A microfluidic device for both on-chip dialysis protein crystallization and in situ X-ray diffraction. Lab on a Chip. 20 (2), 296-310 (2020).
- Junius, N., Vahdatahar, E., Oksanen, E., Ferrer, J. L., Budayova-Spano, M. Optimization of crystallization of biological macromolecules using dialysis combined with temperature control. Journal of Applied Crystallography. 53 (3), 686-698 (2020).
- Vahdatahar, E., Junius, N., Budayova-Spano, M. Optimization of crystal growth for neutron macromolecular crystallography. Journal of Visualized Experiments. 169, (2021).
- Jaho, S., et al. Crystallization of proteins on chip by microdialysis for in situ X-ray diffraction studies. Journal of Visualized Experiments. 170, (2021).
- Liu, W., Cherezov, V. Crystallization of membrane proteins in lipidic mesophases. Journal of Visualized Experiments. 49, 2501 (2011).
- Ujwal, R., Abramson, J. High-throughput crystallization of membrane proteins using the lipidic bicelle method. Journal of Visualized Experiments. 59, (2012).
- Parker, J. L., Newstead, S. Membrane protein crystallisation: Current trends and future perspectives. Advances in Experimental Medicine and Biology. 922. , 61-72 (2016).
- Apostolopoulou, V., Junius, N., Sear, R. P., Budayova-Spano, M. Mixing salts and polyethylene glycol into protein solutions: The effects of diffusion across semi-permeable membranes and of convection. Crystal Growth & Design. 20 (6), 3927-3936 (2020).
- Neutze, R., Wouts, R., Vander Spoel, D., Weckert, E., Hajdu, J. Potential for biomolecular imaging with femtosecond X-ray pulses. Nature. 406 (6797), 752-757 (2000).
- Chapman, H. N., et al. Femtosecond X-ray protein nanocrystallography. Nature. 470 (7332), 73-78 (2011).
- Mizohata, E., Nakane, T., Fukuda, Y., Nango, E., Iwata, S. Serial femtosecond crystallography at the SACLA: breakthrough to dynamic structural biology. Biophysical Reviews. 10 (2), 209-218 (2018).
- Nogly, P., et al. Lipidic cubic phase serial millisecond crystallography using synchrotron radiation. IUCrJ. 2 (2), 168-176 (2015).
- Johansson, L. C., et al. XFEL structures of the human MT2 melatonin receptor reveal the basis of subtype selectivity. Nature. 569 (7755), 289-292 (2019).
- Owen, R. L., et al. Low-dose fixed-target serial synchrotron crystallography. Acta Crystallographica Section D: Structural Biology. 73 (4), 373-378 (2017).
- Weinert, T., et al. Serial millisecond crystallography for routine room-temperature structure determination at synchrotrons. Nature communications. 8 (1), 542 (2017).
- Axford, D., et al. Two states of a light-sensitive membrane protein captured at room temperature using thin-film sample mounts. Acta Crystallographica Section D: Structural Biology. 78 (1), 52-58 (2022).
- Nannenga, B. L., Gonen, T. The cryo-EM method microcrystal electron diffraction (MicroED). Nature Methods. 16 (5), 369-379 (2019).
- Nguyen, C., Gonen, T. Beyond protein structure determination with MicroED. Current Opinion in Structural Biology. 64, 51-58 (2020).
- Mu, X., Gillman, C., Nguyen, C., Gonen, T. An overview of microcrystal electron diffraction (MicroED). Annual Review of Biochemistry. 90, 431-450 (2021).
- Kwan, T. O. C., et al. Selection of biophysical methods for characterisation of membrane proteins. International Journal of Molecular Sciences. 22 (10), 2605 (2019).
- Pos, K. M., Purification Diederichs, K. crystallization and preliminary diffraction studies of AcrB, an inner-membrane multi-drug efflux protein. Acta Crystallographica Section D: Biological Crystallography. 58 (10), 1865-1867 (2002).
- Guan, L., Mirza, O., Verner, G., Iwata, S., Kaback, H. R. Structural determination of wild-type lactose permease. Proceedings of the National Academy of Sciences. 104 (39), 15294-15298 (2007).
- Kwan, T. O. C., Reis, R., Moraes, I. In situ measurements of polypeptide samples by dynamic light scattering: membrane proteins, a case study. Methods in Molecular Biology. , 189-202 (2021).
- Seddon, A. M., Curnow, P., Booth, P. J. Membrane proteins, lipids and detergents: not just a soap opera. Biochimica et Biophysica Acta (BBA)-Biomembranes. (1-2), 105-117 (2004).
- Wickstrand, C., et al. A tool for visualizing protein motions in time-resolved crystallography. Structural Dynamics. 7 (2), 024701 (2020).
- Orville, A. M. Recent results in time resolved serial femtosecond crystallography at XFELs. Current Opinion in Structural Biology. 65, 193-208 (2020).
- Schulz, E. C., Yorke, B. A., Pearson, A. R., Mehrabi, P. Best practices for time-resolved serial synchrotron crystallography. Acta Crystallographica Section D: Structural Biology. 78 (1), 14-29 (2022).
- Neutze, R., Brändén, G., Schertler, G. F. Membrane protein structural biology using X-ray free eletron lasers. Current Opinion in Structural Biology. 33, 115-125 (2015).
- Wierstall, U. Liquid sample delivery techniques for serial femtosecond crystallography. Philosophical Transactions of the Royal Society B: Biological Sciences. 369 (1647), 20130337 (2014).
- Shi, D., Nannenga, B. L., Iadanza, M. G., Gonen, T. Three-dimensional electron crystallography of protein microcrystals. eLife. 2, 01345 (2013).

