In vitro Chemische Kartierung von G-Quadruplex-DNA-Strukturen durch Bis-3-Chlorpiperidine
Summary
Bis-3-Chloropiperidine (B-CePs) sind nützliche chemische Sonden, um G-Quadruplex-Strukturen in DNA-Matrizen in vitro zu identifizieren und zu charakterisieren. Dieses Protokoll beschreibt das Verfahren zur Durchführung von Sondierungsreaktionen mit B-CePs und zur Auflösung von Reaktionsprodukten durch hochauflösende Polyacrylamid-Gelelektrophorese.
Abstract
G-Quadruplexe (G4s) sind biologisch relevante, nicht-kanonische DNA-Strukturen, die eine wichtige Rolle bei der Genexpression und bei Krankheiten spielen und wichtige therapeutische Ziele darstellen. Für die in vitro Charakterisierung von DNA innerhalb potenzieller G-Quadruplex-bildender Sequenzen (PQS) werden zugängliche Methoden benötigt. B-CePs sind eine Klasse von Alkylierungsmitteln, die sich als nützliche chemische Sonden für die Untersuchung der Struktur höherer Ordnung von Nukleinsäuren erwiesen haben. In dieser Arbeit wird ein neuer chemischer Mapping-Assay beschrieben, der die spezifische Reaktivität von B-CePs mit dem N7 von Guaninen ausnutzt, gefolgt von einer direkten Strangspaltung an den alkylierten Gs.
Um G4-Falten von ungefalteten DNA-Formen zu unterscheiden, verwenden wir B-CeP 1, um das Thrombin-bindende Aptamer (TBA) zu untersuchen, eine 15-mer-DNA, die in der Lage ist, die G4-Anordnung anzunehmen. Die Reaktion von B-CeP-antwortenden Guaninen mit B-CeP1 führt zu Produkten, die durch hochauflösende Polyacrylamid-Gelelektrophorese (PAGE) auf Einzelnukleotidebene aufgelöst werden können, indem einzelne Alkylierungsaddukte und DNA-Strangspaltung an den alkylierten Guaninen lokalisiert werden. Die Kartierung mit B-CePs ist ein einfaches und leistungsfähiges Werkzeug für die In-vitro-Charakterisierung von G-Quadruplex-bildenden DNA-Sequenzen, das die genaue Lokalisierung von Guaninen ermöglicht, die an der Bildung von G-Tetrades beteiligt sind.
Introduction
Neben der typischen Watson-Crick-Doppelhelix können Nukleinsäuren aufgrund ihrer Guanin-reichen Sequenzen verschiedene Sekundärstrukturen annehmen, wie z.B. die alternative G-Quadruplex (G4)-Form. Die G4-Struktur basiert auf der Bildung von planaren Tetrameren, sogenannten G-Tetraden, in denen vier Guanine durch Hoogsteen-Wasserstoffbrückenbindungen wechselwirken. G-Tetraden werden durch monovalente Kationen, die im Zentrum des Guaninkerns koordiniert sind, gestapelt und weiter stabilisiert (Abbildung 1)1.
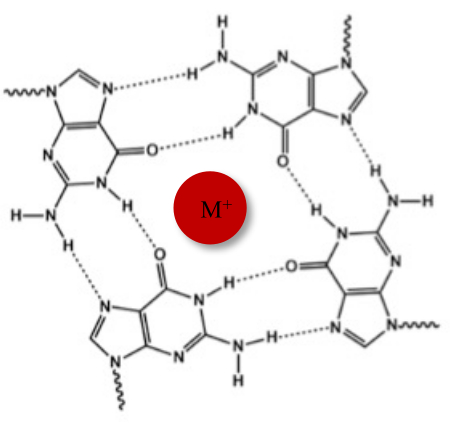
Abbildung 1: Schematische Darstellung einer G-Quadruplex-Struktur. (A) Schematische Darstellung einer G-Tetrade. Das planare Array wird durch Hoogsteen-Basenpaarung und durch ein zentrales Kation (M+) stabilisiert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Sequenzen mit vier oder mehr Durchläufen von mindestens zwei aufeinanderfolgenden Guanin-Nukleotiden sind potentielle G-Quadruplex-bildende Sequenzen (PQS), die sich in G-Quadruplex-Strukturen falten können. PQS befinden sich in vielen verschiedenen zellulären Kontexten, wie z.B. an Telomeren, Genpromotoren, ribosomaler DNA und Rekombinationsstellen, und sind an der Regulation vieler biologischer Prozesse beteiligt2. Daher ist die Identifizierung und experimentelle Validierung von G4s im menschlichen Genom, die derzeit hauptsächlich mit computergestützten Werkzeugen durchgeführt wird, ein biologisch relevantes Thema3. Um computergestützte Vorhersagen zu unterstützen oder unvorhergesehene G4-Strukturen zu erkennen, wird hier eine auf chemischer Kartierung basierende Methode zur Identifizierung der G4-Bildung in einem DNA-Template gezeigt, die die präzise Identifizierung von Guaninen ermöglicht, die die G-Tetradenstruktur bilden.
Der beschriebene chemische Mapping-Assay nutzt die unterschiedliche Reaktivität von Bis-3-Chlorpyperidinen (B-CePs) mit Guaninen nach der Bildung von G4-Strukturen. Aufgrund ihrer hohen Reaktivität mit Nukleophilen 4,5,6,7,8,9 sind B-CePs Nukleinsäure-Alkylanzien mit der Fähigkeit, sehr effizient mit der N7-Position von Guanin-Nukleotiden10 zu reagieren. Auf die Alkylierung folgt die Depurinierung und die Strangspaltung in einzel- und doppelsträngigen DNA-Konstrukten. Im Gegensatz dazu sind Guanine, die an der Bildung der G-Tetraden in G4-Anordnungen beteiligt sind, unempfindlich gegen die B-CeP-Alkylierung, da die N7-Positionvon Guanin an den Hoogsteen-Wasserstoffbrückenbindungen beteiligt ist. Diese spezifische Reaktivität von B-CePs ermöglicht nicht nur den Nachweis von G4-Strukturen, sondern auch die Identifizierung der Guanine, die die Tetrade bilden, da sie aus ihrem relativen Schutz vor Alkylierung im Vergleich zu Guaninen in einzel- und doppelsträngiger DNA abgeleitet werden können.
Das chemische Kartierungsprotokoll wird hier unter Verwendung von B-CeP 1 (Abbildung 2A) als Sonde für die Charakterisierung von Thrombin-bindendem Aptamer (TBA) vorgestellt, einer 15-mer-DNA, die in der Lage ist, die G4-Anordnung in Gegenwart von Kaliumkationen anzunehmen11,12. Die G4-Anordnung von TBA (G4-TBA) wird direkt mit zwei Kontrollen verglichen, nämlich TBA in der einzelsträngigen Form (ssTBA) und TBA, die zu ihrer komplementären Sequenz getempert wurde, um das doppelsträngige Konstrukt (dsTBA) zu bilden (Tabelle 1). Die Produkte der Sondierungsreaktionen werden durch hochauflösende Polyacrylamid-Gelelektrophorese (PAGE) auf Einzelnukleotidebene aufgelöst, indem einzelne Alkylierungsaddukte und DNA-Strangspaltungen an den alkylierten Guaninen lokalisiert werden. Die Visualisierung auf dem Gel wird durch Konjugation des TBA-Oligonukleotids mit einem Fluorophor an seinem 3′-Ende ermöglicht (Tabelle 1). Dieses Protokoll zeigt, wie TBA in seinen verschiedenen Konformationen (G4 und Kontrollen) gefaltet wird und wie Sondierungsreaktionen mit B-CePs gefolgt von PAGE durchgeführt werden.
Protocol
Representative Results
Discussion
G-Quadruplexe sind Nukleinsäure-Sekundärstrukturen, die sich typischerweise innerhalb von Guanin-reichen DNA-Sequenzen falten und aufgrund ihrer Assoziation mit genetischer Kontrolle und Krankheiten wichtige Forschungsziele sind. Die chemische Kartierung durch B-CePs ist ein nützliches Protokoll für die Charakterisierung von DNA-G4s, das verwendet werden kann, um die Guaninbasen zu identifizieren, die an der Bildung von G-Tetrades unter physiologischen Salzbedingungen beteiligt sind.
Die i…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Diese Arbeit wurde vom Institut für Pharmazeutische und Pharmakologische Wissenschaften der Universität Padua (PRIDJ-BIRD2019) unterstützt.
Materials
| Acrylamide/bis-acrylamide solution 40% | Applichem | A3658 | R45-46-20/21-25-36/38-43-48/23/ 24/25-62 |
| Ammonium per-sulfate (APS) | Sigma Aldrich | A7460 | |
| Analytical balance | Mettler Toledo | ||
| Autoclave | pbi international | ||
| Boric acid | Sigma Aldrich | B0252 | |
| Bromophenol blue Brilliant blue R | Sigma Aldrich | B0149 | |
| di-Sodium hydrogen phosphate dodecahydrate | Fluka | 71649 | |
| DMSO | Sigma Aldrich | 276855 | |
| DNA oligonucleotides | Integrated DNA Technologies | synthesis of custom sequences | |
| EDTA disodium | Sigma Aldrich | E5134 | |
| Formamide | Fluka | 40248 | H351-360D-373 |
| Gel imager | GE Healtcare | STORM B40 | |
| Glycerol | Sigma Aldrich | G5516 | |
| Micro tubes 0.5 mL | Sarstedt | 72.704 | |
| Potassium Chloride | Sigma Aldrich | P9541 | |
| Sequencing apparatus | Biometra | Model S2 | |
| Silanization solution I | Fluka | 85126 | H225, 314, 318, 336, 304, 400, 410 |
| Sodium phosphate monobasic | Carlo Erba | 480086 | |
| Speedvac concentrator | Thermo Scientific | Savant DNA 120 | |
| TEMED | Fluka | 87689 | R11-21/22-23-34 |
| Tris-HCl | MERCK | 1.08387.2500 | |
| Urea | Sigma Aldrich | 51456 | |
| UV-Vis spectrophotometer | Thermo Scientific | Nanodrop 1000 |
References
- Davis, J. T. G-quartets 40 years later: from 5′-GMP to molecular biology and supramolecular chemistry. Angewandte Chemie. 43 (6), 668-698 (2004).
- Varshney, D., Spiegel, J., Zyner, K., Tannahill, D., Balasubramanian, S. The regulation and functions of DNA and RNA G-quadruplexes. Nature Reviews Molecular Cell Biology. 21 (8), 459-474 (2020).
- Chambers, V. S., et al. High-throughput sequencing of DNA G-quadruplex structures in the human genome. Nature Biotechnology. 33 (8), 877-881 (2015).
- Zuravka, I., Sosic, A., Gatto, B., Gottlich, R. Synthesis and evaluation of a bis-3-chloropiperidine derivative incorporating an anthraquinone pharmacophore. Bioorganic & Medicinal Chemistry Letters. 25 (20), 4606-4609 (2015).
- Zuravka, I., Roesmann, R., Sosic, A., Gottlich, R., Gatto, B. Bis-3-chloropiperidines containing bridging lysine linkers: Influence of side chain structure on DNA alkylating activity. Bioorganic & Medicinal Chemistry. 23 (6), 1241-1250 (2015).
- Zuravka, I., et al. Synthesis and DNA cleavage activity of bis-3-chloropiperidines as alkylating agents. ChemMedChem. 9 (9), 2178-2185 (2014).
- Sosic, A., Gottlich, R., Fabris, D., Gatto, B. B-CePs as cross-linking probes for the investigation of RNA higher-order structure. Nucleic Acids Research. 49 (12), 6660-6672 (2021).
- Sosic, A., et al. Bis-3-chloropiperidines targeting TAR RNA as a novel strategy to impair the HIV-1 nucleocapsid protein. Molecules. 26 (7), 1874 (2021).
- Sosic, A., et al. In vitro evaluation of bis-3-chloropiperidines as RNA modulators targeting TAR and TAR-protein interaction. International Journal of Molecular Sciences. 23 (2), 582 (2022).
- Sosic, A., et al. Direct and topoisomerase II mediated DNA damage by bis-3-chloropiperidines: The importance of being an earnest G. ChemMedChem. 12 (17), 1471-1479 (2017).
- Bock, L. C., Griffin, L. C., Latham, J. A., Vermaas, E. H., Toole, J. J. Selection of single-stranded DNA molecules that bind and inhibit human thrombin. Nature. 355 (6360), 564-566 (1992).
- Paborsky, L. R., McCurdy, S. N., Griffin, L. C., Toole, J. J., Leung, L. L. The single-stranded DNA aptamer-binding site of human thrombin. The Journal of Biological Chemistry. 268 (28), 20808-20811 (1993).
- Carraro, C., et al. Behind the mirror: chirality tunes the reactivity and cytotoxicity of chloropiperidines as potential anticancer agents. ACS Medicinal Chemistry Letters. 10 (4), 552-557 (2019).
- Carraro, C., et al. Appended aromatic moieties in flexible bis-3-chloropiperidines confer tropism against pancreatic cancer cells. ChemMedChem. 16 (5), 860-868 (2021).
- Kypr, J., Kejnovska, I., Renciuk, D., Vorlickova, M. Circular dichroism and conformational polymorphism of DNA. Nucleic Acids Research. 37 (6), 1713-1725 (2009).
- Onel, B., Wu, G., Sun, D., Lin, C., Yang, D. Electrophoretic mobility shift assay and dimethyl sulfate footprinting for characterization of G-quadruplexes and G-quadruplex-protein complexes. Methods in Molecular Biology. 2035, 201-222 (2019).

