In vitro Cartographie chimique des structures de l’ADN G-quadruplex par Bis-3-chloropipéridines
Summary
Les bis-3-chloropipéridines (B-CePs) sont des sondes chimiques utiles pour identifier et caractériser les structures G-quadruplex dans les matrices d’ADN in vitro. Ce protocole détaille la procédure permettant d’effectuer des réactions de sondage avec des B-CeP et de résoudre les produits de réaction par électrophorèse sur gel de polyacrylamide à haute résolution.
Abstract
Les G-quadruplexes (G4s) sont des structures d’ADN biologiquement pertinentes, non canoniques, qui jouent un rôle important dans l’expression des gènes et les maladies, représentant des cibles thérapeutiques importantes. Des méthodes accessibles sont nécessaires pour la caractérisation in vitro de l’ADN dans les séquences potentielles de formation de G-quadruplex (PQS). Les B-CeP sont une classe d’agents alkylants qui se sont avérés être des sondes chimiques utiles pour l’étude de la structure d’ordre supérieur des acides nucléiques. Cet article décrit un nouveau test de cartographie chimique exploitant la réactivité spécifique des B-CeP avec le N7 des guanines, suivi d’un clivage direct des brins au niveau des G alkylés.
En effet, pour distinguer les plis G4 des formes d’ADN dépliées, nous utilisons B-CeP 1 pour sonder l’aptamère de liaison à la thrombine (TBA), un ADN de 15 mères capable d’assumer l’arrangement G4. La réaction des guanines répondant au B-CeP avec le B-CeP 1 permet d’obtenir des produits qui peuvent être résolus par électrophorèse sur gel de polyacrylamide (PAGE) à haute résolution au niveau d’un seul nucléotide en localisant les adduits d’alkylation individuels et le clivage des brins d’ADN au niveau des guanines alkylées. La cartographie à l’aide de B-CePs est un outil simple et puissant pour la caractérisation in vitro de séquences d’ADN formant G-quadruplex, permettant la localisation précise des guanines impliquées dans la formation des G-tétrades.
Introduction
En plus de la double hélice typique de Watson-Crick, les acides nucléiques peuvent adopter diverses structures secondaires, telles que la forme alternative G-quadruplex (G4), en raison de leurs séquences riches en guanine. La structure G4 est basée sur la formation de tétramères planaires, appelés G-tétrades, dans lesquels quatre guanines interagissent par l’intermédiaire de liaisons hydrogène Hoogsteen. Les G-tétrades sont empilées et stabilisées par des cations monovalents qui sont coordonnés au centre du noyau de guanine (Figure 1)1.
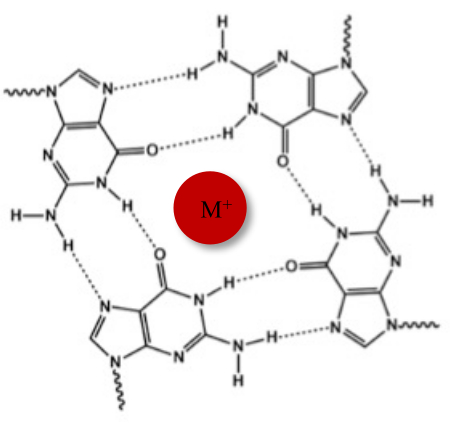
Figure 1 : Représentation schématique d’une structure G-quadruplex. (A) Représentation schématique d’une tétrade G. Le réseau planaire est stabilisé par l’appariement de bases de Hoogsteen et par un cation central (M+). Veuillez cliquer ici pour voir une version agrandie de cette figure.
Les séquences comportant quatre cycles ou plus d’au moins deux nucléotides consécutifs de guanine sont des séquences potentielles formant des G-quadruplex (PQS) qui peuvent se replier dans des structures G-quadruplex. Les PQS sont situés dans de nombreux contextes cellulaires différents, tels que les télomères, les promoteurs de gènes, l’ADN ribosomique et les sites de recombinaison, et sont impliqués dans la régulation de nombreux processus biologiques2. Par conséquent, l’identification et la validation expérimentale des G4 dans le génome humain, qui sont actuellement effectuées principalement à l’aide d’outils informatiques, est une question biologiquement pertinente3. Afin de soutenir les prédictions computationnelles ou de détecter des structures G4 non prédites, une méthode accessible basée sur la cartographie chimique pour identifier la formation de G4 dans une matrice d’ADN est présentée ici, permettant l’identification précise des guanines formant la structure G-tétrade.
Le test de cartographie chimique rapporté exploite la réactivité différente des bis-3-chloropipéridines (B-CePs) avec les guanines suite à la formation de structures G4. En raison de leur forte réactivité avec les nucléophiles 4,5,6,7,8,9, les B-CeP sont des agents alkylants d’acides nucléiques ayant la capacité de réagir très efficacement avec la position N 7 des nucléotides de guanine10. L’alkylation est suivie de la dépurination et du clivage des brins dans les constructions d’ADN simple et double brin. Au contraire, les guanines impliquées dans la formation des G-tétrades dans les arrangements G4 sont imperméables à l’alkylation B-CeP, car la position N7 des guanines est impliquée dans les liaisons hydrogène de Hoogsteen. Cette réactivité spécifique des B-CePs permet non seulement la détection des structures G4, mais aussi l’identification des guanines formant la ou les tétrades, telles qu’elles peuvent être déduites de leur protection relative contre l’alkylation par rapport aux guanines dans l’ADN simple et double brin.
Le protocole de cartographie chimique est présenté ici en utilisant B-CeP 1 (Figure 2A) comme sonde pour la caractérisation de l’aptamère de liaison à la thrombine (TBA), un ADN de 15 mères capable d’assumer l’arrangement G4 en présence de cations potassiques11,12. L’arrangement G4 de TBA (G4-TBA) est directement comparé à deux témoins, à savoir TBA sous la forme simple brin (ssTBA) et TBA recuit à sa séquence complémentaire pour former la construction double brin (dsTBA) (tableau 1). Les produits des réactions de sondage sont résolus par électrophorèse sur gel de polyacrylamide à haute résolution (PAGE) au niveau du mononucléotide en localisant les adduits d’alkylation individuels et le clivage des brins d’ADN au niveau des guanines alkylées. La visualisation sur le gel est rendue possible par la conjugaison de l’oligonucléotide TBA avec un fluorophore à son extrémité 3′ (Tableau 1). Ce protocole montre comment plier TBA dans ses différentes conformations (G4 et témoins), et comment effectuer des réactions de sondage avec des B-CeP suivis de PAGE.
Protocol
Representative Results
Discussion
Les G-quadruplexes sont des structures secondaires d’acides nucléiques qui se replient généralement dans des séquences d’ADN riches en guanine et sont des cibles de recherche importantes en raison de leur association avec le contrôle génétique et les maladies. La cartographie chimique par B-CePs est un protocole utile pour la caractérisation des G4 de l’ADN, qui peut être utilisé pour identifier les bases de guanine impliquées dans la formation des G-tétrades dans des conditions physiologiques de sel.</…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Ce travail a été soutenu par le Département des Sciences Pharmaceutiques et Pharmacologiques de l’Université de Padoue (PRIDJ-BIRD2019).
Materials
| Acrylamide/bis-acrylamide solution 40% | Applichem | A3658 | R45-46-20/21-25-36/38-43-48/23/ 24/25-62 |
| Ammonium per-sulfate (APS) | Sigma Aldrich | A7460 | |
| Analytical balance | Mettler Toledo | ||
| Autoclave | pbi international | ||
| Boric acid | Sigma Aldrich | B0252 | |
| Bromophenol blue Brilliant blue R | Sigma Aldrich | B0149 | |
| di-Sodium hydrogen phosphate dodecahydrate | Fluka | 71649 | |
| DMSO | Sigma Aldrich | 276855 | |
| DNA oligonucleotides | Integrated DNA Technologies | synthesis of custom sequences | |
| EDTA disodium | Sigma Aldrich | E5134 | |
| Formamide | Fluka | 40248 | H351-360D-373 |
| Gel imager | GE Healtcare | STORM B40 | |
| Glycerol | Sigma Aldrich | G5516 | |
| Micro tubes 0.5 mL | Sarstedt | 72.704 | |
| Potassium Chloride | Sigma Aldrich | P9541 | |
| Sequencing apparatus | Biometra | Model S2 | |
| Silanization solution I | Fluka | 85126 | H225, 314, 318, 336, 304, 400, 410 |
| Sodium phosphate monobasic | Carlo Erba | 480086 | |
| Speedvac concentrator | Thermo Scientific | Savant DNA 120 | |
| TEMED | Fluka | 87689 | R11-21/22-23-34 |
| Tris-HCl | MERCK | 1.08387.2500 | |
| Urea | Sigma Aldrich | 51456 | |
| UV-Vis spectrophotometer | Thermo Scientific | Nanodrop 1000 |
References
- Davis, J. T. G-quartets 40 years later: from 5′-GMP to molecular biology and supramolecular chemistry. Angewandte Chemie. 43 (6), 668-698 (2004).
- Varshney, D., Spiegel, J., Zyner, K., Tannahill, D., Balasubramanian, S. The regulation and functions of DNA and RNA G-quadruplexes. Nature Reviews Molecular Cell Biology. 21 (8), 459-474 (2020).
- Chambers, V. S., et al. High-throughput sequencing of DNA G-quadruplex structures in the human genome. Nature Biotechnology. 33 (8), 877-881 (2015).
- Zuravka, I., Sosic, A., Gatto, B., Gottlich, R. Synthesis and evaluation of a bis-3-chloropiperidine derivative incorporating an anthraquinone pharmacophore. Bioorganic & Medicinal Chemistry Letters. 25 (20), 4606-4609 (2015).
- Zuravka, I., Roesmann, R., Sosic, A., Gottlich, R., Gatto, B. Bis-3-chloropiperidines containing bridging lysine linkers: Influence of side chain structure on DNA alkylating activity. Bioorganic & Medicinal Chemistry. 23 (6), 1241-1250 (2015).
- Zuravka, I., et al. Synthesis and DNA cleavage activity of bis-3-chloropiperidines as alkylating agents. ChemMedChem. 9 (9), 2178-2185 (2014).
- Sosic, A., Gottlich, R., Fabris, D., Gatto, B. B-CePs as cross-linking probes for the investigation of RNA higher-order structure. Nucleic Acids Research. 49 (12), 6660-6672 (2021).
- Sosic, A., et al. Bis-3-chloropiperidines targeting TAR RNA as a novel strategy to impair the HIV-1 nucleocapsid protein. Molecules. 26 (7), 1874 (2021).
- Sosic, A., et al. In vitro evaluation of bis-3-chloropiperidines as RNA modulators targeting TAR and TAR-protein interaction. International Journal of Molecular Sciences. 23 (2), 582 (2022).
- Sosic, A., et al. Direct and topoisomerase II mediated DNA damage by bis-3-chloropiperidines: The importance of being an earnest G. ChemMedChem. 12 (17), 1471-1479 (2017).
- Bock, L. C., Griffin, L. C., Latham, J. A., Vermaas, E. H., Toole, J. J. Selection of single-stranded DNA molecules that bind and inhibit human thrombin. Nature. 355 (6360), 564-566 (1992).
- Paborsky, L. R., McCurdy, S. N., Griffin, L. C., Toole, J. J., Leung, L. L. The single-stranded DNA aptamer-binding site of human thrombin. The Journal of Biological Chemistry. 268 (28), 20808-20811 (1993).
- Carraro, C., et al. Behind the mirror: chirality tunes the reactivity and cytotoxicity of chloropiperidines as potential anticancer agents. ACS Medicinal Chemistry Letters. 10 (4), 552-557 (2019).
- Carraro, C., et al. Appended aromatic moieties in flexible bis-3-chloropiperidines confer tropism against pancreatic cancer cells. ChemMedChem. 16 (5), 860-868 (2021).
- Kypr, J., Kejnovska, I., Renciuk, D., Vorlickova, M. Circular dichroism and conformational polymorphism of DNA. Nucleic Acids Research. 37 (6), 1713-1725 (2009).
- Onel, B., Wu, G., Sun, D., Lin, C., Yang, D. Electrophoretic mobility shift assay and dimethyl sulfate footprinting for characterization of G-quadruplexes and G-quadruplex-protein complexes. Methods in Molecular Biology. 2035, 201-222 (2019).

