In Vitro (시험관) Bis-3-Chloropiperidines에 의한 G-Quadruplex DNA 구조의 화학적 매핑
Summary
Bis-3-chloropiperidines(B-CeP)는 in vitro에서 DNA 템플릿의 G-quadruplex 구조를 식별하고 특성화하는 데 유용한 화학 프로브입니다. 이 프로토콜은 B-CeP를 사용하여 프로빙 반응을 수행하고 고분해능 폴리아크릴아미드 겔 전기영동으로 반응 생성물을 분해하는 절차를 자세히 설명합니다.
Abstract
G-quadruplex(G4s)는 유전자 발현 및 질병에 중요한 역할을 하는 생물학적으로 관련된 비표준 DNA 구조로, 중요한 치료 표적을 나타냅니다. 잠재적인 G-quadruplex-forming sequences(PQS) 내에서 DNA의 in vitro 특성 분석을 위해서는 접근 가능한 방법이 필요합니다. B-CeP는 핵산의 고차원 구조 조사에 유용한 화학 프로브로 입증된 알킬화제의 한 종류입니다. 이 논문은 B-CeP와 구아닌의 N7의 특정 반응성을 활용한 후 알킬화된 Gs에서 직접 가닥 절단을 수행하는 새로운 화학 매핑 분석에 대해 설명합니다.
즉, G4 접힘과 풀린 DNA 형태를 구별하기 위해 B-CeP 1을 사용하여 G4 배열을 가정할 수 있는 15-mer DNA인 트롬빈 결합 압타머(TBA)를 조사합니다. B-CeP 반응 구아닌과 B-CeP 1의 반응은 알킬화된 구아닌에서 개별 알킬화 부가물 및 DNA 가닥 절단을 찾아 단일 뉴클레오티드 수준에서 고분해능 폴리아크릴아미드 겔 전기영동(PAGE)으로 분해할 수 있는 산물을 생성합니다. B-CeP를 사용한 맵핑은 G-quadruplex 형성 DNA 염기서열의 in vitro 특성 분석을 위한 간단하고 강력한 도구로, G-tetrads 형성에 관여하는 구아닌의 정확한 위치를 파악할 수 있습니다.
Introduction
전형적인 왓슨-크릭(Watson-Crick) 이중나선 외에도, 핵산은 구아닌이 풍부한 서열로 인해 양자택일 G-4중(G4) 형태와 같은 다양한 2차 구조를 채택할 수 있습니다. G4 구조는 4개의 구아닌이 Hoogsteen 수소 결합을 통해 상호 작용하는 G-tetrads라고 하는 평면 사량체의 형성을 기반으로 합니다. G-tetrad는 구아닌 코어의 중심에서 조정되는 1가 양이온에 의해 적층되고 더욱 안정화됩니다(그림 1)1.
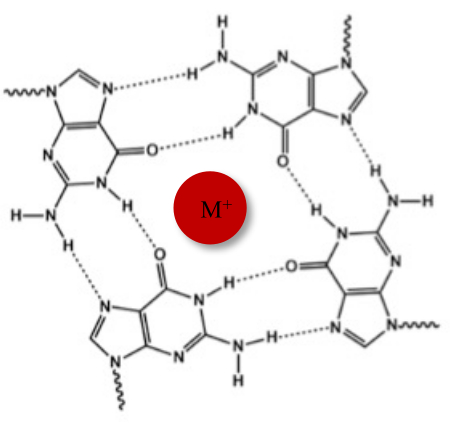
그림 1: G-quadruplex 구조 의 개략도. (A) G-tetrad의 개략적 표현. 평면 어레이는 Hoogsteen 염기쌍과 중심 양이온(M+)에 의해 안정화됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
적어도 2개의 연속적인 구아닌 뉴클레오티드가 4회 이상 실행된 서열은 G-4중복합 구조에서 접힐 수 있는 잠재적인 G-사중-형성 서열(PQS)이다. PQS는 텔로미어, 유전자 프로모터, 리보솜 DNA, 재조합 부위 등 다양한 세포 환경에 위치하며, 많은 생물학적 과정의 조절에 관여한다2. 따라서 현재 주로 계산 도구를 통해 수행되는 인간 게놈에서 G4의 식별 및 실험적 검증은 생물학적으로 관련된 문제입니다3. 전산 예측을 지원하거나 예측하지 못한 G4 구조를 검출하기 위해 DNA 템플릿에서 G4 형성을 식별하기 위한 화학적 매핑을 기반으로 하는 접근 가능한 방법이 여기에 표시되어 G-tetrad 구조를 형성하는 구아닌을 정확하게 식별할 수 있습니다.
보고된 화학 매핑 분석은 G4 구조 형성 후 구아닌과 비스-3-클로로피페리딘(B-CeP)의 다양한 반응성을 이용합니다. 친핵체 4,5,6,7,8,9와의 높은 반응성으로 인해 B-CeP 는 구아닌 뉴클레오티드 10의 N 7 위치와 매우 효율적으로 반응할 수 있는 핵산 알킬화제입니다. 알킬화 다음에는 단일 및 이중 가닥 DNA 구조에서 탈피 및 가닥 절단이 뒤따릅니다. 반대로, G4 배열에서 G-테트라드의 형성에 관여하는 구아닌은 구아닌의 N7 위치가 Hoogsteen 수소 결합에 연루되어 있기 때문에 B-CeP 알킬화에 영향을 받지 않습니다. B-CeP의 이러한 특이적 반응성은 G4 구조의 검출뿐만 아니라 단일 및 이중 가닥 DNA의 구아닌과 비교하여 알킬화로부터 상대적인 보호로부터 추론할 수 있기 때문에 테트라드를 형성하는 구아닌의 식별도 가능하게 합니다.
화학적 매핑 프로토콜은 칼륨 양이온(11,12)의 존재 하에서 G4 배열을 가정할 수 있는 15-mer DNA인 트롬빈 결합 압타머(TBA)의 특성화를 위한 프로브로서 B-CeP 1(그림 2A)을 사용하여 여기에 보고됩니다. TBA(G4-TBA)의 G4 배열은 두 개의 대조군, 즉 단일 가닥 형태의 TBA(ssTBA)와 이중 가닥 구조(dsTBA)를 형성하기 위해 상보적인 서열로 어닐링된 TBA와 직접 비교됩니다(표 1). 프로빙 반응 산물은 알킬화된 구아닌에서 개별 알킬화 부가물과 DNA 가닥 절단을 찾아 단일 뉴클레오티드 수준에서 고분해능 폴리아크릴아미드 겔 전기영동(PAGE)으로 분해됩니다. 겔의 시각화는 TBA 올리고뉴클레오티드와 3′-말단의 형광단을 결합함으로써 가능합니다(표 1). 이 프로토콜은 TBA를 다양한 형태(G4 및 대조군)로 접는 방법과 B-CeP를 사용한 후 PAGE를 사용하여 프로빙 반응을 수행하는 방법을 보여줍니다.
Protocol
Representative Results
Discussion
G-quadruplex는 일반적으로 구아닌이 풍부한 DNA 염기서열 내에서 접히는 핵산 2차 구조이며, 유전적 제어 및 질병과의 연관성 때문에 중요한 연구 대상입니다. B-CePs에 의한 화학적 매핑은 생리학적 염분 조건에서 G-tetrads의 형성에 관여하는 구아닌 염기를 식별하는 데 사용할 수 있는 DNA G4의 특성 분석에 유용한 프로토콜입니다.
이 프로토콜에 사용된 화학 프로브는 B-CeP 1(<strong cl…
Disclosures
The authors have nothing to disclose.
Acknowledgements
이 연구는 파도바 대학교 제약 및 약리학과(PRIDJ-BIRD2019)의 지원을 받았습니다.
Materials
| Acrylamide/bis-acrylamide solution 40% | Applichem | A3658 | R45-46-20/21-25-36/38-43-48/23/ 24/25-62 |
| Ammonium per-sulfate (APS) | Sigma Aldrich | A7460 | |
| Analytical balance | Mettler Toledo | ||
| Autoclave | pbi international | ||
| Boric acid | Sigma Aldrich | B0252 | |
| Bromophenol blue Brilliant blue R | Sigma Aldrich | B0149 | |
| di-Sodium hydrogen phosphate dodecahydrate | Fluka | 71649 | |
| DMSO | Sigma Aldrich | 276855 | |
| DNA oligonucleotides | Integrated DNA Technologies | synthesis of custom sequences | |
| EDTA disodium | Sigma Aldrich | E5134 | |
| Formamide | Fluka | 40248 | H351-360D-373 |
| Gel imager | GE Healtcare | STORM B40 | |
| Glycerol | Sigma Aldrich | G5516 | |
| Micro tubes 0.5 mL | Sarstedt | 72.704 | |
| Potassium Chloride | Sigma Aldrich | P9541 | |
| Sequencing apparatus | Biometra | Model S2 | |
| Silanization solution I | Fluka | 85126 | H225, 314, 318, 336, 304, 400, 410 |
| Sodium phosphate monobasic | Carlo Erba | 480086 | |
| Speedvac concentrator | Thermo Scientific | Savant DNA 120 | |
| TEMED | Fluka | 87689 | R11-21/22-23-34 |
| Tris-HCl | MERCK | 1.08387.2500 | |
| Urea | Sigma Aldrich | 51456 | |
| UV-Vis spectrophotometer | Thermo Scientific | Nanodrop 1000 |
References
- Davis, J. T. G-quartets 40 years later: from 5′-GMP to molecular biology and supramolecular chemistry. Angewandte Chemie. 43 (6), 668-698 (2004).
- Varshney, D., Spiegel, J., Zyner, K., Tannahill, D., Balasubramanian, S. The regulation and functions of DNA and RNA G-quadruplexes. Nature Reviews Molecular Cell Biology. 21 (8), 459-474 (2020).
- Chambers, V. S., et al. High-throughput sequencing of DNA G-quadruplex structures in the human genome. Nature Biotechnology. 33 (8), 877-881 (2015).
- Zuravka, I., Sosic, A., Gatto, B., Gottlich, R. Synthesis and evaluation of a bis-3-chloropiperidine derivative incorporating an anthraquinone pharmacophore. Bioorganic & Medicinal Chemistry Letters. 25 (20), 4606-4609 (2015).
- Zuravka, I., Roesmann, R., Sosic, A., Gottlich, R., Gatto, B. Bis-3-chloropiperidines containing bridging lysine linkers: Influence of side chain structure on DNA alkylating activity. Bioorganic & Medicinal Chemistry. 23 (6), 1241-1250 (2015).
- Zuravka, I., et al. Synthesis and DNA cleavage activity of bis-3-chloropiperidines as alkylating agents. ChemMedChem. 9 (9), 2178-2185 (2014).
- Sosic, A., Gottlich, R., Fabris, D., Gatto, B. B-CePs as cross-linking probes for the investigation of RNA higher-order structure. Nucleic Acids Research. 49 (12), 6660-6672 (2021).
- Sosic, A., et al. Bis-3-chloropiperidines targeting TAR RNA as a novel strategy to impair the HIV-1 nucleocapsid protein. Molecules. 26 (7), 1874 (2021).
- Sosic, A., et al. In vitro evaluation of bis-3-chloropiperidines as RNA modulators targeting TAR and TAR-protein interaction. International Journal of Molecular Sciences. 23 (2), 582 (2022).
- Sosic, A., et al. Direct and topoisomerase II mediated DNA damage by bis-3-chloropiperidines: The importance of being an earnest G. ChemMedChem. 12 (17), 1471-1479 (2017).
- Bock, L. C., Griffin, L. C., Latham, J. A., Vermaas, E. H., Toole, J. J. Selection of single-stranded DNA molecules that bind and inhibit human thrombin. Nature. 355 (6360), 564-566 (1992).
- Paborsky, L. R., McCurdy, S. N., Griffin, L. C., Toole, J. J., Leung, L. L. The single-stranded DNA aptamer-binding site of human thrombin. The Journal of Biological Chemistry. 268 (28), 20808-20811 (1993).
- Carraro, C., et al. Behind the mirror: chirality tunes the reactivity and cytotoxicity of chloropiperidines as potential anticancer agents. ACS Medicinal Chemistry Letters. 10 (4), 552-557 (2019).
- Carraro, C., et al. Appended aromatic moieties in flexible bis-3-chloropiperidines confer tropism against pancreatic cancer cells. ChemMedChem. 16 (5), 860-868 (2021).
- Kypr, J., Kejnovska, I., Renciuk, D., Vorlickova, M. Circular dichroism and conformational polymorphism of DNA. Nucleic Acids Research. 37 (6), 1713-1725 (2009).
- Onel, B., Wu, G., Sun, D., Lin, C., Yang, D. Electrophoretic mobility shift assay and dimethyl sulfate footprinting for characterization of G-quadruplexes and G-quadruplex-protein complexes. Methods in Molecular Biology. 2035, 201-222 (2019).

