इन विट्रो "बीआईएस -3-क्लोरोपिपेरिडिन द्वारा जी-चौगुनी डीएनए संरचनाओं का रासायनिक मानचित्रण"।
Summary
बीआईएस -3-क्लोरोपिपेरिडीन (बी-सीईपी) विट्रो में डीएनए टेम्प्लेट में जी-चौगुनी संरचनाओं की पहचान और विशेषता के लिए उपयोगी रासायनिक जांच हैं। यह प्रोटोकॉल बी-सीईपी के साथ जांच प्रतिक्रियाओं को करने और उच्च-रिज़ॉल्यूशन पॉलीक्रिलामाइड जेल वैद्युतकणसंचलन द्वारा प्रतिक्रिया उत्पादों को हल करने की प्रक्रिया का विवरण देता है।
Abstract
जी-चौगुनी (जी 4 एस) जैविक रूप से प्रासंगिक, गैर-कैननिकल डीएनए संरचनाएं हैं जो जीन अभिव्यक्ति और बीमारियों में महत्वपूर्ण भूमिका निभाती हैं, महत्वपूर्ण चिकित्सीय लक्ष्यों का प्रतिनिधित्व करती हैं। संभावित जी-चौगुनी बनाने वाले अनुक्रमों (पीक्यूएस) के भीतर डीएनए के इन विट्रो लक्षण वर्णन के लिए सुलभ तरीकों की आवश्यकता होती है। बी-सीईपी अल्काइलेटिंग एजेंटों का एक वर्ग है जो न्यूक्लिक एसिड की उच्च-क्रम संरचना की जांच के लिए उपयोगी रासायनिक जांच साबित हुए हैं। यह पेपर एक नए रासायनिक मानचित्रण परख का वर्णन करता है जो गुआनिन के एन7 के साथ बी-सीईपी की विशिष्ट प्रतिक्रिया का फायदा उठाता है, इसके बाद अल्काइलेटेड जीएस पर प्रत्यक्ष स्ट्रैंड क्लीवेज होता है।
अर्थात्, जी 4 सिलवटों को अनफोल्डेड डीएनए रूपों से अलग करने के लिए, हम थ्रोम्बिन-बाइंडिंग एपटामर (टीबीए) की जांच करने के लिए बी-सीईपी 1 का उपयोग करते हैं, एक 15-मेर डीएनए जो जी 4 व्यवस्था को ग्रहण करने में सक्षम है। बी-सीईपी 1 के साथ बी-सीईपी-प्रतिक्रिया देने वाले गुआनिन की प्रतिक्रिया से ऐसे उत्पाद उत्पन्न होते हैं जिन्हें एकल-न्यूक्लियोटाइड स्तर पर उच्च-रिज़ॉल्यूशन पॉलीक्रिलामाइड जेल वैद्युतकणसंचलन (पेज) द्वारा हल किया जा सकता है। बी-सीईपी का उपयोग करके मैपिंग जी-चौगुनी बनाने वाले डीएनए अनुक्रमों के इन विट्रो लक्षण वर्णन के लिए एक सरल और शक्तिशाली उपकरण है, जो जी-टेट्राड्स के गठन में शामिल गुआनिन के सटीक स्थान को सक्षम करता है।
Introduction
विशिष्ट वाटसन-क्रिक डबल हेलिक्स के अलावा, न्यूक्लिक एसिड विभिन्न माध्यमिक संरचनाओं को अपना सकते हैं, जैसे कि वैकल्पिक जी-चौगुनी (जी 4) रूप, उनके गुआनिन-समृद्ध अनुक्रमों के कारण। जी 4 संरचना प्लानर टेट्रामर्स के गठन पर आधारित है, जिसे जी-टेट्राड्स कहा जाता है, जिसमें चार गुआनिन हूगस्टीन हाइड्रोजन बॉन्ड के माध्यम से बातचीत करते हैं। जी-टेट्राड्स को मोनोवेलेंट पिंजरों द्वारा स्टैक और आगे स्थिर किया जाता है जो गुआनिन कोर (चित्रा 1)1 के केंद्र में समन्वित होते हैं।
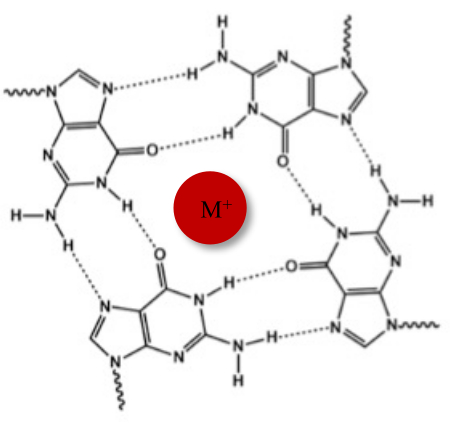
चित्र 1: जी-चौगुनी संरचना का योजनाबद्ध प्रतिनिधित्व। (ए) जी-टेट्राड का योजनाबद्ध प्रतिनिधित्व। प्लानर सरणी को हूगस्टीन बेस-पेयरिंग और एक केंद्रीय केशन (एम +) द्वारा स्थिर किया जाता है। कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहाँ क्लिक करें.
कम से कम दो लगातार गुआनिन न्यूक्लियोटाइड के चार या अधिक रन वाले अनुक्रम संभावित जी-चौगुनी बनाने वाले अनुक्रम (पीक्यूएस) हैं जो जी-चौगुनी संरचनाओं में फोल्ड हो सकते हैं। पीक्यूएस कई अलग-अलग सेलुलर संदर्भों में स्थित हैं, जैसे कि टेलोमेरेस, जीन प्रमोटर, राइबोसोमल डीएनए और पुनर्संयोजन साइटों पर, और कईजैविक प्रक्रियाओं के विनियमन में शामिल हैं। इसलिए, मानव जीनोम में जी 4 एस की पहचान और प्रयोगात्मक सत्यापन, जो वर्तमान में मुख्य रूप से कम्प्यूटेशनल टूल के माध्यम से किया जाता है, एक जैविक रूप से प्रासंगिक मुद्दाहै। कम्प्यूटेशनल भविष्यवाणियों का समर्थन करने या अप्रत्याशित जी 4 संरचनाओं का पता लगाने के लिए, डीएनए टेम्पलेट में जी 4 गठन की पहचान करने के लिए रासायनिक मानचित्रण पर आधारित एक सुलभ विधि यहां दिखाई गई है, जिससे जी-टेट्राड संरचना बनाने वाले गुआनिन की सटीक पहचान हो सकती है।
रिपोर्ट की गई रासायनिक मानचित्रण परख जी 4 संरचनाओं के गठन के बाद गुआनिन के साथ बीआईएस -3-क्लोरोपिपेरिडिन (बी-सीईपी) की विभिन्न प्रतिक्रिया का फायदा उठाती है। न्यूक्लियोफाइल 4,5,6,7,8,9 के साथ उनकी उच्च प्रतिक्रिया के कारण, बी-सीईपी न्यूक्लिक एसिड-अल्काइलेटिंग एजेंट हैं जो गुआनिन न्यूक्लियोटाइड10 की एन7 स्थिति के साथ बहुत कुशलता से प्रतिक्रिया करने की क्षमता रखते हैं। अल्काइलेशन के बाद एकल और डबल-फंसे डीएनए संरचनाओं में डिप्यूरिनेशन और स्ट्रैंड क्लीवेज होता है। इसके विपरीत, जी 4 व्यवस्था में जी-टेट्राड्स के गठन में शामिल गुआनिन बी-सीईपी अल्काइलेशन के लिए अभेद्य हैं, क्योंकि गुआनिन की एन7 स्थिति को हूगस्टीन हाइड्रोजन बॉन्ड में फंसाया गया है। बी-सीईपी की यह विशिष्ट प्रतिक्रिया न केवल जी 4 संरचनाओं का पता लगाने की अनुमति देती है, बल्कि टेट्राड (एस) बनाने वाले गुआनिन की पहचान भी करती है, क्योंकि उन्हें एकल और डबल-फंसे डीएनए में गुआनिन की तुलना में अल्काइलेशन से उनकी सापेक्ष सुरक्षा से अनुमान लगाया जा सकता है।
रासायनिक मानचित्रण प्रोटोकॉल को यहां बी-सीईपी 1 (चित्रा 2 ए) का उपयोग करके थ्रोम्बिन-बाइंडिंग एपटामर (टीबीए) के लक्षण वर्णन के लिए एक जांच के रूप में रिपोर्ट किया गया है, एक 15-मेर डीएनए पोटेशियम केशन11,12 की उपस्थिति में जी 4 व्यवस्था को ग्रहण करने में सक्षम है। टीबीए (जी 4-टीबीए) की जी 4 व्यवस्था की तुलना सीधे दो नियंत्रणों के साथ की जाती है, अर्थात् एकल-फंसे हुए रूप (एसएसटीबीए) में टीबीए और डबल-स्ट्रैंडेड कंस्ट्रक्शन (डीएसटीबीए) बनाने के लिए टीबीए को इसके पूरक अनुक्रम में लगाया जाता है (तालिका 1)। जांच प्रतिक्रियाओं के उत्पादों को एकल-न्यूक्लियोटाइड स्तर पर उच्च-रिज़ॉल्यूशन पॉलीक्रिलामाइड जेल वैद्युतकणसंचलन (पेज) द्वारा अल्काइलेटेड गुआनिन में व्यक्तिगत अल्काइलेशन जोड़ों और डीएनए स्ट्रैंड दरार का पता लगाकर हल किया जाता है। जेल पर विज़ुअलाइज़ेशन टीबीए ऑलिगोन्यूक्लियोटाइड के संयुग्मन द्वारा इसके 3′-अंत में फ्लोरोफोरे के साथ सक्षम होता है (तालिका 1)। यह प्रोटोकॉल दिखाता है कि टीबीए को इसके विभिन्न अनुरूपताओं (जी 4 और नियंत्रण) में कैसे मोड़ना है, और पेज के बाद बी-सीईपी के साथ जांच प्रतिक्रियाएं कैसे करें।
Protocol
Representative Results
Discussion
जी-चौगुनी न्यूक्लिक एसिड माध्यमिक संरचनाएं हैं जो आमतौर पर गुआनिन युक्त डीएनए अनुक्रमों के भीतर मुड़ती हैं, और आनुवंशिक नियंत्रण और बीमारियों के साथ उनके संबंध के कारण महत्वपूर्ण शोध लक्ष्य हैं। बी-?…
Disclosures
The authors have nothing to disclose.
Acknowledgements
यह काम फार्मास्युटिकल और फार्माकोलॉजिकल साइंसेज विभाग, पडोवा विश्वविद्यालय (पीआरआईडीजे -BIRD2019) द्वारा समर्थित था।
Materials
| Acrylamide/bis-acrylamide solution 40% | Applichem | A3658 | R45-46-20/21-25-36/38-43-48/23/ 24/25-62 |
| Ammonium per-sulfate (APS) | Sigma Aldrich | A7460 | |
| Analytical balance | Mettler Toledo | ||
| Autoclave | pbi international | ||
| Boric acid | Sigma Aldrich | B0252 | |
| Bromophenol blue Brilliant blue R | Sigma Aldrich | B0149 | |
| di-Sodium hydrogen phosphate dodecahydrate | Fluka | 71649 | |
| DMSO | Sigma Aldrich | 276855 | |
| DNA oligonucleotides | Integrated DNA Technologies | synthesis of custom sequences | |
| EDTA disodium | Sigma Aldrich | E5134 | |
| Formamide | Fluka | 40248 | H351-360D-373 |
| Gel imager | GE Healtcare | STORM B40 | |
| Glycerol | Sigma Aldrich | G5516 | |
| Micro tubes 0.5 mL | Sarstedt | 72.704 | |
| Potassium Chloride | Sigma Aldrich | P9541 | |
| Sequencing apparatus | Biometra | Model S2 | |
| Silanization solution I | Fluka | 85126 | H225, 314, 318, 336, 304, 400, 410 |
| Sodium phosphate monobasic | Carlo Erba | 480086 | |
| Speedvac concentrator | Thermo Scientific | Savant DNA 120 | |
| TEMED | Fluka | 87689 | R11-21/22-23-34 |
| Tris-HCl | MERCK | 1.08387.2500 | |
| Urea | Sigma Aldrich | 51456 | |
| UV-Vis spectrophotometer | Thermo Scientific | Nanodrop 1000 |
References
- Davis, J. T. G-quartets 40 years later: from 5′-GMP to molecular biology and supramolecular chemistry. Angewandte Chemie. 43 (6), 668-698 (2004).
- Varshney, D., Spiegel, J., Zyner, K., Tannahill, D., Balasubramanian, S. The regulation and functions of DNA and RNA G-quadruplexes. Nature Reviews Molecular Cell Biology. 21 (8), 459-474 (2020).
- Chambers, V. S., et al. High-throughput sequencing of DNA G-quadruplex structures in the human genome. Nature Biotechnology. 33 (8), 877-881 (2015).
- Zuravka, I., Sosic, A., Gatto, B., Gottlich, R. Synthesis and evaluation of a bis-3-chloropiperidine derivative incorporating an anthraquinone pharmacophore. Bioorganic & Medicinal Chemistry Letters. 25 (20), 4606-4609 (2015).
- Zuravka, I., Roesmann, R., Sosic, A., Gottlich, R., Gatto, B. Bis-3-chloropiperidines containing bridging lysine linkers: Influence of side chain structure on DNA alkylating activity. Bioorganic & Medicinal Chemistry. 23 (6), 1241-1250 (2015).
- Zuravka, I., et al. Synthesis and DNA cleavage activity of bis-3-chloropiperidines as alkylating agents. ChemMedChem. 9 (9), 2178-2185 (2014).
- Sosic, A., Gottlich, R., Fabris, D., Gatto, B. B-CePs as cross-linking probes for the investigation of RNA higher-order structure. Nucleic Acids Research. 49 (12), 6660-6672 (2021).
- Sosic, A., et al. Bis-3-chloropiperidines targeting TAR RNA as a novel strategy to impair the HIV-1 nucleocapsid protein. Molecules. 26 (7), 1874 (2021).
- Sosic, A., et al. In vitro evaluation of bis-3-chloropiperidines as RNA modulators targeting TAR and TAR-protein interaction. International Journal of Molecular Sciences. 23 (2), 582 (2022).
- Sosic, A., et al. Direct and topoisomerase II mediated DNA damage by bis-3-chloropiperidines: The importance of being an earnest G. ChemMedChem. 12 (17), 1471-1479 (2017).
- Bock, L. C., Griffin, L. C., Latham, J. A., Vermaas, E. H., Toole, J. J. Selection of single-stranded DNA molecules that bind and inhibit human thrombin. Nature. 355 (6360), 564-566 (1992).
- Paborsky, L. R., McCurdy, S. N., Griffin, L. C., Toole, J. J., Leung, L. L. The single-stranded DNA aptamer-binding site of human thrombin. The Journal of Biological Chemistry. 268 (28), 20808-20811 (1993).
- Carraro, C., et al. Behind the mirror: chirality tunes the reactivity and cytotoxicity of chloropiperidines as potential anticancer agents. ACS Medicinal Chemistry Letters. 10 (4), 552-557 (2019).
- Carraro, C., et al. Appended aromatic moieties in flexible bis-3-chloropiperidines confer tropism against pancreatic cancer cells. ChemMedChem. 16 (5), 860-868 (2021).
- Kypr, J., Kejnovska, I., Renciuk, D., Vorlickova, M. Circular dichroism and conformational polymorphism of DNA. Nucleic Acids Research. 37 (6), 1713-1725 (2009).
- Onel, B., Wu, G., Sun, D., Lin, C., Yang, D. Electrophoretic mobility shift assay and dimethyl sulfate footprinting for characterization of G-quadruplexes and G-quadruplex-protein complexes. Methods in Molecular Biology. 2035, 201-222 (2019).

