In vitro Kemisk kartläggning av G-Quadruplex DNA-strukturer med Bis-3-kloropiperidiner
Summary
Bis-3-kloropiperidiner (B-CePs) är användbara kemiska prober för att identifiera och karakterisera G-quadruplex-strukturer i DNA-mallar in vitro. Detta protokoll beskriver proceduren för att utföra sonderande reaktioner med B-CePs och för att lösa reaktionsprodukter genom högupplösande polyakrylamidgelelektrofores.
Abstract
G-quadruplexer (G4s) är biologiskt relevanta, icke-kanoniska DNA-strukturer som spelar en viktig roll i genuttryck och sjukdomar, och representerar betydande terapeutiska mål. Tillgängliga metoder krävs för in vitro-karakterisering av DNA inom potentiella G-quadruplex-bildande sekvenser (PQS). B-CePs är en klass av alkylerande medel som har visat sig vara användbara kemiska sonder för undersökning av nukleinsyrors högre ordningsstruktur. Denna artikel beskriver en ny kemisk kartläggningsanalys som utnyttjar den specifika reaktiviteten hos B-CePs med N7 av guaniner, följt av direkt strängklyvning vid de alkylerade Gs.
För att skilja G4-veck från ovikta DNA-former använder vi nämligen B-CeP 1 för att undersöka den trombinbindande aptameren (TBA), ett 15-mer-DNA som kan anta G4-arrangemanget. Reaktion mellan B-CeP-reagerande guaniner och B-CeP 1 ger produkter som kan lösas upp genom högupplösande polyakrylamidgelelektrofores (PAGE) på en enda nukleotidnivå genom att lokalisera enskilda alkyleringsaddukter och DNA-strängklyvning vid de alkylerade guaninerna. Kartläggning med hjälp av B-CePs är ett enkelt och kraftfullt verktyg för in vitro-karakterisering av G-quadruplex-bildande DNA-sekvenser, vilket möjliggör exakt lokalisering av guaniner som är involverade i bildandet av G-tetrader.
Introduction
Förutom den typiska Watson-Crick-dubbelhelixen kan nukleinsyror anta olika sekundära strukturer, såsom den alternativa G-quadruplex-formen (G4), på grund av deras guaninrika sekvenser. G4-strukturen är baserad på bildandet av plana tetramerer, kallade G-tetrader, där fyra guaniner interagerar genom Hoogsteen vätebindningar. G-tetrader staplas och stabiliseras ytterligare av monovalenta katjoner som samordnas i mitten av guaninkärnan (figur 1)1.
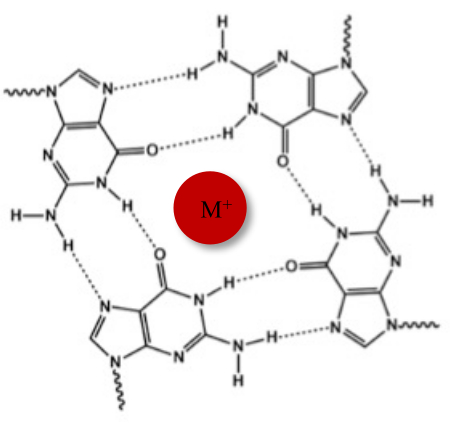
Figur 1: Schematisk representation av en G-quadruplexstruktur. (A) Schematisk representation av en G-tetrad. Den plana matrisen stabiliseras av Hoogsteen-basparning och av en central katjon (M+). Klicka här för att se en större version av denna figur.
Sekvenser med fyra eller fler körningar av minst två på varandra följande guaninnukleotider är potentiella G-quadruplex-bildande sekvenser (PQS) som kan veckas i G-quadruplexstrukturer. PQS finns i många olika cellulära sammanhang, såsom vid telomerer, genpromotorer, ribosomalt DNA och rekombinationsställen, och är involverade i regleringen av många biologiska processer2. Därför är identifiering och experimentell validering av G4s i det mänskliga genomet, som för närvarande främst utförs genom beräkningsverktyg, en biologiskt relevant fråga3. För att stödja beräkningsförutsägelser eller upptäcka oförutsedda G4-strukturer visas här en tillgänglig metod baserad på kemisk kartläggning för att identifiera G4-bildningen i en DNA-mall, vilket möjliggör exakt identifiering av guaniner som bildar G-tetradstrukturen.
Den rapporterade kemiska kartläggningsanalysen utnyttjar den olika reaktiviteten hos bis-3-kloropiperidiner (B-CePs) med guaniner efter bildandet av G4-strukturer. På grund av sin höga reaktivitet med nukleofiler 4,5,6,7,8,9 är B-CeP nukleinsyraalkylerande medel med förmågan att reagera mycket effektivt med N7-positionen hos guaninnukleotider 10. Alkylering följs av avurinering och strängklyvning i enkel- och dubbelsträngade DNA-konstruktioner. Tvärtom är guaniner som är involverade i bildandet av G-tetraderna i G4-arrangemang ogenomträngliga för B-CeP-alkylering, eftersom guaninernas N7-positionär inblandad i Hoogsteen-vätebindningarna. Denna specifika reaktivitet hos B-CePs möjliggör inte bara detektion av G4-strukturer, utan också identifiering av de guaniner som bildar tetraden eller tetraderna, eftersom de kan härledas från deras relativa skydd mot alkylering jämfört med guaniner i enkel- och dubbelsträngat DNA.
Det kemiska kartläggningsprotokollet rapporteras här med användning av B-CeP 1 (Figur 2A) som en sond för karakterisering av trombinbindande aptamer (TBA), ett 15-mer-DNA som kan anta G4-arrangemanget i närvaro av kaliumkatjoner11,12. G4-arrangemanget av TBA (G4-TBA) jämförs direkt med två kontroller, nämligen TBA i enkelsträngad form (ssTBA) och TBA glödgad till sin komplementära sekvens för att bilda den dubbelsträngade konstruktionen (dsTBA) (tabell 1). Produkter av sonderande reaktioner löses upp med högupplösande polyakrylamidgelelektrofores (PAGE) på enkelnukleotidnivå genom att lokalisera enskilda alkyleringsaddukter och DNA-strängklyvning vid de alkylerade guaninerna. Visualisering på gelen möjliggörs genom konjugering av TBA-oligonukleotiden med en fluorofor i dess 3′-ände (tabell 1). Detta protokoll visar hur man viker TBA i dess olika konformationer (G4 och kontroller), och hur man utför sonderingsreaktioner med B-CePs följt av PAGE.
Protocol
Representative Results
Discussion
G-quadruplexer är nukleinsyrasekundära strukturer som vanligtvis veckar sig inom guaninrika DNA-sekvenser och är viktiga forskningsmål på grund av deras koppling till genetisk kontroll och sjukdomar. Kemisk kartläggning med B-CePs är ett användbart protokoll för karakterisering av DNA G4s, som kan användas för att identifiera guaninbaserna som är involverade i bildandet av G-tetrader under fysiologiska saltförhållanden.
Den kemiska sond som används i detta protokoll är B-CeP 1 …
Disclosures
The authors have nothing to disclose.
Acknowledgements
Detta arbete stöddes av Institutionen för farmaceutiska och farmakologiska vetenskaper, University of Padova (PRIDJ-BIRD2019).
Materials
| Acrylamide/bis-acrylamide solution 40% | Applichem | A3658 | R45-46-20/21-25-36/38-43-48/23/ 24/25-62 |
| Ammonium per-sulfate (APS) | Sigma Aldrich | A7460 | |
| Analytical balance | Mettler Toledo | ||
| Autoclave | pbi international | ||
| Boric acid | Sigma Aldrich | B0252 | |
| Bromophenol blue Brilliant blue R | Sigma Aldrich | B0149 | |
| di-Sodium hydrogen phosphate dodecahydrate | Fluka | 71649 | |
| DMSO | Sigma Aldrich | 276855 | |
| DNA oligonucleotides | Integrated DNA Technologies | synthesis of custom sequences | |
| EDTA disodium | Sigma Aldrich | E5134 | |
| Formamide | Fluka | 40248 | H351-360D-373 |
| Gel imager | GE Healtcare | STORM B40 | |
| Glycerol | Sigma Aldrich | G5516 | |
| Micro tubes 0.5 mL | Sarstedt | 72.704 | |
| Potassium Chloride | Sigma Aldrich | P9541 | |
| Sequencing apparatus | Biometra | Model S2 | |
| Silanization solution I | Fluka | 85126 | H225, 314, 318, 336, 304, 400, 410 |
| Sodium phosphate monobasic | Carlo Erba | 480086 | |
| Speedvac concentrator | Thermo Scientific | Savant DNA 120 | |
| TEMED | Fluka | 87689 | R11-21/22-23-34 |
| Tris-HCl | MERCK | 1.08387.2500 | |
| Urea | Sigma Aldrich | 51456 | |
| UV-Vis spectrophotometer | Thermo Scientific | Nanodrop 1000 |
References
- Davis, J. T. G-quartets 40 years later: from 5′-GMP to molecular biology and supramolecular chemistry. Angewandte Chemie. 43 (6), 668-698 (2004).
- Varshney, D., Spiegel, J., Zyner, K., Tannahill, D., Balasubramanian, S. The regulation and functions of DNA and RNA G-quadruplexes. Nature Reviews Molecular Cell Biology. 21 (8), 459-474 (2020).
- Chambers, V. S., et al. High-throughput sequencing of DNA G-quadruplex structures in the human genome. Nature Biotechnology. 33 (8), 877-881 (2015).
- Zuravka, I., Sosic, A., Gatto, B., Gottlich, R. Synthesis and evaluation of a bis-3-chloropiperidine derivative incorporating an anthraquinone pharmacophore. Bioorganic & Medicinal Chemistry Letters. 25 (20), 4606-4609 (2015).
- Zuravka, I., Roesmann, R., Sosic, A., Gottlich, R., Gatto, B. Bis-3-chloropiperidines containing bridging lysine linkers: Influence of side chain structure on DNA alkylating activity. Bioorganic & Medicinal Chemistry. 23 (6), 1241-1250 (2015).
- Zuravka, I., et al. Synthesis and DNA cleavage activity of bis-3-chloropiperidines as alkylating agents. ChemMedChem. 9 (9), 2178-2185 (2014).
- Sosic, A., Gottlich, R., Fabris, D., Gatto, B. B-CePs as cross-linking probes for the investigation of RNA higher-order structure. Nucleic Acids Research. 49 (12), 6660-6672 (2021).
- Sosic, A., et al. Bis-3-chloropiperidines targeting TAR RNA as a novel strategy to impair the HIV-1 nucleocapsid protein. Molecules. 26 (7), 1874 (2021).
- Sosic, A., et al. In vitro evaluation of bis-3-chloropiperidines as RNA modulators targeting TAR and TAR-protein interaction. International Journal of Molecular Sciences. 23 (2), 582 (2022).
- Sosic, A., et al. Direct and topoisomerase II mediated DNA damage by bis-3-chloropiperidines: The importance of being an earnest G. ChemMedChem. 12 (17), 1471-1479 (2017).
- Bock, L. C., Griffin, L. C., Latham, J. A., Vermaas, E. H., Toole, J. J. Selection of single-stranded DNA molecules that bind and inhibit human thrombin. Nature. 355 (6360), 564-566 (1992).
- Paborsky, L. R., McCurdy, S. N., Griffin, L. C., Toole, J. J., Leung, L. L. The single-stranded DNA aptamer-binding site of human thrombin. The Journal of Biological Chemistry. 268 (28), 20808-20811 (1993).
- Carraro, C., et al. Behind the mirror: chirality tunes the reactivity and cytotoxicity of chloropiperidines as potential anticancer agents. ACS Medicinal Chemistry Letters. 10 (4), 552-557 (2019).
- Carraro, C., et al. Appended aromatic moieties in flexible bis-3-chloropiperidines confer tropism against pancreatic cancer cells. ChemMedChem. 16 (5), 860-868 (2021).
- Kypr, J., Kejnovska, I., Renciuk, D., Vorlickova, M. Circular dichroism and conformational polymorphism of DNA. Nucleic Acids Research. 37 (6), 1713-1725 (2009).
- Onel, B., Wu, G., Sun, D., Lin, C., Yang, D. Electrophoretic mobility shift assay and dimethyl sulfate footprinting for characterization of G-quadruplexes and G-quadruplex-protein complexes. Methods in Molecular Biology. 2035, 201-222 (2019).

