تحضير نوى الدماغ ونخاع العظم عالي الجودة لمقايسات النوى المتعددة أحادية النوى
Summary
يعتمد نجاح النسخ أحادي الخلية / أحادي النوى ومتعدد الأوميكس إلى حد كبير على جودة الخلايا / النوى. لذلك ، يجب أن يكون عزل الخلايا / النوى عن الأنسجة وتنقيتها موحدا للغاية. يصف هذا البروتوكول تحضير النوى من الدماغ ونخاع العظم لمقايسة النوى المتعددة أحادية النوى في اتجاه مجرى النهر.
Abstract
أصبح تحليل الخلية الواحدة هو النهج المفضل لكشف تعقيد العمليات البيولوجية التي تتطلب تقييم تباين الاستجابات الخلوية الفردية للعلاج أو العدوى بدقة خلية واحدة.
تم تطوير العديد من التقنيات للتنميط الجزيئي أحادي الخلية على مدى السنوات ال 10 الماضية ، وتم تسويق العديد من التقنيات المخصصة. يعد التنميط أحادي الخلية القائم على قطرات 10X Genomics تقنية واسعة الانتشار توفر كواشف جاهزة للاستخدام للتنميط أحادي الخلية النسخي ومتعدد الأوميك. تتضمن التقنية سير العمل لتسلسل الحمض النووي الريبي أحادي الخلية وأحادي النوى (scRNA-Seq و snRNA-Seq ، على التوالي) ، scATAC-Seq ، التنميط المناعي أحادي الخلية (تسلسل BCR / TCR) ، و multiome. يجمع الأخير بين النسخ (scRNA-Seq) والمعلومات اللاجينية (scATAC-Seq) القادمة من نفس الخلية.
تعد جودة (الجدوى والسلامة والنقاء) للمعلقات أحادية الخلية أو أحادية النوى المعزولة من الأنسجة وتحليلها بواسطة أي من هذه الأساليب أمرا بالغ الأهمية لتوليد بيانات عالية الجودة. لذلك ، يجب تكييف بروتوكولات تحضير العينات مع خصوصيات كل نسيج بيولوجي وضمان توليد معلقات عالية الجودة للخلايا والنوى.
توضح هذه المقالة بروتوكولين لإعداد عينات الدماغ ونخاع العظام لخط أنابيب Multiome 10X Genomics في اتجاه مجرى النهر. يتم تنفيذ البروتوكولات بشكل تدريجي وتغطي تفكك الأنسجة ، وفرز الخلايا ، وعزل النوى ، ومراقبة جودة تعليق النوى المحضر الذي يستخدم كمواد أولية لتقسيم الخلايا والترميز الشريطي وإعداد المكتبة والتسلسل. تنتج هذه البروتوكولات الموحدة مكتبات نوى عالية الجودة وبيانات قوية وموثوقة.
Introduction
لسنوات عديدة ، كانت تقنيات الخلية الواحدة هي المعيار الذهبي لتحليل العمليات البيولوجية. كانت تقتصر في البداية على التنميط الظاهري أحادي الخلية من خلال الفحص المجهري وقياس التدفق الخلوي والمقايسات المماثلة. جاء اختراق في تحليل الخلية الواحدة مع تطوير مناهج التنميط الجزيئي أحادي الخلية ، ولا سيما تسلسل الحمض النووي الريبي أحادي الخلية (scRNA-Seq) الذي مكن من توصيف النسخ الكامل للخلايا الفردية. قوي للغاية ، يولد scRNA-Seq معلومات حول حالة النسخ للخلية في حالة ونقطة زمنية معينة. ومع ذلك ، فإنه لا يوفر رؤية حول تنظيم الجينات الذي يدفع النسخ ، أو على التعديلات الجزيئية التي تحدث بمرور الوقت. للتغلب على هذا القيد ، تم استثمار العديد من الجهود في تطوير مقايسات متعددة الخلايا أحادية الخلية تمكن من تحليل عوامل وعمليات متعددة من نفس الخلية1،2،3،4. جاء أول قياس ناجح لطريقتين داخل الخلايا المفردة من خلال ربط أنماط تعبير البروتين السطحي المتعدد مع النسخ الكامل للخلايا الفردية في نهج CITE-Seq5. تجمع التطورات الحديثة بين التعبير الجيني وإمكانية الوصول إلى الكروماتين (فحص الكروماتين الذي يمكن الوصول إليه باستخدام التسلسل ، ATAC-Seq) ، وبالتالي التقاط طرائق النسخ وفوق الجينوم في نفس الخلايا في نفس الخلايا (على سبيل المثال ، sci-CAR) 6. جاءت الحلول التجارية الأولى التي سمحت بربط النسخ بالنمط الظاهري للخلية أو بالتغيرات اللاجينية لنفس الخلية من 10X Genomics.
تحتوي تجارب التنميط الجزيئي أحادي الخلية على الخطوات التالية: (1) تفكك الأنسجة أو تحضير معلقات أحادية الخلية. (2) تنقية الخلايا و / أو عزل النوى ؛ (3) التقسيم والترميز الشريطي ؛ (4) بناء المكتبة ومراقبة الجودة ؛ (5) تسلسل الجيل التالي ؛ (6) تحليل البيانات. في حين أن الخطوات (3) – (6) قد تختلف اختلافا كبيرا اعتمادا على التكنولوجيا المستخدمة ، فإن الخطوات الأولية مشتركة بشكل عام مع كل منهم. ستحدد جودة تعليق الخلية / النوى المحضرة النتيجة الإجمالية للتجربة. اعتمادا على نوع الأنسجة ، قد يكون الحصول على معلقات أحادية الخلية / نوى عالية الجودة أمرا صعبا. تتطلب خصوصيات بعض الأنسجة ، مثل القلب والعضلات والدماغ والرئة والأمعاء وغيرها ، طرقا لتعطيل الأنسجة وعزل النوى تتكيف مع كل نوع من العينات من أجل ضمان إنتاج نوى عالية الجودة للتحليل الجزيئي7،8،9،10. يمكن أن تكون طرق تعطيل الأنسجة وبروتوكولات التفكك ميكانيكية أو إنزيمية (على سبيل المثال ، مزيج من الكولاجين و DNase) ، أو مزيج من الاثنين ، ويمكن إجراؤها يدويا أو بواسطة أدوات (على سبيل المثال ، Qiagen DSC-400 ، gentleMACS).
أصبحت تقنيات الخلية الواحدة أداة مفضلة للبحوث الطبية الحيوية. في علم الأعصاب ، يتطلب تنوع الخلايا في الدماغ وتعقيد وظائفها تحليلا عالي الدقة وعالي الإنتاجية لتصور مجموعات الخلايا النادرة ولتقييم عدم تجانسها11،12،13،14. يوفر ربط الهوية الخلوية والآليات التنظيمية الجينية للخلايا الفردية نظرة ثاقبة لنمو الدماغ وعلم وظائف الأعضاء. مثال آخر هو دراسات الاستجابة المناعية في سياق الأمراض المعدية أو المناعة الذاتية أو السرطان ، والتي تعتمد بشدة على تحليلات الخلية الواحدة. يتطلب عدم تجانس المجموعات الفرعية للخلايا المناعية وتعقيد نشاطها وتفاعلاتها مع أنواع الخلايا الأخرى دقة خلية واحدة في فك رموز الآليات الكامنة وراء الاستجابة المناعية. تنشأ الخلايا المناعية من نخاع العظم ، حيث تتكون الأسلاف المكونة للدم من خلايا متمايزة تدريجيا تكتسب وتفقد علامات سطح الخلية خلال عملية تدريجية قبل الخروج من نخاع العظم إلى المنزل في المحيط. يسمح تحليل الخلية الواحدة بالتوصيف الدقيق لمراحل النمو الخلوي. يمكن تحقيق ذلك من خلال التنميط الظاهري أحادي الخلية ، والذي يتم إجراؤه تقليديا بواسطة قياس التدفق الخلوي متعدد المعلمات. ومع ذلك ، فقد ثبت أن التوقيعات النسخية أحادية الخلية تكشف عن تحديد أكثر دقة للأنواع الفرعية للخلايا السلفية نظرا لأن هذه الخلايا موزعة في مجموعات تقع في بعضها البعض وبالتالي يمكن التعرف عليها بشكل خاطئ عند استخدام نهج علامة سطح الخليةالخشنة 15. يكشف عدد متزايد من الدراسات عن التعديلات اللاجينية التي يمكن أن تكتسبها الخلايا الجذعية والسلفية المكونة للدم (HSPCs) من التعرض لعوامل مختلفة ، مما يؤدي إلى تأثير كبير على الاستجابة طويلة المدى لجهاز المناعة16،17،18،19. تتيح تقنيات multi-omics الجديدة دراسة هذه العمليات بدقة خلية واحدة.
تم وصف العديد من بروتوكولات عزل الخلايا والنوى للدماغ11،20،21،22 وعينات نخاع العظم23،24. لتقليل التحيز بسبب التباين التجريبي ، من الضروري التحقق من صحة بروتوكولات تحضير النوى المفردة المحسنة للتسلسل النسخي وفوق الجيني المشترك أحادي الخلية ، وبالتالي ضمان قابلية استنساخ المقايسات متعددة الخلايا أحادية الخلية.
هنا ، يتم وصف بروتوكولين قويين لإعداد النوى من (1) أنسجة المخ المجمدة الطازجة و (2) HSPCs لنخاع العظم الطازج لتحليل Multiome أحادي الخلية في المصب (الشكل 1).
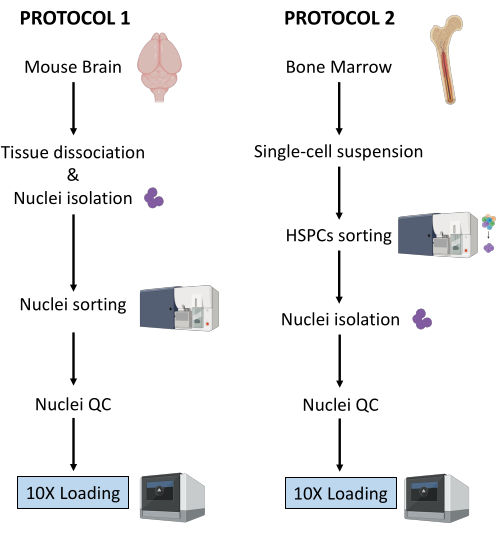
الشكل 1: تمثيل تخطيطي لبروتوكولات عزل النوى من أنسجة الدماغ ونخاع العظم المجمدة حديثا. يرجى النقر هنا لعرض نسخة أكبر من هذا الشكل.
Protocol
Representative Results
Discussion
يعد تحضير معلق خلية أو نوى عالي الجودة ذا أهمية حاسمة لنجاح تحليل RNA-Seq أحادي الخلية أو أحادي النوىوتحليلات متعددة الخلايا أحادية الخلية 29،30،31. هنا ، وصفنا بروتوكولات تحضير العينات وعزل النوى لمقايسات multiome من نوعين من الأنسجة: الدماغ ونخاع العظام.
يسمح بروتوكول الدماغ الموصوف في هذه الورقة باستعادة نوى عالية الجودة من أنسجة المخ الطازجة المجمدة. ويشمل الخطوات التالية: تعطيل الأنسجة المجمدة ، وعزل النوى ، وتنقية النوى ، ومراقبة جودة المواد المحضرة. يتكون نسيج المخ من العديد من أنواع الخلايا المختلفة ، ويجب أن يحافظ إجراء تفكك الأنسجة وعزل النوى على نسب مجموعات الخلايا الموجودة في الأنسجة الأولية. هنا ، تم تحسين تكوين المخزن المؤقت للتحلل ووقت الحضانة لتمكين التحلل الكامل واللطيف لجميع مجموعات الخلايا التي تشكل الأنسجة.
يختلف بروتوكول HSPCs لنخاع العظم إلى حد ما لأنه يتطلب خطوة إضافية واحدة في بداية التجربة لعزل مجموعة الخلايا ذات الأهمية من تعليق خلوي غير متجانس. بعد جمع الأنسجة الطازجة ، يتم تحليل خلايا الدم الحمراء ، ويتم إثراء العينة لمجموعة فرعية من الخلايا ذات الأهمية. يتم تحليل الخلايا المستهدفة ، ويتم عزل النوى ، ويتم التحكم في جودة المواد المعدة.
يوفر 10X Genomics العديد من البروتوكولات التي تم التحقق من صحتها لعزل النوى في العديد من الأنسجة المختلفة32,33. تقوم الشركة أيضا بتسويق مجموعة عزل النوى مع خط أنابيب مباشر لعزل النوى من الأنسجة التي تم التحقق من صحتها34. ومع ذلك ، تحتاج هذه البروتوكولات إلى تحسين إضافي لتكييف خصوصيات عينات معينة. مثال على ذلك هو العينات التي تتطلب العمل مع إدخال خلية منخفضة. بالنسبة لهذه العينات ، فإن الخطوات الأكثر تحديا هي أجهزة الطرد المركزي التي يجب أن تكون صارمة بما يكفي لتنظيف العينة ولطيفة بما يكفي لتجنب فقدان الخلية / النوى. باستخدام البروتوكول الموضح هنا ، قمنا بتكييف بروتوكول 10X Genomics الموضح – عزل النوى لتسلسل ATAC + GEX متعدد الخلية أحادي الخلية (CG000365 – Rev C) 27 لإيجاد توازن دقيق بين هذين المتطلبين. كما هو موضح في مثال تحضير النوى من HSPCs المصنفة ، قمنا بتحسين استعادة النوى دون أي تأثير على جودة العينة.
التحدي الإضافي هو خطوة تحلل الخلايا النقية لعزل النوى. يمكن أن تؤدي ظروف التحلل الأكثر قسوة وأوقات الحضانة الأطول إلى إتلاف النوى وبالتالي التأثير على جودة بيانات التسلسل. يوضح الشكل 5 تصوير النوى التمثيلية من عينات نخاع العظم في أوقات حضانة مختلفة باستخدام مخزن التحلل، ويوضح مدى اختلاف حالة النوى اعتمادا على تحلل الخلية. في مثال HSPCs ، حددنا تحلل 3 دقائق كحالة تؤدي إلى أعلى نسبة من النوى السليمة ذات المظهر الصحي وأقل نسبة من النوى التالفة. يجب تحسين أوقات حضانة التحلل لكل نوع جديد من العينات.
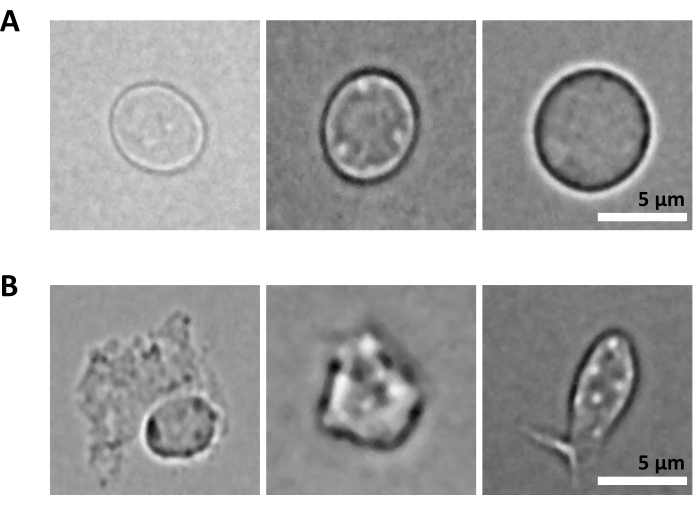
الشكل 5: مراقبة جودة النوى عن طريق الفحص المجهري. تظهر صور برايت فيلد تمثيلية لنوى معزولة من نخاع عظم الفأر مع (أ) سليمة و (ب) نوى تالفة. مقياس الرسم 5 ميكرومتر. تم التقاط الصور باستخدام مجهر مقلوب باستخدام هدف 40x ELWD NA 0.60 وتقريب رقمي 1.5x. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
يعتمد كلا البروتوكولين المفصلين في هذا العمل على تنقية الخلايا أو النوى المستهدفة بواسطة أدوات FACS عالية الإنتاجية. هذه الخطوة ذات أهمية حاسمة لبروتوكولات تحضير الخلية الواحدة / النوى حيث يتم عزل مجموعات فرعية نادرة من الخلايا من المعلقات غير المتجانسة. في هذه ، كما هو الحال في المثال الموضح هنا لفرز HSPCs ، قد تكون هناك حاجة إلى لوحة قياس التدفق الخلوي عالية الأبعاد لتمكين “البوابة” على مجموعة الخلايا ذات الأهمية. الفرز سريع ودقيق للغاية ، مما يؤدي إلى نقاء أكثر من 95٪ من المجموعات الفرعية للخلايا التي تم فرزها. يعرض هذا النهج التعليق الخلوي لضغط يصل إلى 70 رطل لكل بوصة مربعة ، وبالتالي قد يكون مقيدا لفرز الخلايا الهشة (على سبيل المثال ، الخلايا المتغصنة ، العدلات) لأنه قد يتسبب في تمزق غشاء الخلية. وفي هذه الحالات، ينبغي اختيار حلول بديلة للتنقية الخلوية، بما في ذلك الفرز المغناطيسي، وتطبيق أدوات الجيل الجديد (مثل CellenOne، وCellenion؛ وCellenion؛ وCellenion). MACSQuant Tyto، Miltenyi)35,36، أو الأنظمة القائمة على القطيرات (على سبيل المثال، ODIN، Sensific)37. ومع ذلك ، فإن سرعة الفرز البطيئة لهذه التقنيات ، مع فرز الخلايا الذي يستمر لساعات بدلا من دقائق ، هو عامل مقيد قوي لتطبيق هذه الأساليب في إعداد خلايا قابلة للحياة ل Multiome وغيرها من تطبيقات الخلية المفردة بناء على تحليل أعداد الخلايا الكبيرة.
لتنقية النوى المعزولة من الأنسجة ، FACS هي الطريقة المفضلة بسبب إنتاجيتها ونقاء المادة المعزولة. النوى ليست حساسة للضغط ، ويمكن بسهولة تنقية عزلات الأنسجة المفلترة من خلال فارز الخلايا. وإذا لم يكن المختبر مجهزا بأداة من أدوات نظام مراقبة الأصول الميدانية، توجد بدائل أخرى، أقل كفاءة إلى حد ما ولكنها جيدة بما فيه الكفاية. ومن الأمثلة على ذلك الطرد المركزي الفائق أو استخدام معدات صغيرة مثل MARS (الخلية التطبيقية) التي تفصل الجسيمات بناء على اختلافها في الحجم ، باستخدام الموجات الصوتية. غسالة CURIOX الصفائحية التي تستخدم خصائص كارهة للماء لمعلقات الخلية / النوى ؛ أو LEVITAS bio الذي يعتمد على الخصائص الفيزيائية للخلايا (الرفع) لفصلها عن الحطام.
هنا ، نصف البروتوكولات للحصول على عدد كبير من النوى وأفضل نقاء لبروتوكول Multiome النهائي. ويؤدي فرز نظام مراقبة الأصول الميدانية وخطوات الطرد المركزي المتكررة إلى خسارة كبيرة في المواد الأولية. لهذا السبب ، في بروتوكول تحضير النوى من الدماغ الذي نصفه هنا يتطلب مواد أولية وفيرة بما فيه الكفاية لينتج عنها جمع ما لا يقل عن 500000 نواة بعد فرز FACS. يجب تطبيق بروتوكولات بديلة إذا لم يكن من الممكن مطابقة هذا المعيار. عند العمل مع مجموعات الخلايا النادرة أو أقسام الأنسجة الصغيرة ، يمكن أن تكون الكمية المتاحة من المواد الأولية عاملا مقيدا. لمعالجة هذه المشكلة ، من الممكن تحسين استعادة النوى عن طريق (أ) تقليل حجم التحلل ، (ب) تقليل حجم الغسيل ، (ج) استخدام غسلة واحدة مع وقت طرد مركزي ممتد لمحاولة تحسين الاسترداد كما هو موضح في بروتوكولات الجينوم 10X لعزل نوى مدخلات الخلايا المنخفضة. بالنسبة للتحليل متعدد الألوان للمواد منخفضة المحتوى ، يجدر النظر في التطبيقات القائمة على الألواح مثل scNMT و SNARE-seq و Paired-seq38 التي تتطلب عينات إدخال أقل بكثير.
باختصار ، لقد وصفنا بروتوكولين قويين لإعداد النوى من الدماغ ونخاع العظم HSPCs لتحليل Multiome في اتجاه مجرى النهر. هذه البروتوكولات قابلة للتطبيق في أي مشروع علمي يتطلب معلقات نوى مفردة عالية الجودة من هذين النوعين من الأنسجة ، بغض النظر عن السؤال العلمي المطروح. تقوم مجموعتنا بتطبيق بروتوكول عزل نوى الدماغ في دراسات نمو الدماغ عند تعطيل مختلف الجينات المستهدفة وفي دراسات الاستجابة المناعية في سياق الأمراض العصبية. نحن نستخدم بروتوكول عزل نوى نخاع العظم لفك رموز مشاركة مختلف المجموعات السكانية الفرعية المكونة للدم في إنشاء جهاز المناعة.
Disclosures
The authors have nothing to disclose.
Acknowledgements
حصلت آنا جيمين تشوي على دعم من راتب من برنامج الدكتوراه الدولي بجامعة باستور – باريس (PPU).
Materials
| 18 G x 1 ½ (1.2 mm x 38 mm) Agani needles | Terumo | AN*1838S1 | |
| 15 mL tubes | Falcon | 352097 | |
| 5 mL round bottom FACS tube with cell strainer cap 35 µm | falcon | 352235 | |
| 50 mL tubes | Falcon | 352070 | |
| 7-AAD | BD pharmagen | 559925 | |
| ACK Lysing Buffer | Gibco | A10492-01 | |
| APC anti-mouse CD117 (c-Kit) | BioLegend | 105812 | Clone: 2B8 |
| APC/Cyanine 7 anti-mouse CD16/32 (FcγR) | BioLegend | 101328 | Clone: 93 |
| BD FACSAria III | BD Biosciences | non-applicable | |
| BD FACSDiva Software v8.0.1 | BD Biosciences | non-applicable | |
| Bovine Serum Albumin stock solution 10% | Miltenyi Biotec | 130-091-376 | |
| Cell staining buffer | Biolegend | 420201 | |
| CFI Suprplan Fluor ELWD 40XC ON 0.6 | Nikon | non-applicable | |
| CMOS camera Prime 95B 25 mm | Photometrix | non-applicable | |
| Countess II FL Automated Cell Counter | Invitrogen | AMQAF1000 | |
| Countess cell counting chamber slide | Invitrogen | C10283 | |
| Coverglass 24 mm x 24 mm 0.13-0.17 mm | Brand | BR470819 | |
| Digitonine 5% | Invitrogen | BN2006 | |
| Disposable Scalpels | Swann-Morton | 0508 | |
| DMEM (1x) + GlutaMAX-I | Gibco | 31966-021 | |
| DPBS (10x) | Gibco | 14200-067 | |
| DTT | Sigma aldrich | 646563 | |
| Epifluorescence inverted microscope Nikon Ti2 -E | Nikon | non-applicable | |
| Eppendorf Safe-Lock Tubes 0.5 mL | Eppendorf | 30123603 | |
| Ethanol 70% | VWR | 83801.290 | |
| FITC anti-mouse CD34 | Invitrogen | 11-0341-85 | Clone: RAM34 |
| Forceps for dissection | FST | 11152-10 | |
| Heat-inactivated Fetal Bovine Serum (FBS) | Gibco | 11533387 | |
| Dounce Homogeniser 2 mL | Bellco glass | 1984-10002 | Pestle “A” Large Clearance: .0030-.0050″ and Pestle “B” Small Clearance: .0005-.0025″ |
| LIVE/DEAD fixable aqua dead cell stain kit | Invitrogen | L34957 | |
| Magnesium chloride solution 1 M | Sigma aldrich | M1028 | |
| Microcentrifuge | Eppendorf | 5424R | |
| Mounting medium Fluoromount-G | invitrogen | 00-4958-02 | |
| Nonidet P40 substitute | Sigma aldrich | 74385 | |
| Nuclease free water | ThermoFischer | AM9932 | |
| Nuclei buffer 20x | 10X Genomics | 2000153/2000207 | |
| Nuclei isolation kit EZ prep | Sigma Aldrich | NUC-101 | |
| OneComp eBeads compensation beads | Invitrogen | 01-1111-41 | |
| Pacific Blue anti-mouse lineage cocktail (including anti-mouse CD3, Ly-6G/Ly-6C, CD11b, CD45R/B220, TER-119) |
BioLegend | 133310 | Clones (in the same order as the antibodies listed): 17A2, RB6-8C5, M1/70, RA3-6B2, Ter-119 |
| PCR Tube Strips 0.2 mL | Eppendorf | 951010022 | |
| PE anti-mouse Ly-6A/E (Sca-1) | BioLegend | 122507 | Clone: E13-161.7 |
| Petri dish 100 mm x 20 mm OPTILUX | Falcon | 353003 | |
| Ply-L-lysine 0.01% sterile-filtered suitable for cell culture | Sigma | P4707 | |
| Printed microscope slides 8 well 6 mm numbered | Epredia | ER-301B-CE24 | |
| Protein LoBind Tubes 1.5 mL | Eppendorf | 30108116 | |
| Recombinant Rnase inhibitor 5000 U | Takara | 2313A | |
| Scissors for dissection | FST | 14090-09 | |
| Sodium chloride solution 5 M | Sigma aldrich | 59222C | |
| Syringe filters, PES, 0.2 µm | Fisher Scientific | 15206869 | |
| Transparent nail polish | any | non-applicable | |
| Trizma Hydrochloride solution pH 7.4 | Sigma aldrich | T2194 | |
| Trypan Blue 0.4% | gibco | 15250061 | |
| Tween 20 | Biorad | 1662404 | |
| UltraPure Distilated Water Dnase/Rnase Free | Invitrogen | 10977-035 |
References
- Clark, S. J., et al. scNMT-seq enables joint profiling of chromatin accessibility DNA methylation and transcription in single cells. Nature Communications. 9 (1), 781 (2018).
- Lee, J., Hyeon, D. Y., Hwang, D. Single-cell multiomics: technologies and data analysis methods. Experimental & Molecular Medicine. 52 (9), 1428-1442 (2020).
- Cerrizuela, S., et al. High-throughput scNMT protocol for multiomics profiling of single cells from mouse brain and pancreatic organoids. STAR Protocols. 3 (3), 101555 (2022).
- Dimitriu, M. A., Lazar-Contes, I., Roszkowski, M., Mansuy, I. M. Single-cell multiomics techniques: From conception to applications. Frontiers in Cell and Developmental Biology. 10, 854317 (2022).
- Stoeckius, M., et al. Simultaneous epitope and transcriptome measurement in single cells. Nature Methods. 14 (9), 865-868 (2017).
- Cao, J., et al. . Joint profiling of chromatin accessibility and gene expression in thousands of single cells. 361 (6409), 1380-1385 (2018).
- Narayanan, A., et al. Nuclei Isolation from Fresh Frozen Brain Tumors for Single-Nucleus RNA-seq and ATAC-seq.Journal of Visualized Experiments. JoVE. 162, 61542 (2020).
- Kim, M., et al. Single-nucleus transcriptomics reveals functional compartmentalization in syncytial skeletal muscle cells. Nature Communications. 11 (1), 6375 (2020).
- Santos, M. D., et al. Extraction and sequencing of single nuclei from murine skeletal muscles. STAR Protocols. 2 (3), 100694 (2021).
- Safabakhsh, S., et al. Isolating nuclei from frozen human heart tissue for single-nucleus RNA sequencing. Current Protocols. 2 (7), (2022).
- Lau, S. -. F., Cao, H., Fu, A. K. Y., Ip, N. Y. Single-nucleus transcriptome analysis reveals dysregulation of angiogenic endothelial cells and neuroprotective glia in Alzheimer’s disease. Proceedings of the National Academy of Sciences of the United States of America. 117 (41), 25800-25809 (2020).
- Armand, E. J., Li, J., Xie, F., Luo, C., Mukamel, E. A. Single-cell sequencing of brain cell transcriptomes and epigenomes. Neuron. 109 (1), 11-26 (2021).
- Morabito, S., et al. Single-nucleus chromatin accessibility and transcriptomic characterization of Alzheimer’s disease. Nature Genetics. 53 (8), 1143-1155 (2021).
- Chen, S., et al. Spatially resolved transcriptomics reveals genes associated with the vulnerability of middle temporal gyrus in Alzheimer’s disease. Acta Neuropathologica Communications. 10 (1), (2022).
- Paul, F., et al. Transcriptional heterogeneity and lineage commitment in myeloid progenitors. Cell. 163 (7), 1663-1677 (2015).
- Kaufmann, E., et al. BCG educates hematopoietic stem cells to generate protective innate immunity against tuberculosis. Cell. 172 (1-2), 176-190 (2018).
- Christ, A., et al. diet triggers NLRP3-dependent innate immune reprogramming. Cell. 172 (1-2), 162-175 (2018).
- Moorlag, S. J. C. F. M., et al. β-Glucan Induces protective trained immunity against mycobacterium tuberculosis infection: A key role for IL-1. Cell Reports. 31 (7), 107634 (2020).
- de Laval, B., et al. C/EBPβ-dependent epigenetic memory induces trained immunity in hematopoietic stem cells. Cell Stem Cell. 26 (5), 657-674 (2020).
- Renthal, W., et al. Characterization of human mosaic Rett syndrome brain tissue by single-nucleus RNA sequencing. Nature Neuroscience. 21 (12), 1670-1679 (2018).
- Yang, A. C., et al. A human brain vascular atlas reveals diverse mediators of Alzheimer’s risk. Nature. 603 (7903), 885-892 (2022).
- Lee, D. R., Zhang, Y., Rhodes, C. T., Petros, T. J. Generation of single-cell and single-nuclei suspensions from embryonic and adult mouse brains. STAR Protocols. 4 (1), 101944 (2022).
- Corces, M. R., et al. Lineage-specific and single-cell chromatin accessibility charts human hematopoiesis and leukemia evolution. Nature Genetics. 48 (10), 1193-1203 (2016).
- Ranzoni, A. M., et al. Integrative single-cell RNA-seq and ATAC-seq analysis of human developmental hematopoiesis. Cell Stem Cell. 28 (3), 472-487 (2021).
- . 10X Genomics Chromium Next GEM Single Cell Multiome ATAC + Gene Expression User Guide. Document Number CG000338 Rev F. At. , (2022).
- J, , et al. Microdissection of mouse brain into functionally and anatomically different regions. Journal of Visualized Experiments: JoVE. 168, 61941 (2021).
- . . 10X Genomics 10X Genomics Demonstrated Protocol – Nuclei Isolation for Single Cell Multiome ATAC + GEX sequencing (CG000365 – Rev C). At. , (2022).
- Haag, S., Murthy, A. Murine monocyte and macrophage culture. Bio-Protocol. 11 (6), (2021).
- Haque, A., Engel, J., Teichmann, S. A., Lönnberg, T. A practical guide to single-cell RNA-sequencing for biomedical research and clinical applications. Genome Medicine. 9 (1), (2017).
- Jiang, P. Quality control of single-cell RNA-seq. Methods in Molecular Biology. , 1-9 (1935).
- Regan, C., Preall, J. Practical considerations for single-cell genomics. Current Protocols. 2 (8), (2022).
- . . 10X Genomics 10X Genomics Demonstrated Protocol – Nuclei Isolation for Single Cell ATAC Sequencing (CG000169 – Rev E). At. , (2022).
- . 10X Genomics 10X Genomics Demonstrated Protocol – Nuclei Isolation from Complex Tissues for Single Cell Multiome. ATAC + Gene Expression Sequencing. (CG000375 – Rev C). At. , (2022).
- . . 10X Genomics 10X Genomics – Chromium Nuclei Isolation Kit (CG000505 – Rev A). AT. , (2022).
- Shomroni, O., et al. A novel single-cell RNA-sequencing approach and its applicability connecting genotype to phenotype in ageing disease. Scientific Reports. 12 (1), 4091 (2022).
- Ocañas, S. R., Pham, K. D., Blankenship, H. E., Machalinski, A. H., Chucair-Elliott, A. J., Freeman, W. M. Minimizing the ex vivo confounds of cell-isolation techniques on transcriptomic and translatomic profiles of purified microglia. 9 (2), 0348-0321 (2022).
- Gérard, A., et al. High-throughput single-cell activity-based screening and sequencing of antibodies using droplet microfluidics. Nature Biotechnology. 38 (6), 715-721 (2020).
- Vandereyken, K., Sifrim, A., Thienpont, B., Voet, T. Methods and applications for single-cell and spatial multi-omics. Nature Reviews. Genetics. 24, 494-515 (2023).

