מיפוי הגנום של תרופות-DNA בתאי אינטראקציות עם COSMIC (Crosslinking של מולקולות קטנות לבודד הכרומטין)
Summary
זיהוי המטרות הישירות של מולקולות מיקוד הגנום עדיין מהווה אתגר גדול. כדי להבין כיצד מולקולות ה- DNA מחייבת לעסוק הגנום, פיתחנו שיטה המסתמכת על crosslinking של מולקולות קטנות לבודד את הכרומטין (קוסמי).
Abstract
הגנום הוא היעד של חלק מchemotherapeutics היעיל ביותר, אבל רוב התרופות האלה חסרות ייחוד רצף ה- DNA, מה שמוביל לרעילות ורבות תופעות לוואי שליליות הגבלת מינון. מיקוד הגנום עם מולקולות קטנות רצף ספציפי עשויה לאפשר מולקולות עם מדד טיפולי מוגבר ופחות השפעות חוץ-היעד. -methylpyrrole N / מפוליאמידים -methylimidazole N הם מולקולות שיכולים להיות מתוכנן בצורה רציונלית למקד רצפי DNA ספציפיים בדייקנות מופלאה. ושלא כמו גורמי שעתוק הטבעיים ביותר, מפוליאמידים יכולים להיקשר ל- DNA מפוגל וchromatinized ללא הפסד בזיקה. הסגוליות הרצף של מפוליאמידים נחקר בהרחבה במבחנה עם זיהוי אתרו מקור (CSI) ועם גישות ביוכימיות וbiophysical מסורתיות, אבל המחקר של פוליאמיד מחייב מטרות הגנומי בתאים נותר חמקמק. כאן אנו מדווחים על שיטה, crosslinking של מולקולות קטנות לאיזולההכרומטין te (קוסמי), המזהה אתרי פוליאמיד מחייב על פני הגנום. COSMIC דומה לimmunoprecipitation הכרומטין (שבב), אך שונה בשתי דרכים חשובות: (1) photocrosslinker מועסק כדי לאפשר לכידה סלקטיבית, באופן זמני בשליטה של אירועים מחייבים פוליאמיד, ו- (2) ידית זיקת ביוטין משמשת כדי לטהר פוליאמיד conjugates -DNA בתנאי denaturing-למחצה כדי להקטין את ה- DNA שהוא אינן מחויב קוולנטית. קוסמי הוא אסטרטגיה כללית שניתן להשתמש בי כדי לחשוף את האירועים מחייבים הגנום של מפוליאמידים ותרופות כימותרפיות מיקוד הגנום אחרים.
Introduction
המידע להפוך כל תא בגוף האדם מקודד בדנ"א. השימוש סלקטיבי במידע שמסדיר את גורלו של תא. גורמי שעתוק (TFS) הם חלבונים שנקשרים לרצפי DNA ספציפיים להביע משנה מסוים של גנים בגנום, והתקלה של TFS צמודה לתחילתה של מגוון רחב של מחלות, כולל פגמים התפתחותיים, סרטן, סוכרת ו . 1,2 אנחנו כבר מתעניינים בפיתוח מולקולות שיכולות באופן סלקטיבי להיקשר לגנום ולווסת רשתות רגולציה גן.
Polyamides מורכבת מ-methylpyrrole N ו- N -methylimidazole הם מעוצבים באופן רציונלי מולקולות שיכולים למקד את ה- DNA עם ספציפיות וזיקות שגורמים יריבים טבעיים שעתוק. 3-6 מולקולות אלה נקשרים לרצפים ספציפיים בחריץ הקטן של ה- DNA. 4,5,7 -11 Polyamides להיות מועסק לשני להדחיק ולהפעיל את הביטוי של ז הספציפיEnes. 4,12-19 יש להם גם תכונות אנטי 12,13,25-30 20-24 ואנטי סרטניות מעניינות. תכונה מושכת אחד מפוליאמידים היא היכולת שלהם כדי לגשת לרצפי DNA המפוגלים 31,32 ועוטפים את חלבוני היסטון 9,10,33.
כדי למדוד את ספציפיות מחייבות המקיפה של מולקולות ה- DNA מחייבת, המעבדה שלנו יצרה מזהה אתרו מקור השיטה (CSI). 34-39 ההתרחשות הצפויה של אתרי קישור המבוסס על במבחנה ספציפיות (genomescapes) יכול להיות מוצג בגנום, כי במבחנה עוצמות מחייבות הן ביחס ישר לקבועי עמותה (K). 34,35,37 genomescapes אלה מספק תובנה תפוסת פוליאמיד פני הגנום, אבל פוליאמיד מדידה מחייב בתאים חיים כבר אתגר. ה- DNA ארוז היטב בגרעין, שעלול להשפיע על הנגישות של אתרי קישור.ccessibility של רצפי DNA chromatinized אלה למפוליאמידים נשאר בגדר תעלומה.
לאחרונה, שיטות רבות ללמוד אינטראקציות בין מולקולות קטנות וחומצות גרעין צמחו. 40-48 הלכידה הכימית הזיקה ורצפי DNA מקבילים מסיבי (Chem-seq) הוא טכניקה אחת כזו. Chem-seq משתמש פורמלדהיד לcrosslink מולקולות קטנות ליעד הגנומי של ריבית ונגזרת biotinylated של מולקולה קטנה של עניין כדי ללכוד את האינטראקציה יגנד-היעד. 48,49
crosslinking פורמלדהיד מוביל לאינטראקציות עקיפות שיכול לייצר תוצאות חיוביות שגויות. 50 פיתחנו שיטה חדשה, crosslinking של מולקולות קטנות לבודד את הכרומטין (קוסמי), 51 עם photocrosslinker לחסל פסגות אלה מה שנקרא "פנטום". 50 כדי להתחיל, עיצבנו ומסונתז נגזרי trifunctional של מפוליאמידים. מולקולות אלה הכילו Pol ה- DNA מחייבתyamide, photocrosslinker (psoralen), וידית זיקה (ביוטין, איור 1). עם מפוליאמידים trifunctional, אנחנו יכולים קוולנטית ללכוד אינטראקציות פוליאמיד-DNA עם קרינת UV 365 ננומטר, אורך גל שאינו לגרום נזק לדנ"א או לגרום crosslinking-לא-psoralen מבוססת. 51 הבא, אנו מפצלים את הגנום ולטהר את ה- DNA שנתפס תחת מחמיר, חצי תנאי -denaturing להקטין DNA שהוא אינן מחויב קוולנטית. לפיכך, אנו רואים COSMIC כשיטה הקשורים לכימיה-seq, אבל עם קריאת נתונים ישירים יותר של ה- DNA מיקוד. חשוב לציין, החלש (K 10 3 -10 4 M -1) זיקה של psoralen לDNA לא במובחן, פוליאמיד השפעה סגוליות. 51,52 ניתן לנתח קטעי DNA המועשר או על ידי תגובת השרשרת של פולימראז כמותיים 51 (קוסמי qPCR) או על ידי רצף 53 (COSMIC-seq) של הדור הבא. נתונים אלה מאפשרים עיצוב של ligands משוחד, מודרך הגנום שביןלפעול עם הלוקוסים הגנומי הרצויים שלהם ולמזער את ההשפעות חוץ-היעד.
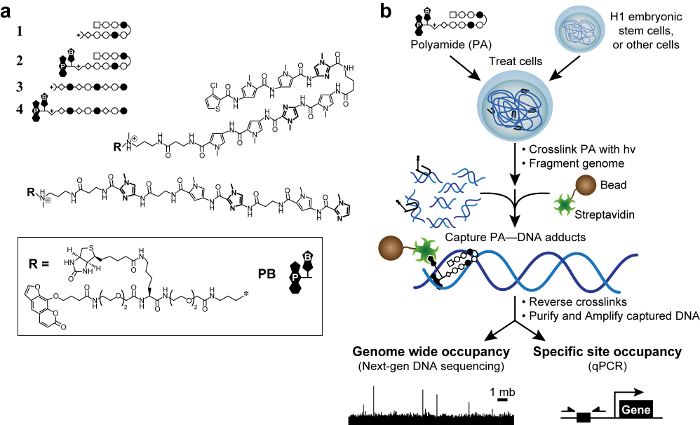
איור 1. מפוליאמידים ביו ותכנית קוסמית. () מפוליאמידים מכבנה 1 – 2 יעד רצף ה- DNA 5'-WACGTW-3 ". מפוליאמידים יניארי 3 – 4 יעד 5'-AAGAAGAAG-3 ". טבעות של N-methylimidazole מודגשות לבהירות. חוגים פתוחים ומילאו לייצג -methylpyrrole N ו-methylimidazole N, בהתאמה. הכיכר מייצגת 3-chlorothiophene, ויהלומים מייצגים β-אלאנין. Psoralen וביוטין מסומנים על ידי P ו- B, בהתאמה. תכנית קוסמית (B). תאי מטופלים עם נגזרי trifunctional של מפוליאמידים. לאחר crosslinking עם קרינת UV 365 ננומטר, תאים LDNA ysed והגנומי הוא טעון. חרוזים מגנטיים streptavidin המצופה מתווספים ללכוד adducts פוליאמיד-DNA. ה- DNA משתחרר ויכול להיות מנותחת על ידי PCR כמותית (QPCR) או על ידי רצף של הדור הבא (NGS). אנא לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
Protocol
Representative Results
Discussion
One of the primary challenges with conventional ChIP is the identification of suitable antibodies. ChIP depends heavily upon the quality of the antibody, and most commercial antibodies are unacceptable for ChIP. In fact, the Encyclopedia of DNA Elements (ENCODE) consortium found only 20% of commercial antibodies to be suitable for ChIP assays.50 With COSMIC, antibodies are replaced by streptavidin. Because polyamides are functionalized with biotin, streptavidin is used in place of an antibody to capture polyam…
Declarações
The authors have nothing to disclose.
Acknowledgements
We thank members of the Ansari lab and Prof. Parameswaran Ramanathan for helpful discussions. This work was supported by NIH grants CA133508 and HL099773, the H. I. Romnes faculty fellowship, and the W. M. Keck Medical Research Award to A.Z.A. G.S.E. was supported by a Peterson Fellowship from the Department of Biochemistry and Molecular Biosciences Training Grant NIH T32 GM07215. A.E. was supported by the Morgridge Graduate Fellowship and the Stem Cell and Regenerative Medicine Center Fellowship, and D.B. was supported by the NSEC grant from NSF.
Materials
| Phenylmethylsulfonyl fluoride (PMSF) | any source | ||
| Benzamidine | any source | ||
| Pepstatin | any source | ||
| Proteinase K | any source | ||
| Dynabeads MyOne Streptavidin C1 | Life Technologies | 65001 | |
| PBS, pH 7.4 | Life Technologies | 10010-023 | Other sources can be used |
| StemPro® Accutase® Cell Dissociation Reagent | Life Technologies | A1110501 | |
| QIAquick PCR Purification Kit | Qiagen | 28104 | We have tried other manufacturers of DNA columns with success. |
| TruSeq ChIP Sample Prep Kit | Illumina | IP-202-1012 | This Kit can be used to prepare COSMIC DNA for next-generation sequencing |
| Matrigel Basement Membrane Matrix | BD Biosciences | 356231 | Used to coat plates in order to grow H1 ESCs |
| pH paper | any source | ||
| amber microcentrifuge tubes | any source | ||
| microcentrifuge tubes | any source | ||
| pyrex filter | any source | Pyrex baking dishes are suitable | |
| qPCR master mix | any source | ||
| RNase | any source | ||
| HCl (6 N) | any source | ||
| 10-cm tissue culture dishes | any source | ||
| Serological pipettes | any source | ||
| Pasteur pipettes | any source | ||
| Pipette tips | any source | ||
| 15-mL conical tubes | any source | ||
| centrifuge | any source | ||
| microcentrifuge | any source | ||
| nutator | any source | ||
| Magnetic separation rack | any source | ||
| UV source | CalSun | B001BH0A1A | Other UV sources can be used, but crosslinking time must be optimized empirically |
| Misonix Sonicator | Qsonica | S4000 with 431C1 cup horn | Other sonicators can be used, but sonication conditions must be optimized empirically |
| Humidified CO2 incubator | any source | ||
| Biological safety cabinet with vacuum outlet | any source |
Referências
- Lee, T. I., Young, R. A. Transcriptional Regulation and Its Misregulation in Disease. Cell. 152, 1237-1251 (2013).
- Vaquerizas, J. M., Kummerfeld, S. K., Teichmann, S. A., Luscombe, N. M. A census of human transcription factors: function, expression and evolution. Nat Rev Genet. 10, 252-263 (2009).
- Meier, J. L., Yu, A. S., Korf, I., Segal, D. J., Dervan, P. B. Guiding the Design of Synthetic DNA-Binding Molecules with Massively Parallel Sequencing. J. Am. Chem. Soc. 134, 17814-17822 (2012).
- Dervan, P. B. Molecular recognition of DNA by small molecules. Bioorg. Med. Chem. 9, 2215-2235 (2001).
- Wemmer, D. E., Dervan, P. B. Targeting the minor groove of DNA. Curr. Opin. Struct. Biol. 7, 355-361 (1997).
- Eguchi, A., Lee, G. O., Wan, F., Erwin, G. S., Ansari, A. Z. Controlling gene networks and cell fate with precision-targeted DNA-binding proteins and small-molecule-based genome readers. Biochem. J. 462, 397-413 (2014).
- Mrksich, M., et al. Antiparallel side-by-side dimeric motif for sequence-specific recognition in the minor groove of DNA by the designed peptide 1-methylimidazole-2-carboxamide netropsin. Proc Natl Acad Sci U S A. 89, 7586-7590 (1992).
- Edayathumangalam, R. S., Weyermann, P., Gottesfeld, J. M., Dervan, P. B., Luger, K. Molecular recognition of the nucleosomal “supergroove”. Proc Natl Acad Sci U S A. 101, 6864-6869 (2004).
- Suto, R. K., et al. Crystal structures of nucleosome core particles in complex with minor groove DNA-binding ligands. J. Mol. Biol. 326, 371-380 (2003).
- Gottesfeld, J. M., et al. Sequence-specific Recognition of DNA in the Nucleosome by Pyrrole-Imidazole Polyamides. J. Mol. Biol. 309, 615-629 (2001).
- Chenoweth, D. M., Dervan, P. B. Structural Basis for Cyclic Py-Im Polyamide Allosteric Inhibition of Nuclear Receptor Binding. J. Am. Chem. Soc. 132, 14521-14529 (2010).
- Raskatov, J. A., et al. Modulation of NF-κB-dependent gene transcription using programmable DNA minor groove binders. Proc. Natl. Acad. Sci. U.S.A. 109, 1023-1028 (2012).
- Yang, F., et al. Antitumor activity of a pyrrole-imidazole polyamide. Proc. Natl. Acad. Sci. U.S.A. 110, 1863-1868 (2013).
- Mapp, A. K., Ansari, A. Z., Ptashne, M., Dervan, P. B. Activation of gene expression by small molecule transcription factors. Proc. Natl. Acad. Sci. U.S.A. 97, 3930-3935 (2000).
- Ansari, A. Z., Mapp, A. K., Nguyen, D. H., Dervan, P. B., Ptashne, M. Towards a minimal motif for artificial transcriptional activators. Chem. Biol. 8, 583-592 (2001).
- Arora, P. S., Ansari, A. Z., Best, T. P., Ptashne, M., Dervan, P. B. Design of artificial transcriptional activators with rigid poly-L-proline linkers. J. Am. Chem. Soc. 124, 13067-13071 (2002).
- Nickols, N. G., Jacobs, C. S., Farkas, M. E., Dervan, P. B. Modulating Hypoxia-Inducible Transcription by Disrupting the HIF-1–DNA Interface. ACS Chemical Biology. 2, 561-571 (2007).
- Pandian, G. N., et al. A synthetic small molecule for rapid induction of multiple pluripotency genes in mouse embryonic fibroblasts. Sci. Rep. 2, 544 (2012).
- Pandian, G. N., et al. Synthetic Small Molecules for Epigenetic Activation of Pluripotency Genes in Mouse Embryonic Fibroblasts. Chem Bio Chem. 12, 2822-2828 (2011).
- He, G., et al. Binding studies of a large antiviral polyamide to a natural HPV sequence. Biochimie. 102, 83-91 (2014).
- Edwards, T. G., Vidmar, T. J., Koeller, K., Bashkin, J. K., Fisher, C. DNA Damage Repair Genes Controlling Human Papillomavirus (HPV) Episome Levels under Conditions of Stability and Extreme Instability. PLoS ONE. 8, e75406 (2013).
- Edwards, T. G., Helmus, M. J., Koeller, K., Bashkin, J. K., Fisher, C. HPV Episome Stability is Reduced by Aphidicolin and Controlled by DNA Damage Response Pathways. Journal of Virology. , (2013).
- Edwards, T. G., et al. HPV episome levels are potently decreased by pyrrole-imidazole polyamides. Antiviral Res. 91, 177-186 (2011).
- Dickinson, L. A., et al. Inhibition of RNA polymerase II transcription in human cells by synthetic DNA-binding ligands. Proc. Natl. Acad. Sci. U.S.A. 95, 12890-12895 (1998).
- Dickinson, L. A., et al. Arresting Cancer Proliferation by Small-Molecule Gene Regulation. Chem. Biol. 11, 1583-1594 (2004).
- Nickols, N. G., et al. Activity of a Py–Im Polyamide Targeted to the Estrogen Response Element. Molecular Cancer Therapeutics. 12, 675-684 (2013).
- Raskatov, J. A., Puckett, J. W., Dervan, P. B. A C-14 labeled Py–Im polyamide localizes to a subcutaneous prostate cancer tumor. Bioorg. Med. Chem. 22, 4371-4375 (2014).
- Jespersen, C., et al. Chromatin structure determines accessibility of a hairpin polyamide–chlorambucil conjugate at histone H4 genes in pancreatic cancer cells. Bioorg. Med. Chem. Lett. 22, 4068-4071 (2012).
- Chou, C. J., et al. Small molecules targeting histone H4 as potential therapeutics for chronic myelogenous leukemia. Molecular Cancer Therapeutics. 7, 769-778 (2008).
- Nickols, N. G., Dervan, P. B. Suppression of androgen receptor-mediated gene expression by a sequence-specific DNA-binding polyamide. Proc. Natl. Acad. Sci. U.S.A. 104, 10418-10423 (2007).
- Minoshima, M., Bando, T., Sasaki, S., Fujimoto, J., Sugiyama, H. Pyrrole-imidazole hairpin polyamides with high affinity at 5CGCG3 DNA sequence; influence of cytosine methylation on binding. Nucleic Acids Res. 36, 2889-2894 (2008).
- Warren, C. L., et al. Fabrication of duplex DNA microarrays incorporating methyl-5-cytosine. Lab on a Chip. 12, 376-380 (2012).
- Dudouet, B., et al. Accessibility of nuclear chromatin by DNA binding polyamides. Chem. Biol. 10, 859-867 (2003).
- Carlson, C. D., et al. Specificity landscapes of DNA binding molecules elucidate biological function. Proc. Natl. Acad. Sci. U.S.A. 107, 4544-4549 (2010).
- Warren, C. L., et al. Defining the sequence-recognition profile of DNA-binding molecules. Proc. Natl. Acad. Sci. U.S.A. 103, 867-872 (2006).
- Tietjen, J. R., Donato, L. J., Bhimisaria, D., Ansari, A. Z., Voigt, C. Chapter One – Sequence-Specificity and Energy Landscapes of DNA-Binding Molecules. Methods Enzymol. 497, 3-30 (2011).
- Puckett, J. W., et al. Quantitative microarray profiling of DNA-binding molecules. J. Am. Chem. Soc. 129, 12310-12319 (2007).
- Keles, S., Warren, C. L., Carlson, C. D., Ansari, A. Z. CSI-Tree: a regression tree approach for modeling binding properties of DNA-binding molecules based on cognate site identification (CSI) data. Nucleic Acids Res. 36, 3171-3184 (2008).
- Hauschild, K. E., Stover, J. S., Boger, D. L., Ansari, A. Z. CSI-FID: High throughput label-free detection of DNA binding molecules. Bioorg. Med. Chem. Lett. 19, 3779-3782 (2009).
- Lee, M., Roldan, M. C., Haskell, M. K., McAdam, S. R., Hartley, J. A. . In vitro Photoinduced Cytotoxicity and DNA Binding Properties of Psoralen and Coumarin Conjugates of Netropsin Analogs: DNA Sequence-Directed Alkylation and Cross-Link. 37, 1208-1213 (1994).
- Wurtz, N. R., Dervan, P. B. Sequence specific alkylation of DNA by hairpin pyrrole–imidazole polyamide conjugates. Chem. Biol. 7, 153-161 (2000).
- Tung, S. -. Y., Hong, J. -. Y., Walz, T., Moazed, D., Liou, G. -. G. Chromatin affinity-precipitation using a small metabolic molecule: its application to analysis of O-acetyl-ADP-ribose. Cell. Mol. Life Sci. 69, 641-650 (2012).
- Rodriguez, R., Miller, K. M. Unravelling the genomic targets of small molecules using high-throughput sequencing. Nat Rev Genet. 15, 783-796 (2014).
- Guan, L., Disney, M. D. Covalent Small-Molecule–RNA Complex Formation Enables Cellular Profiling of Small-Molecule–RNA Interactions. Angew. Chem. Int. Ed. 52, 10010-10013 (2013).
- White, J. D., et al. Picazoplatin, an Azide-Containing Platinum(II) Derivative for Target Analysis by Click Chemistry. J. Am. Chem. Soc. 135, 11680-11683 (2013).
- Rodriguez, R., et al. Small-molecule–induced DNA damage identifies alternative DNA structures in human genes. Nat Chem Biol. 8, 301-310 (2012).
- Bando, T., Sugiyama, H. Synthesis and Biological Properties of Sequence-Specific DNA-Alkylating Pyrrole−Imidazole Polyamides. Acc. Chem. Res. 39, 935-944 (2006).
- Anders, L., et al. Genome-wide localization of small molecules. Nat. Biotechnol. 32, 92-96 (2014).
- Jin, C., et al. Chem-seq permits identification of genomic targets of drugs against androgen receptor regulation selected by functional phenotypic screens. Proc. Natl. Acad. Sci. U.S.A. 111, 9235-9240 (2014).
- Landt, S. G., et al. ChIP-seq guidelines and practices of the ENCODE and modENCODE consortia. Genome Research. 22, 1813-1831 (2012).
- Erwin, G. S., Bhimsaria, D., Eguchi, A., Ansari, A. Z. Mapping Polyamide–DNA Interactions in Human Cells Reveals a New Design Strategy for Effective Targeting of Genomic Sites. Angew. Chem. Int. Ed. 53, 10124-10128 (2014).
- Hyde, J. E., Hearst, J. E. Binding of psoralen derivatives to DNA and chromatin: influence of the ionic environment on dark binding and photoreactivity. Bioquímica. 17, 1251-1257 (1978).
- Erwin, G. S., Bhimsaria, D., Rodríguez-Martínez, J. A., Grieshop, M. P., Ansari, A. Z. Genome-wide localization of polyamide-based genome readers reveals sequence-based binding to repressive heterochromatin. In preparation. , (2015).
- Chen, G., et al. Chemically defined conditions for human iPSC derivation and culture. Nat Meth. 8, 424-429 (2011).
- Deliard, S., Zhao, J., Xia, Q., Grant, S. F. A. Generation of High Quality Chromatin Immunoprecipitation DNA Template for High-throughput Sequencing (ChIP-seq). J Vis Exp. (74), e50286 (2013).
- Shi, Y. B., Spielmann, H. P., Hearst, J. E. Base-catalyzed reversal of a psoralen-DNA cross-link. Bioquímica. 27, 5174-5178 (1988).
- Kumaresan, K. R., Hang, B., Lambert, M. W. Human Endonucleolytic Incision of DNA 3′ and 5′ to a Site-directed Psoralen Monoadduct and Interstrand. J. Biol. Chem. 270, 30709-30716 (1995).
- Cimino, G. D., Shi, Y. B., Hearst, J. E. Wavelength dependence for the photoreversal of a psoralen-DNA crosslink. Bioquímica. 25, 3013-3020 (1986).
- Heinz, S., et al. Simple Combinations of Lineage-Determining Transcription Factors Prime cis-Regulatory Elements Required for Macrophage and B Cell Identities. Mol. Cell. 38, 576-589 (2010).
- Zhang, Y., et al. Model-based analysis of ChIP-Seq (MACS). Genome Biol. 9, R137 (2008).
- Kharchenko, P. V., Tolstorukov, M. Y., Park, P. J. Design and analysis of ChIP-seq experiments for DNA-binding proteins. Nat Biotech. 26, 1351-1359 (2008).
- Diamandis, E. P., Christopoulos, T. K. The biotin-(strept)avidin system: principles and applications in biotechnology. Clin. Chem. 37, 625-636 (1991).
- Martinson, H. G., True, R. J. On the mechanism of nucleosome unfolding. Bioquímica. 18, 1089-1094 (1979).
- Gloss, L. M., Placek, B. J. The Effect of Salts on the Stability of the H2A−H2B Histone Dimer. Bioquímica. 41, 14951-14959 (2002).
- Jackson, V. Formaldehyde Cross-Linking for Studying Nucleosomal Dynamics. Methods. 17, 125-139 (1999).
- Kasinathan, S., Orsi, G. A., Zentner, G. E., Ahmad, K., Henikoff, S. High-resolution mapping of transcription factor binding sites on native chromatin. Nat Meth. 11, 203-209 (2014).
- Teytelman, L., Thurtle, D. M., Rine, J., van Oudenaarden, A. Highly expressed loci are vulnerable to misleading ChIP localization of multiple unrelated proteins. Proc. Natl. Acad. Sci. U.S.A. 110, 18602-18607 (2013).
- . Phantompeakqualtools home page Available from: https://www.encodeproject.org/software/phantompeakqualtools/ (2010)
- Wang, D., Lippard, S. J. Cellular processing of platinum anticancer drugs. Nature Reviews Drug Discovery. 4, 307-320 (2005).
- Hurley, L. H. DNA and its associated processes as targets for cancer therapy. Nat. Rev. Cancer. 2, 188-200 (2002).

