Oligomerisatie dynamiek van celoppervlak receptoren in levende cellen door totale inwendige reflectie fluorescentiemicroscopie gecombineerd met cijfer-en helderheids analyse
Summary
We beschrijven een beeldvormings aanpak voor de bepaling van de gemiddelde oligomere toestand van mEGFP-Tagged-receptor oligomeren geïnduceerd door ligand binding in het plasma membraan van levende cellen. Het protocol is gebaseerd op de totale inwendige reflectie fluorescentie (TIRF) microscopie gecombineerd met getal en helderheid (N & B) analyse.
Abstract
Ondanks het belang en de alomtegenwoordigheid van de receptor oligomisatie, zijn enkele methoden van toepassing voor het detecteren van clustering gebeurtenissen en het meten van de mate van clustering. Hier beschrijven we een beeldvormings benadering om de gemiddelde oligomere toestand van mEGFP-Tagged-receptor-homo complexen in het membraan van levende cellen te bepalen. Het protocol is gebaseerd op de totale inwendige reflectie fluorescentie (TIRF) microscopie gecombineerd met getal en helderheid (N & B) analyse. N & B is een methode die vergelijkbaar is met fluorescentie-correlatie spectroscopie (FCS) en foton Counting histogram (PCH), die gebaseerd zijn op de statistische analyse van de schommelingen van de fluorescentie intensiteit van fluoroforen die in en uit een verlichtings volume tijdens een observatietijd. In het bijzonder is N & B een vereenvoudiging van PCH om informatie te verkrijgen over het gemiddelde aantal eiwitten in oligomere mengsels. De intensiteit fluctuatie amplitudes worden beschreven door de moleculaire helderheid van de de en het gemiddelde aantal fluor Foren binnen het verlichtings volume. Zo beschouwt N & B alleen de eerste en tweede momenten van de amplitude verdeling, namelijk de gemiddelde intensiteit en de variantie. Dit is tegelijkertijd de sterkte en de zwakte van de methode. Omdat slechts twee momenten worden overwogen, kan N & B de molaire fractie van onbekende oligomeren in een mengsel niet bepalen, maar het schat alleen de gemiddelde oligomerisatie toestand van het mengsel. Niettemin kan het worden toegepast op relatief kleine tijdreeksen (vergeleken met andere moment methoden) van beelden van levende cellen op pixel-voor-pixel basis, simpelweg door de tijds schommelingen van de intensiteit van de fluorescentie te bewaken. Het vermindert de effectieve tijd per pixel tot een paar microseconden, waardoor acquisitie in het tijdsbereik van seconden tot milliseconden, die nodig is voor snelle oligomerisatie kinetiek. Ten slotte kunnen grote celgebieden en sub-cellulaire compartimenten worden verkend.
Introduction
We beschrijven een totale interne reflectie fluorescentie-nummer en helderheid (TIRF-N & B) Imaging benadering voor het bepalen van de gemiddelde oligomere toestand van receptor moleculen op het plasma membraan van levende cellen, gericht op het koppelen van de receptor assemblage dynamiek aan de biologische functie van de eiwitten (Figuur 1).
Bij extracellulaire ligand binding initiëren receptoren de intracellulaire signaaltransductie afhankelijk van hun conformatie, oligomerisatie, potentiële co-receptoren en membraan samenstelling. Ondanks het belang en de alomtegenwoordigheid van de receptor oligomerisatie, herkend als een belangrijke gebeurtenis in cellulaire signalering1,2,3,4,5,6, 7, enkele methoden kunnen clustering gebeurtenissen detecteren en meten van de mate van clustering experimentaal8,9. Het confocale volume (x, y ≈ 300 nm, z ≈ 900 nm) is onvoldoende verdwenen voor het aantonen van moleculaire interactie en stoichiometrie, zelfs na optimalisatie door beeld restauratie algoritmen10. De samenstelling van de subeenheid van eiwit oligomeren kan niet op zuiver ruimtelijke basis worden opgelost, zelfs niet door super-resolutie methoden bij x, y-resolutie van 20-70 nm zoals PALM11, Storm12en STED13. Bovendien kan de tijdelijke resolutie (in de volgorde van minuten per afbeelding) kinetiek niet in het bereik van seconden volgen. Enkelvoudige molecuul stap-bleaching lost de stoichiometrie van eiwit oligomeren alleen op als ze immobiel zijn14.
Een van de meest veelzijdige methoden om dichtheid en oligomisatie van fluorescently gelabelde eiwitten binnen enkele afbeeldingen te meten, is de analyse van ruimtelijke intensiteits verdeling (SpIDA), die afhankelijk is van ruimtelijke bemonstering. Het is toepasbaar op zowel chemisch vaste als levende cellen, en maakt het mogelijk de analyse van verschillende gebieden van belang van de cel tegelijkertijd met behulp van standaard fluorescentiemicroscopie15. Als alternatief zijn moment methoden, zoals fluorescentie-correlatie spectroscopie (FCS)16, foton Counting histogram (PCH)17, en aantal en helderheid (N & B)18,19, geschikt voor kwantitatief oligomeren Metingen. Deze methoden analyseren de intensiteit van de fluorescentie schommelingen die in de tijd kunnen worden waargenomen wanneer de fluor Foren zich in en uit een verlichtings volume verspreiden. De amplitudes van de intensiteits schommelingen kunnen op unieke wijze worden beschreven door de moleculaire helderheid van de de (ε) en het gemiddelde aantal fluor Foren (n) binnen het verlichtings volume17 (Figuur 2). Normaalgesproken kan de diffusiecoëfficiënt van de fluor Foren en het gemiddelde aantal moleculen (omgekeerd gerelateerd aan de G (0)-waarde) binnen het verlichtings volume worden verkregen door FCS20. Aangezien de diffusie tijd echter alleen wordt geschaald met de kubieke wortel van de massa, is FCS niet voldoende gevoelig voor het detecteren van veranderingen in molecuulmassa21. In de praktijk, single Color FCS kan niet detecteren door dimerisatie van membraan receptoren. PCH lost mengsels van verschillende oligomeren nauwkeurig op. Met behulp van meer dan twee momenten van de amplitude-verdeling, detecteert het moleculen van verschillende helderheid die hetzelfde verlichtings volume innemen. Scanning FCS22 en ontwikkelingen, zoals de interessante pair-correlatie van moleculaire helderheid (pcomb) approach23, geïntroduceerd om het bereik van toepasbaarheid van fluorescentie correlatie methoden in biologische systemen uit te breiden24 , blijven methoden met één punt ontbreken van snelle metingen in een groot gebied van een cel, waarbij veel opeenvolgende waarnemingen bij elke pixel en het verzamelen van gegevens in de volgorde van seconden worden vereist.
N & B is een vereenvoudigde versie van PCH die alleen de eerste en tweede momenten van de amplitude van de fluorescentie verdeling beschouwt, namelijk de gemiddelde intensiteit, , en de variantie, σ2 (Figuur 2)18,19 en daarom kan het de molaire fractie van onbekende oligomeren in een mengsel niet bepalen, maar slechts schattingen van de gemiddelde oligomerisatie toestand van het mengsel. Niettemin heeft N & B het voordeel van het werken met relatief kleinere tijdreeksen beelden van levende cellen dan PCH per pixel, simpelweg door de schommelingen op de tijd van de intensiteit van de fluorescentie te bewaken. Omdat N & B de tijd per pixel tot een paar microseconden verlaagt, kan het snelle oligomerisatie kinetiek volgen op grote celgebieden, waardoor beeld verwerving op een tijdschaal van seconden in raster scanning microscopie (bijv. confocale, 2-photon) en milliseconden op camera gebaseerde microscopie (bijv. TIRFM).
Verschillende rapporten hebben aangetoond dat N & B het aantal subeenheden in eiwit clusters kwantificeren door uitgebreide celregio’s. Paxillin-EGFP-clusters werden aangetroffen op de adhesieve plekken in de CHO-K1-cellen25, en de intracellulaire aggregatie van het pathogene Httex1p peptide werd beschreven in cos-7-cellen26. N & B werd toegepast voor het volgen van de ligand-gestuurde oligomisatie van de ErbB-receptor27, en het effect van de LIGAND FGF21 op Klothob (klb) en FGFR1c in HeLa-cellen28. De combinatie van tirf-beeldvorming en N & B-analyse werd gebruikt om aan te tonen dat dynamin-2 voornamelijk tetramere is in het gehele celmembraan29. We hebben N & B toegepast op zowel raster scanning als tirf-beelden om ligand-gestuurde door dimerisatie van upar en FGFR1 celmembraan receptoren30,31te bewijzen.
Fluorescentie-correlatie methoden, zoals N & B, FCS en PCH, zijn gebaseerd op de gedachte dat in een open volume het bezettings getal van deeltjes een Poisson-verdeling volgt. Omdat alleen de fotonen die de fluoroforen uitzenden kunnen worden gedetecteerd, de gemiddelde waarde voor een gemeten fluorescentie intensiteit versus tijd in een pixel van het beeld,  , is het product van het gemiddelde aantal fluor Foren in het verlichtings volume, n, en hun moleculaire helderheid, ε17:
, is het product van het gemiddelde aantal fluor Foren in het verlichtings volume, n, en hun moleculaire helderheid, ε17:

waarbij ε wordt uitgedrukt als het aantal fotonen dat per tijdseenheid (conventioneel per seconde) per molecuul wordt uitgezonden wanneer het molecuul zich in het midden van het verlichtings volume bevindt.
Helderheid is een eigenschap van elke de in een bepaalde overname instellen, terwijl intensiteit de som is van alle bijdragen van alle fluor Foren. Bij biologische wedstrijden zal de helderheid toenemen met de toename van het aantal fluoroforen dat samen fluctueert, wat informatie geeft over de oligomerisatie toestand van het fluorescently-gelabelde eiwit. De fluctuatie amplituden op een bepaalde pixel wordt gemeten aan de variantie van het fluorescentie signaal, σ2:

Wanneer het gemiddelde van het kwadraat van de intensiteit  , en het kwadraat van het gemiddelde van de intensiteit
, en het kwadraat van het gemiddelde van de intensiteit  , worden berekend op basis van de individuele intensiteitswaarden in elke pixel van elk frame:
, worden berekend op basis van de individuele intensiteitswaarden in elke pixel van elk frame:
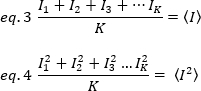
waarbij K het totale aantal frames in de tijdreeks is. Experimenteel is het noodzakelijk om voor de hele Afbeeldingsreeks de variantie te berekenen die de spreiding van de individuele intensiteitswaarden op elke pixel van één afbeelding rond de gemiddelde intensiteitswaarde beschrijft. De variantie omvat alle schommelingen van verschillende oorsprongen. Bij een eerste benadering kan de variantie als gevolg van de diffusisolerende deeltjes in het verlichtings volume σ20worden gescheiden van de variantie als gevolg van de detector schot ruis, σ2d. De twee varianties zijn onafhankelijk; Zo wordt de totale variantie gegeven door hun som:

De variantie, als gevolg van moleculaire schommelingen in en uit het detectie volume, is lineair afhankelijk van de moleculaire helderheid en intensiteit:

EQ. 6 opnieuw rangschikken volgens EQ. 1:

Volgens het typische concept in fluorescentie correlatie spectroscopie, stelt vergelijking 7 dat de variantie als gevolg van het aantal schommelingen afhangt van het kwadraat van de deeltjes helderheid.
Vervolgens is de variantie als gevolg van schommelingen in de detector een lineaire functie van de gedetecteerde intensiteit, onder de aanname dat de detector onder de verzadigings limiet19wordt bediend:

In het geval van foon tellings detectoren a= 1 en c= 0 is de variantie van de detector gelijk aan de gemiddelde intensiteit:

Om deze concepten op echte metingen in levende cellen toe te passen, definiëren Gratton en collega’s18 de schijnbare helderheid, B, voor elke pixel als de verhouding van de variantie over de gemiddelde intensiteit:

B is de parameter die experimenteel wordt gemeten. In dit werk, time series beelden van FGFR1 receptoren op het plasma membraan van HeLa cellen worden gevangen door TIRF microscopie en de gemiddelde schijnbare helderheid, B, wordt bepaald door de N & B analyse. Vervolgens, na toevoeging van FGF2, opeenvolgende tijdreeksen worden gevangen om de veranderingen in de zelf-assemblage van de receptor moleculen in het membraanoppervlak na stimulatie van de receptor met de canonieke ligand te volgen.
Aangezien de detector van de TIRF-Microscoop echter een EMCCD-camera is, moet de uitdrukking voor de schijnbare helderheid worden gewijzigd als19:

waarbij Offset de intensiteit offset is van de detectie-elektronica die kenmerkend is voor de detector instellingen. De variantie en gemiddelde intensiteit voor een analoge detector worden respectievelijk gegeven door:
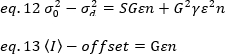
waarbij G de analoge winst in digitale niveaus (DL/fotonen), S, de digitale niveaus per foton19, wordt gegeven door de helling van een intensiteit versus variantie plot voor een lichtbron met constante intensiteit (geen tijdelijke schommelingen). De γ-factor is gerelateerd aan de vorm van het pixel detectie volume. Volgens Hassler et al.32is de γ-factor gelijk aan 0,3 voor tirf-beeldvorming die werkt op de maximale Gain van de detectie camera19. De parameters offset, S en G zijn kenmerken van de camera en de Microscoop. De schijnbare helderheid, B, wordt verkregen door het herschikken van EQ. 11 volgens EQ. 12 en 13:

Experimenteel is ε een complexe functie van laserintensiteit en de detectie-efficiëntie van het systeem. Niettemin, aangezien B/S lineair afhankelijk is van ε, is het alleen belangrijk om de relatieve waarde van ε te bepalen voor een bepaalde detectiemodus:

waar ε ‘ evenredig is aan ε. Toch wordt een kalibratie uitgevoerd met een interne referentie.
Protocol
Representative Results
Discussion
N & B vereist verschillende voorzorgsmaatregelen bij de keuze van het celmodel en de etiketterings strategie. Het kan alleen worden toegepast op levende cellen die stabiel blijven gehecht tijdens de opnametijd van de foto. Extra schommelingen als gevolg van de gehele cel rigide verplaatsing kunnen worden behandeld met de juiste beeld restauratie benaderingen38. Echter, in het algemeen wanneer een cel beweegt, de celmembraan ook vervormt, en structuur vervorming, produceren van grote extra varianti…
Declarações
The authors have nothing to disclose.
Acknowledgements
De CNIC wordt ondersteund door het ministerie van Ciencia, innovacion y Universidades en de Pro CNIC Foundation, en is een Severo Ochoa Center of Excellence (SEV-2015-0505). We worden ook gesteund door het Europees Fonds voor regionale ontwikkeling (FEDER) “una manera de hacer Europa”. UC erkent de steun van de Associazione Italiana ricerca Sul cancro, de vereniging voor internationaal kankeronderzoek (nu bekend als wereldwijd kankeronderzoek) en het Italiaanse ministerie van volksgezondheid. A.T. erkent de “Fondazione Banca del Monte di Lombardia” voor het gedeeltelijk ondersteunen van zijn werk met de PV Fellowship “Progetto professionalità Ivano Becchi” 2011-2012.
Materials
| 3-Colour Fast TIRF Leica AM TIRF MC inverted microscope, with smi-automatic TIRF alignment. The microscope is equipped with a diode 488 nm laser, a 100×1.46 oil TIRF objective, Ex/Em Bandpass filters at 490/20 and 525/50, temperature/CO2 incubator and Andor DU 8285 VP EMCCD camera. The microscope is operated by Leica LIF software. | Leica Microsystems, Wetzlar, Germany | ||
| Albumin from Bovine Serum 98% minimun | Sigma-Aldrich, St. Louis, MI, USA | A7906-100G | |
| DMEM without Phenol Red with 25 mM HEPES | GIBCO Thermo Fisher Scientific,Waltham, MA, USA | 21063029 | Used serum free for microscopy |
| DMEM high-glucose GlutaMAX I | GIBCO Thermo Fisher Scientific,Waltham, MA, USA | 10566-016 | Used for complete medium |
| Dulbecco's Phosphate Buffered Saline 10x (PBS) | Biowest, Nuaillé, France | X0515-500 | |
| Emission splitting system Photometrics DV2 | TeledynePhotometrics, Tucson, AZ, USA | ||
| Fetal Bovine Serum, qualified, Brazil | GIBCO Thermo Fisher Scientific,Waltham, MA, USA | 10270106 | 10% inactivated supplement for complete medium |
| Glass bottom 35-mm sterile 1.5 dishes | MatTek, Ashland, MA, USA | P35G-0.170-14-C | uncoated, glass thickness 0.17 microns |
| GraphPad Prism | GraphPad Software Inc., San Diego, CA, USA | ||
| Human cervical carcinoma (HeLa), serum-free animal component (AC) cells | Millipore-Sigma ECACC, Darmstadt, Germany | CB_08011102 | |
| iXonEM+ 897 EMCCD (back-illuminated) ANDOR camera controlled by ANDOR Solis software | Oxford Instruments, Andor TM Technology, Abingdon-on-Thames, UK | This camera, installed in an additional port of the microscope, is used for acquiring the N&B time series | |
| Matlab Executable N&B routine | Unit of Microscopy and Dynamic Imaging, CNIC, Madrid, Spain | download at https://www.cnic.es/en/investigacion/2/1187/tecnologia | |
| MatLab v.2018b | The MathWorks, Inc. Natick, MA, USA | download at https://www.mathworks.com/products/matlab.html | |
| Penicillin:Streptomycin for tissue culture 100x | Biowhittaker Inc. Walkersville, MD, USA | LONZA 17-602E | supplement for medium at Penicillin/Streptomycin 100U/100µg. |
| pN1-mEGFP-FGFR1 expression vector | Unit of Gynecological Oncology Research, European Institute of Oncology IRCCS, Milan, Italy | Zamai et al., 2019 | |
| pN1-N-Gly-mEGFP-GPI expression vector | Unit of Microscopy and Dynamic Imaging, CNIC, Madrid, Spain | Hellriegel et al., 2011 | |
| pN1-N-Gly-mEGFP-mEGFP-GPI expression vector | Unit of Microscopy and Dynamic Imaging, CNIC, Madrid, Spain | Hellriegel et al., 2011 | |
| Recombinant FGF2 | PeproTech EC, Ltd., London, UK | Ligand solution: 20ng/mL of FGF2 in PBS supplemented with 0.01%BSA. | |
| Sodium pyruvate GIBCO | ThermoFisher Scientific | 11360070 | 1mM supplement for medium |
| TransIt-LT1 Transfection Reagent | MirusBio LLC, Madison, WI, USA | MIR 2300 | |
| Trypsin-EDTA (0.25%), phenol red | GIBCO Thermo Fisher Scientific,Waltham, MA, USA | 25200056 | |
| Type F Immersion liquid 10 mL | Leica Microsystems, Wetzlar, Germany | 11513 859 | |
| UltraPure BSA (50 mg/mL) | ThermoFisher Scientific | AM2618 | 0.1% supplement for medium without phenol red used for transfections |
Referências
- Agwuegbo, U. C., Jonas, K. C. Molecular and functional insights into gonadotropin hormone receptor dimerization and oligomerization. Minerva Ginecologica. 70 (5), 539-548 (2018).
- Ferre, S., et al. G protein-coupled receptor oligomerization revisited: functional and pharmacological perspectives. Pharmacological Reviews. 66 (2), 413-434 (2014).
- Marsango, S., Ward, R. J., Alvarez-Curto, E., Milligan, G. Muscarinic receptor oligomerization. Neuropharmacology. 136 (Pt C), 401-410 (2018).
- Oishi, A., Cecon, E., Jockers, R. Melatonin Receptor Signaling: Impact of Receptor Oligomerization on Receptor Function. International Review of Cell and Molecular Biology. 338, 59-77 (2018).
- Thelen, M., Munoz, L. M., Rodriguez-Frade, J. M., Mellado, M. Chemokine receptor oligomerization: functional considerations. Current Opinion in Pharmacology. 10 (1), 38-43 (2010).
- Van Craenenbroeck, K. GPCR oligomerization: contribution to receptor biogenesis. Subcellular Biochemistry. 63, 43-65 (2012).
- Wnorowski, A., Jozwiak, K. Homo- and hetero-oligomerization of beta2-adrenergic receptor in receptor trafficking, signaling pathways and receptor pharmacology. Cell Signaling Technology. 26 (10), 2259-2265 (2014).
- Fricke, F., Dietz, M. S., Heilemann, M. Single-molecule methods to study membrane receptor oligomerization. Chemphyschem. 16 (4), 713-721 (2015).
- Vidi, P. A., Ejendal, K. F., Przybyla, J. A., Watts, V. J. Fluorescent protein complementation assays: new tools to study G protein-coupled receptor oligomerization and GPCR-mediated signaling. Molecular and Cellular Endocrinology. 331 (2), 185-193 (2011).
- Trussell, H. J., et al., Trussell, J., et al. . Academic Press Library in Signal Processing. 4, 3-9 (2014).
- Betzig, E., et al. Imaging intracellular fluorescent proteins at nanometer resolution. Science. 313 (5793), 1642-1645 (2006).
- Rust, M. J., Bates, M., Zhuang, X. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM). Nature Methods. 3 (10), 793-795 (2006).
- Nagerl, U. V., Willig, K. I., Hein, B., Hell, S. W., Bonhoeffer, T. Live-cell imaging of dendritic spines by STED microscopy. Proceedings of the National Academy of Sciences of the United States of America. 105 (48), 18982-18987 (2008).
- Tsekouras, K., Custer, T. C., Jashnsaz, H., Walter, N. G., Presse, S. A novel method to accurately locate and count large numbers of steps by photobleaching. Molecular Biology of the Cell. 27 (22), 3601-3615 (2016).
- Godin, A. G., et al. Revealing protein oligomerization and densities in situ using spatial intensity distribution analysis. Proceedings of the National Academy of Sciences of the United States of America. 108 (17), 7010-7015 (2011).
- Qian, H., Elson, E. L. Distribution of molecular aggregation by analysis of fluctuation moments. Proceedings of the National Academy of Sciences of the United States of America. 87 (14), 5479-5483 (1990).
- Chen, Y., Muller, J. D., So, P. T., Gratton, E. The photon counting histogram in fluorescence fluctuation spectroscopy. Biophysical Journal. 77 (1), 553-567 (1999).
- Dalal, R. B., Digman, M. A., Horwitz, A. F., Vetri, V., Gratton, E. Determination of particle number and brightness using a laser scanning confocal microscope operating in the analog mode. Microscopy Research and Technique. 71 (1), 69-81 (2008).
- Unruh, J. R., Gratton, E. Analysis of molecular concentration and brightness from fluorescence fluctuation data with an electron multiplied CCD camera. Biophysical Journal. 95 (11), 5385-5398 (2008).
- Hess, S. T., Huang, S., Heikal, A. A., Webb, W. W. Biological and chemical applications of fluorescence correlation spectroscopy: a review. Bioquímica. 41 (3), 697-705 (2002).
- Muller, J. D., Chen, Y., Gratton, E. Fluorescence correlation spectroscopy. Methods in Enzymology. 361, 69-92 (2003).
- Levi, V., Ruan, Q., Kis-Petikova, K., Gratton, E. Scanning FCS, a novel method for three-dimensional particle tracking. Biochemical Society Transactions. 31 (Pt 5), 997-1000 (2003).
- Hinde, E., et al. Quantifying the dynamics of the oligomeric transcription factor STAT3 by pair correlation of molecular brightness. Nature Communications. 7, 11047 (2016).
- Waithe, D., et al. Optimized processing and analysis of conventional confocal microscopy generated scanning FCS data. Methods. 140-141, 62-73 (2018).
- Digman, M. A., Dalal, R., Horwitz, A. F., Gratton, E. Mapping the number of molecules and brightness in the laser scanning microscope. Biophysical Journal. 94 (6), 2320-2332 (2008).
- Ossato, G., et al. A two-step path to inclusion formation of huntingtin peptides revealed by number and brightness analysis. Biophysical Journal. 98 (12), 3078-3085 (2010).
- Nagy, P., Claus, J., Jovin, T. M., Arndt-Jovin, D. J. Distribution of resting and ligand-bound ErbB1 and ErbB2 receptor tyrosine kinases in living cells using number and brightness analysis. Proceedings of the National Academy of Sciences of the United States of America. 107 (38), 16524-16529 (2010).
- Ming, A. Y., et al. Dynamics and Distribution of Klothobeta (KLB) and fibroblast growth factor receptor-1 (FGFR1) in living cells reveal the fibroblast growth factor-21 (FGF21)-induced receptor complex. Journal of Biological Chemistry. 287 (24), 19997-20006 (2012).
- Ross, J. A., et al. Oligomerization state of dynamin 2 in cell membranes using TIRF and number and brightness analysis. Biophysical Journal. 100 (3), L15-L17 (2011).
- Hellriegel, C., Caiolfa, V. R., Corti, V., Sidenius, N., Zamai, M. Number and brightness image analysis reveals ATF-induced dimerization kinetics of uPAR in the cell membrane. FASEB J. 25 (9), 2883-2897 (2011).
- Zamai, M., et al. Number and brightness analysis reveals that NCAM and FGF2 elicit different assembly and dynamics of FGFR1 in live cells. Journal of Cell Science. 132 (1), (2019).
- Hassler, K., et al. Total internal reflection fluorescence correlation spectroscopy (TIR-FCS) with low background and high count-rate per molecule. Optics Express. 13 (19), 7415-7423 (2005).
- Di Rienzo, C., Gratton, E., Beltram, F., Cardarelli, F. From fast fluorescence imaging to molecular diffusion law on live cell membranes in a commercial microscope. Journal of Visualized Experiments. (92), e51994 (2014).
- Beenken, A., Mohammadi, M. The FGF family: biology, pathophysiology and therapy. Nature Reviews Drug Discovery. 8 (3), 235-253 (2009).
- Joubert, J., Sharma, D. Light microscopy digital imaging. Current Protocols in Cytometry. , (2011).
- Gell, C., Berndt, M., Enderlein, J., Diez, S. TIRF microscopy evanescent field calibration using tilted fluorescent microtubules. Journal of Microscopy. 234 (1), 38-46 (2009).
- Burghardt, T. P. Measuring incidence angle for through-the-objective total internal reflection fluorescence microscopy. Journal of Biomedical Optics. 17 (12), 126007 (2012).
- Trullo, A., Corti, V., Arza, E., Caiolfa, V. R., Zamai, M. Application limits and data correction in number of molecules and brightness analysis. Microscopy Research and Technique. 76 (11), 1135-1146 (2013).
- Caiolfa, V. R., et al. Monomer-dimer dynamics and distribution of GPI-anchored uPAR are determined by cell surface protein assemblies. Journal of Cell Biology. 179 (5), 1067-1082 (2007).
- Campbell, R. E., et al. A monomeric red fluorescent protein. Proceedings of the National Academy of Sciences of the United States of America. 99 (12), 7877-7882 (2002).
- Cutrale, F., et al. Using enhanced number and brightness to measure protein oligomerization dynamics in live cells. Nature Protocols. 14 (2), 616-638 (2019).
- Dunsing, V., Chiantia, S. A Fluorescence Fluctuation Spectroscopy Assay of Protein-Protein Interactions at Cell-Cell Contacts. Journal of Visualized Experiments. (142), (2018).

