Toplam İç Yansıma Floresan Mikroskobu Ile Canlı Hücrelerdeki Hücre Yüzey Reseptörlerinin Oligomerizasyon Dinamiği Sayı ve Parlaklık Analizi ile Birlikte
Summary
Canlı hücrelerin plazma zarında ligand bağlanması ile indüklenen mEGFP etiketli-reseptör oligomerlerinin ortalama oligomerik durumunun belirlenmesi için bir görüntüleme yaklaşımını tanımladık. Protokol, Sayı ve Parlaklık (N&B) analizi ile birlikte Toplam İç Yansıma Floresan (TIRF) mikroskobuna dayanmaktadır.
Abstract
Reseptör oligomerizasyonunun önemi ne kadar önemli ve her yerde yer alsa da, kümeleme olaylarının saptandırılması ve kümelenme derecesinin ölçülmesi için çok az yöntem uygulanmaktadır. Burada, canlı hücrelerin zarındaki mEGFP etiketli-reseptör homokomplekslerinin ortalama oligomerik durumunu belirlemek için bir görüntüleme yaklaşımını tanımladık. Protokol, Sayı ve Parlaklık (N&B) analizi ile birlikte Toplam İç Yansıma Floresan (TIRF) mikroskobuna dayanmaktadır. N&B, floresan-korelasyon spektroskopisi (FCS) ve foton sayma histogramına (PCH) benzer bir yöntemdir ve floresan ların floresan şiddetindeki dalgalanmaların bir aydınlatmaya girip çıkmasındaki dalgalanmaların istatistiksel analizine dayanır. bir gözlem süresi boyunca hacim. Özellikle, N & B oligomerik karışımların proteinlerin ortalama sayısı hakkında bilgi edinmek için PCH bir basitleştirme olduğunu. Yoğunluk dalgalanma genlikleri floroformoleküler parlaklık ve aydınlatma hacmi içinde floroforların ortalama sayısı ile tanımlanır. Böylece, N & B genlik dağılımı, yani, ortalama yoğunluğu ve varyans sadece ilk ve ikinci anları dikkate alır. Bu, aynı zamanda, güç ve yöntemin zayıflığıdır. Sadece iki dakika düşünüldüğünden, N&B bir karışımdaki bilinmeyen oligomerlerin molar fraksiyonunu belirleyemez, ancak sadece karışımın ortalama oliomerizasyon durumunu tahmin eder. Bununla birlikte, sadece floresan yoğunluğunun zaman dalgalanmalarını izleyerek, canlı hücrelerin görüntülerinin piksel piksel bazında nispeten küçük zaman serilerine (diğer an yöntemleriyle karşılaştırıldığında) uygulanabilir. Piksel başına etkili süreyi birkaç mikrosaniyeye düşürerek saniyeler ile milisaniye arasında zaman aralığında edinme sağlar ve bu da hızlı oligomerizasyon kinetikleri için gereklidir. Son olarak, büyük hücre alanları ve alt hücre altbölümleri incelenebilir.
Introduction
Canlı hücrelerin plazma zarındaki reseptör moleküllerinin ortalama oligomerik durumunu belirlemek için toplam Internal Yansıma Floresan-Sayı ve Parlaklık (TIRF-Sayı ve Parlaklık) görüntüleme yaklaşımını, reseptör diziliyi bağlamayı amaçlayan bir toplam iç yansıma floresan-sayı ve parlaklık (TIRF-N&B) görüntüleme yaklaşımını tanımladık. proteinlerin biyolojik işlevine dinamikler (Şekil 1).
Hücre dışı ligand bağlanması üzerine reseptörler konformasyonlarına, oliomerizasyonlarına, potansiyel ko-reseptörlerine ve membran kompozisyonlarına bağlı olarak hücre içi sinyal iletimini başlatırlar. Reseptör oligomerizasyonunun önemi ve her yerde, hücreselsinyalizasyon daönemli bir olay olarak kabul rağmen 1,2,3,4,5,6, 7, birkaç yöntem kümeleme olayları algılayabilir ve deneyselkümelemederecesini ölçmek 8,9. Konfokal hacim (x,y 』 300 nm, z 』 900 nm) moleküler etkileşimi ve stokiyometriyi kanıtlamak için yeterli olarak çözülmüş, görüntü restorasyon algoritmaları tarafından optimizasyon dan sonra bile10. Protein oligomerlerinin alt birim bileşimi, PALM11, STORM12ve STED13gibi 20-70 nm x,y çözünürlükte süper çözünürlük yöntemleriyle bile tamamen mekansal olarak çözülemez. Ayrıca, onların zamansal çözünürlük (görüntü başına dakika sırasına göre) saniye aralığında kinetik takip edemez. Tek moleküllü adım ağartma protein oligomerlerinin stoiyometrisini ancak hareketsiz olduklarında çözer14.
Floresan etiketli proteinlerin tek bir görüntü içinde yoğunluk ve oliomerizasyonunu ölçmek için en çok yönlü yöntemlerden biri, mekansal örneklemeye dayanan mekansal yoğunluk dağılım analizidir (SpIDA). Hem kimyasal olarak sabit hem de canlı hücreler için geçerlidir ve aynı anda standart floresan mikroskobu15kullanarak hücrenin ilgi çeşitli bölgelerin analizisağlar. Alternatif olarak, floresan-korelasyon spektroskopisi (FCS)16,foton sayma histogramı (PCH)17ve Sayı ve Parlaklık (N&B)18,19gibi moment yöntemleri kantitatif oligomer için uygundur Ölçüm. Bu yöntemler, floresan ların aydınlatma hacmine girip çıktığı zaman gözlemlenebilen floresan yoğunluk dalgalanmalarını analiz eder. Yoğunluk dalgalanmalarının genlikleri, floroforun moleküler parlaklığı (ε) ve aydınlatma hacmindeki ortalama florofor (n) sayısı(şekil 2)ile benzersiz bir şekilde tanımlanabilir. Tipik olarak, floroforların difüzyon katsayısı ve aydınlatma hacmi içindeki ortalama molekül sayısı (G(0) değeriile ters orantılı) FCS20ile elde edilebilir. Ancak, difüzyon süresi sadece kütlenin kübik kökü ile ölçeklenir beri, FCS moleküler kütle değişiklikleri tespit etmek için yeterince duyarlı değildir21. Uygulamada, tek renkli FCS membran reseptörlerinin dimerizasyon unu algılayamaz. PCH, farklı oligomerlerin karışımlarını doğru bir şekilde çözer. Genlik dağılımının iki dakikadan fazlaını kullanarak, aynı aydınlatma hacmini kaplayan farklı parlaklıktaki molekülleri algılar. Tarama FCS22 ve gelişmeler, moleküler parlaklık ilginç çift korelasyon gibi (pCOMB) yaklaşım23, biyolojik sistemlerde floresan korelasyon yöntemlerinin uygulanabilirlik aralığını genişletmek için tanıtıldı24 , bir hücrenin geniş bir alanında hızlı ölçüm yeteneği nden yoksun tek noktalı yöntemler kalır, saniye sırasına göre her piksel ve veri toplama birçok ardışık gözlemler gerektiren.
N&B, PCH’nin floresan dağılımının genliğin inkişanın sadece birinci ve ikinci anlarını, yani ortalama yoğunluğunu, , ve varyans, σ2 (Şekil 2) 18,19’udikkate alan basitleştirilmiş bir sürümüdür. ve bu nedenle, bir karışımda bilinmeyen oligomerlerin molar fraksiyonunu belirleyemez, ancak sadece karışımın ortalama oliomerizasyon durumunu tahmin eder. Bununla birlikte, N & B sadece floresan yoğunluğu zamanında dalgalanmaları izleyerek, bir piksel-piksel bazında PCH daha canlı hücrelerin görüntüleri nispeten daha küçük zaman serisi ile çalışma avantajı vardır. N&B piksel başına zamanı birkaç mikrosaniyeye düşürebildiği için, büyük hücre alanları üzerinde hızlı oligomerizasyon kinetiği izleyebilir ve raster tarama mikroskopisinde (örn. konfokal, 2-foton) ve milisaniyelerde saniyeler ölçeğinde görüntü elde edilmesine izin verir. kamera tabanlı mikroskopide (örn. TIRFM).
Çeşitli raporlar, Genişletilmiş hücre bölgelerini görüntüleyerek protein kümelerinde alt birim sayısını ölçebilme yeteneğini göstermiştir. PAXIllin-EGFP kümeleri CHO-K1 hücrelerinde adezyon bölgelerinde tespit edildi25, ve patojenik Httex1p peptid in hücre içi agregasyonu COS-7 hücrelerinde tanımlanmıştır26. N & B ErbB reseptörünün ligand tahrikli oligomerizasyonu aşağıdaki için uygulandı27, ve HeLa hücrelerinde Klothob üzerinde ligand FGF21 etkisi (KLB) ve FGFR1c HeLa hücrelerinde28. TIRF görüntüleme ve N & B analizi kombinasyonu dinamin-2 öncelikle tüm hücre zarı29boyunca tetramerik olduğunu göstermek için kullanılmıştır. UPAR ve FGFR1 hücre zarı reseptörlerinin ligand tahrikli dimerizasyonunu kanıtlamak için hem raster tarama sınahem de TIRF görüntülerine N&B uyguladık30,31.
N&B, FCS ve PCH gibi floresan korelasyon yöntemleri, açık bir hacimde parçacıkların işgal sayısının Bir Poisson dağılımını izlediği fikrine dayanır. Sadece floroforların emit tespit edilebildiği fotonlar, görüntünün  bir pikselinde ölçülen floresan yoğunluğuile zaman arasındaki ortalama değer, aydınlatma hacmindeki ortalama floresan sayısının, n ve bunların moleküler parlaklık, ε17:
bir pikselinde ölçülen floresan yoğunluğuile zaman arasındaki ortalama değer, aydınlatma hacmindeki ortalama floresan sayısının, n ve bunların moleküler parlaklık, ε17:

molekül aydınlatma hacminin merkezindeyken molekül başına zaman birimi başına yayılan foton sayısı (geleneksel olarak saniyede) olarak ifade edilir.
Parlaklık belirli bir edinim kurmak her florofor bir özelliğidir, yoğunluk tüm floroforlardan tüm katkıların toplamı ise. Biyolojik yarışmalarda, birlikte dalgalanan florofor sayısının artmasıyla parlaklık artarak floresan etiketli proteinin oliomerizasyon durumu hakkında bilgi verecektir. Belirli bir pikseldeki dalgalanma genlikleri floresan sinyalinin varyansından ölçülür, σ2:

Yoğunluk  karesinin ortalaması ve yoğunluk ortalamasının karesi,
karesinin ortalaması ve yoğunluk ortalamasının karesi,  her karenin her pikselindeki tek tek yoğunluk değerlerinden hesaplanır:
her karenin her pikselindeki tek tek yoğunluk değerlerinden hesaplanır:
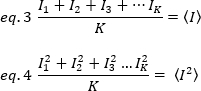
k zaman serisindeki toplam kare sayısıdır. Deneysel olarak, tüm görüntü serisi için ortalama yoğunluk değeri etrafında tek bir görüntünün her pikselinde tek tek yoğunluk değerlerinin dağılım açıklayan varyans hesaplamak için gereklidir. Varyans, farklı kökenlerdeki tüm dalgalanmaları içerir. İlk yaklaşık olarak, aydınlatma hacmindeki difüzyon parçacıklar nedeniyle varyans, σ20, dedektör atış gürültüsü nedeniyle varyans ayrılabilir, σ2d. İki varyans bağımsızdır; böylece, toplam varyans onların toplamı ile verilir:

Algılama hacmindeki ve dışında meydana gelen moleküler dalgalanmalar nedeniyle varyans, moleküler parlaklığa ve yoğunluğa doğrusal olarak bağlıdır:

Eq 1’e göre eq 6’nın yeniden düzenlenmesi:

Floresan korelasyon spektroskopisinde tipik kavrama göre, 7.
Daha sonra, dedektör dalgalanmaları nedeniyle varyans dedektör doygunluk sınırı nın altında çalıştırılır varsayımı altında, tespit yoğunluğunun doğrusal bir fonksiyonudur19:

Foton sayma dedektörleri durumunda a=1 ve c=0, böylece dedektör varyans ortalama yoğunluğuna eşittir:

Bu kavramları canlı hücrelerdeki gerçek ölçümlere uygulamak için Gratton ve meslektaşları18, her piksel için görünür parlaklığı, B’yi ortalama yoğunluk üzerinde varyans oranı olarak tanımlar:

B, deneysel olarak ölçülen parametredir. Bu çalışmada, HeLa hücrelerinin plazma zarındaki FGFR1 reseptörlerinin zaman serisi görüntüleri TIRF mikroskobu ile yakalanır ve ortalama parlaklık olan B, N&B analizi ile belirlenir. Daha sonra, FGF2 eklenmesinden sonra, ardışık zaman serileri kanonik ligand ile reseptör uyarılmasından sonra membran yüzeyinde reseptör moleküllerinin kendi kendine montaj değişiklikleri takip yakalanır.
Ancak, TIRF mikroskobu dedektörü bir EMCCD kamera olduğundan, görünür parlaklık için ifade19olarak değiştirilmesi gerekir:

ofset dedektör ayarlarının bir özelliğidir algılama elektroniği yoğunluğu ofset nerede. Bir analog dedektör için varyans ve ortalama yoğunluğu sırasıyla tarafından verilir:
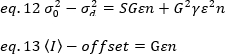
nerede G dijital düzeylerde analog kazanç (DL / foton), S, foton başına dijital düzeyleri19, sabit yoğunluğu (hiçbir zamansal dalgalanmalar) ile bir ışık kaynağı için bir yoğunluk karşı varyans arsa eğimi tarafından verilir. Γ faktörü piksel algılama hacminin şekli ile ilgilidir. Hassler ve ark.32’yegöre γ faktörü, algılama kamerası19’unmaksimum kazancıyla çalışan TIRF görüntülemeiçin 0,3’e eşittir. Ofset, S ve G parametreleri kamera ve mikroskop özellikleridir. Görünür parlaklık, B, eq 11 eq 12 ve 13 göre yeniden düzenlenmesi ile elde edilir:

Deneysel olarak, ε lazer yoğunluğu ve sistemin algılama verimliliği karmaşık bir fonksiyonudur. Bununla birlikte, B/S doğrusal olarak ε’ya bağlı olduğundan, yalnızca belirli bir algılama modu için ε’nin göreli değerini belirlemek önemlidir:

ε’ ile orantılı dır. Yine de, bir kalibrasyon dahili bir başvuru kullanılarak gerçekleştirilir.
Protocol
Representative Results
Discussion
N & B hücre modeli ve etiketleme stratejisi seçiminde çeşitli önlemler gerektirir. Yalnızca görüntü yakalama süresi boyunca uygun şekilde yapışmış kalan canlı hücrelere uygulanabilir. Tüm hücre katı deplasman nedeniyle ekstra dalgalanmalar uygun görüntü restorasyon yaklaşımları ile ele alınabilir38. Ancak, genellikle bir hücre hareket ettiğinde, hücre zarı da deforme olur, ve yapı deformasyonu, büyük ekstra varyans üreten, membran proteinlerinin analizi için cid…
Declarações
The authors have nothing to disclose.
Acknowledgements
CNIC, Ciencia Bakanlığı, Innovacion y Universidades ve Pro CNIC Vakfı tarafından desteklenir ve Severo Ochoa Mükemmellik Merkezi ‘dir (SEV-2015-0505). Avrupa Bölgesel Kalkınma Fonu (FEDER) “Una manera de hacer Europa” tarafından da destekliyoruz. UC Associazione Italiana Ricerca sul Cancro, Uluslararası Kanser Araştırma Derneği (şimdi Dünya Çapında Kanser Araştırma olarak da bilinir) ve İtalya Sağlık Bakanlığı desteğini kabul eder. A.T., 2011-2012’de PV Bursu “Progetto Professionalità Ivano Becchi” ile yaptığı çalışmaları kısmen desteklediği için “Fondazione Banca del Monte di Lombardia”yı kabul etti.
Materials
| 3-Colour Fast TIRF Leica AM TIRF MC inverted microscope, with smi-automatic TIRF alignment. The microscope is equipped with a diode 488 nm laser, a 100×1.46 oil TIRF objective, Ex/Em Bandpass filters at 490/20 and 525/50, temperature/CO2 incubator and Andor DU 8285 VP EMCCD camera. The microscope is operated by Leica LIF software. | Leica Microsystems, Wetzlar, Germany | ||
| Albumin from Bovine Serum 98% minimun | Sigma-Aldrich, St. Louis, MI, USA | A7906-100G | |
| DMEM without Phenol Red with 25 mM HEPES | GIBCO Thermo Fisher Scientific,Waltham, MA, USA | 21063029 | Used serum free for microscopy |
| DMEM high-glucose GlutaMAX I | GIBCO Thermo Fisher Scientific,Waltham, MA, USA | 10566-016 | Used for complete medium |
| Dulbecco's Phosphate Buffered Saline 10x (PBS) | Biowest, Nuaillé, France | X0515-500 | |
| Emission splitting system Photometrics DV2 | TeledynePhotometrics, Tucson, AZ, USA | ||
| Fetal Bovine Serum, qualified, Brazil | GIBCO Thermo Fisher Scientific,Waltham, MA, USA | 10270106 | 10% inactivated supplement for complete medium |
| Glass bottom 35-mm sterile 1.5 dishes | MatTek, Ashland, MA, USA | P35G-0.170-14-C | uncoated, glass thickness 0.17 microns |
| GraphPad Prism | GraphPad Software Inc., San Diego, CA, USA | ||
| Human cervical carcinoma (HeLa), serum-free animal component (AC) cells | Millipore-Sigma ECACC, Darmstadt, Germany | CB_08011102 | |
| iXonEM+ 897 EMCCD (back-illuminated) ANDOR camera controlled by ANDOR Solis software | Oxford Instruments, Andor TM Technology, Abingdon-on-Thames, UK | This camera, installed in an additional port of the microscope, is used for acquiring the N&B time series | |
| Matlab Executable N&B routine | Unit of Microscopy and Dynamic Imaging, CNIC, Madrid, Spain | download at https://www.cnic.es/en/investigacion/2/1187/tecnologia | |
| MatLab v.2018b | The MathWorks, Inc. Natick, MA, USA | download at https://www.mathworks.com/products/matlab.html | |
| Penicillin:Streptomycin for tissue culture 100x | Biowhittaker Inc. Walkersville, MD, USA | LONZA 17-602E | supplement for medium at Penicillin/Streptomycin 100U/100µg. |
| pN1-mEGFP-FGFR1 expression vector | Unit of Gynecological Oncology Research, European Institute of Oncology IRCCS, Milan, Italy | Zamai et al., 2019 | |
| pN1-N-Gly-mEGFP-GPI expression vector | Unit of Microscopy and Dynamic Imaging, CNIC, Madrid, Spain | Hellriegel et al., 2011 | |
| pN1-N-Gly-mEGFP-mEGFP-GPI expression vector | Unit of Microscopy and Dynamic Imaging, CNIC, Madrid, Spain | Hellriegel et al., 2011 | |
| Recombinant FGF2 | PeproTech EC, Ltd., London, UK | Ligand solution: 20ng/mL of FGF2 in PBS supplemented with 0.01%BSA. | |
| Sodium pyruvate GIBCO | ThermoFisher Scientific | 11360070 | 1mM supplement for medium |
| TransIt-LT1 Transfection Reagent | MirusBio LLC, Madison, WI, USA | MIR 2300 | |
| Trypsin-EDTA (0.25%), phenol red | GIBCO Thermo Fisher Scientific,Waltham, MA, USA | 25200056 | |
| Type F Immersion liquid 10 mL | Leica Microsystems, Wetzlar, Germany | 11513 859 | |
| UltraPure BSA (50 mg/mL) | ThermoFisher Scientific | AM2618 | 0.1% supplement for medium without phenol red used for transfections |
Referências
- Agwuegbo, U. C., Jonas, K. C. Molecular and functional insights into gonadotropin hormone receptor dimerization and oligomerization. Minerva Ginecologica. 70 (5), 539-548 (2018).
- Ferre, S., et al. G protein-coupled receptor oligomerization revisited: functional and pharmacological perspectives. Pharmacological Reviews. 66 (2), 413-434 (2014).
- Marsango, S., Ward, R. J., Alvarez-Curto, E., Milligan, G. Muscarinic receptor oligomerization. Neuropharmacology. 136 (Pt C), 401-410 (2018).
- Oishi, A., Cecon, E., Jockers, R. Melatonin Receptor Signaling: Impact of Receptor Oligomerization on Receptor Function. International Review of Cell and Molecular Biology. 338, 59-77 (2018).
- Thelen, M., Munoz, L. M., Rodriguez-Frade, J. M., Mellado, M. Chemokine receptor oligomerization: functional considerations. Current Opinion in Pharmacology. 10 (1), 38-43 (2010).
- Van Craenenbroeck, K. GPCR oligomerization: contribution to receptor biogenesis. Subcellular Biochemistry. 63, 43-65 (2012).
- Wnorowski, A., Jozwiak, K. Homo- and hetero-oligomerization of beta2-adrenergic receptor in receptor trafficking, signaling pathways and receptor pharmacology. Cell Signaling Technology. 26 (10), 2259-2265 (2014).
- Fricke, F., Dietz, M. S., Heilemann, M. Single-molecule methods to study membrane receptor oligomerization. Chemphyschem. 16 (4), 713-721 (2015).
- Vidi, P. A., Ejendal, K. F., Przybyla, J. A., Watts, V. J. Fluorescent protein complementation assays: new tools to study G protein-coupled receptor oligomerization and GPCR-mediated signaling. Molecular and Cellular Endocrinology. 331 (2), 185-193 (2011).
- Trussell, H. J., et al., Trussell, J., et al. . Academic Press Library in Signal Processing. 4, 3-9 (2014).
- Betzig, E., et al. Imaging intracellular fluorescent proteins at nanometer resolution. Science. 313 (5793), 1642-1645 (2006).
- Rust, M. J., Bates, M., Zhuang, X. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM). Nature Methods. 3 (10), 793-795 (2006).
- Nagerl, U. V., Willig, K. I., Hein, B., Hell, S. W., Bonhoeffer, T. Live-cell imaging of dendritic spines by STED microscopy. Proceedings of the National Academy of Sciences of the United States of America. 105 (48), 18982-18987 (2008).
- Tsekouras, K., Custer, T. C., Jashnsaz, H., Walter, N. G., Presse, S. A novel method to accurately locate and count large numbers of steps by photobleaching. Molecular Biology of the Cell. 27 (22), 3601-3615 (2016).
- Godin, A. G., et al. Revealing protein oligomerization and densities in situ using spatial intensity distribution analysis. Proceedings of the National Academy of Sciences of the United States of America. 108 (17), 7010-7015 (2011).
- Qian, H., Elson, E. L. Distribution of molecular aggregation by analysis of fluctuation moments. Proceedings of the National Academy of Sciences of the United States of America. 87 (14), 5479-5483 (1990).
- Chen, Y., Muller, J. D., So, P. T., Gratton, E. The photon counting histogram in fluorescence fluctuation spectroscopy. Biophysical Journal. 77 (1), 553-567 (1999).
- Dalal, R. B., Digman, M. A., Horwitz, A. F., Vetri, V., Gratton, E. Determination of particle number and brightness using a laser scanning confocal microscope operating in the analog mode. Microscopy Research and Technique. 71 (1), 69-81 (2008).
- Unruh, J. R., Gratton, E. Analysis of molecular concentration and brightness from fluorescence fluctuation data with an electron multiplied CCD camera. Biophysical Journal. 95 (11), 5385-5398 (2008).
- Hess, S. T., Huang, S., Heikal, A. A., Webb, W. W. Biological and chemical applications of fluorescence correlation spectroscopy: a review. Bioquímica. 41 (3), 697-705 (2002).
- Muller, J. D., Chen, Y., Gratton, E. Fluorescence correlation spectroscopy. Methods in Enzymology. 361, 69-92 (2003).
- Levi, V., Ruan, Q., Kis-Petikova, K., Gratton, E. Scanning FCS, a novel method for three-dimensional particle tracking. Biochemical Society Transactions. 31 (Pt 5), 997-1000 (2003).
- Hinde, E., et al. Quantifying the dynamics of the oligomeric transcription factor STAT3 by pair correlation of molecular brightness. Nature Communications. 7, 11047 (2016).
- Waithe, D., et al. Optimized processing and analysis of conventional confocal microscopy generated scanning FCS data. Methods. 140-141, 62-73 (2018).
- Digman, M. A., Dalal, R., Horwitz, A. F., Gratton, E. Mapping the number of molecules and brightness in the laser scanning microscope. Biophysical Journal. 94 (6), 2320-2332 (2008).
- Ossato, G., et al. A two-step path to inclusion formation of huntingtin peptides revealed by number and brightness analysis. Biophysical Journal. 98 (12), 3078-3085 (2010).
- Nagy, P., Claus, J., Jovin, T. M., Arndt-Jovin, D. J. Distribution of resting and ligand-bound ErbB1 and ErbB2 receptor tyrosine kinases in living cells using number and brightness analysis. Proceedings of the National Academy of Sciences of the United States of America. 107 (38), 16524-16529 (2010).
- Ming, A. Y., et al. Dynamics and Distribution of Klothobeta (KLB) and fibroblast growth factor receptor-1 (FGFR1) in living cells reveal the fibroblast growth factor-21 (FGF21)-induced receptor complex. Journal of Biological Chemistry. 287 (24), 19997-20006 (2012).
- Ross, J. A., et al. Oligomerization state of dynamin 2 in cell membranes using TIRF and number and brightness analysis. Biophysical Journal. 100 (3), L15-L17 (2011).
- Hellriegel, C., Caiolfa, V. R., Corti, V., Sidenius, N., Zamai, M. Number and brightness image analysis reveals ATF-induced dimerization kinetics of uPAR in the cell membrane. FASEB J. 25 (9), 2883-2897 (2011).
- Zamai, M., et al. Number and brightness analysis reveals that NCAM and FGF2 elicit different assembly and dynamics of FGFR1 in live cells. Journal of Cell Science. 132 (1), (2019).
- Hassler, K., et al. Total internal reflection fluorescence correlation spectroscopy (TIR-FCS) with low background and high count-rate per molecule. Optics Express. 13 (19), 7415-7423 (2005).
- Di Rienzo, C., Gratton, E., Beltram, F., Cardarelli, F. From fast fluorescence imaging to molecular diffusion law on live cell membranes in a commercial microscope. Journal of Visualized Experiments. (92), e51994 (2014).
- Beenken, A., Mohammadi, M. The FGF family: biology, pathophysiology and therapy. Nature Reviews Drug Discovery. 8 (3), 235-253 (2009).
- Joubert, J., Sharma, D. Light microscopy digital imaging. Current Protocols in Cytometry. , (2011).
- Gell, C., Berndt, M., Enderlein, J., Diez, S. TIRF microscopy evanescent field calibration using tilted fluorescent microtubules. Journal of Microscopy. 234 (1), 38-46 (2009).
- Burghardt, T. P. Measuring incidence angle for through-the-objective total internal reflection fluorescence microscopy. Journal of Biomedical Optics. 17 (12), 126007 (2012).
- Trullo, A., Corti, V., Arza, E., Caiolfa, V. R., Zamai, M. Application limits and data correction in number of molecules and brightness analysis. Microscopy Research and Technique. 76 (11), 1135-1146 (2013).
- Caiolfa, V. R., et al. Monomer-dimer dynamics and distribution of GPI-anchored uPAR are determined by cell surface protein assemblies. Journal of Cell Biology. 179 (5), 1067-1082 (2007).
- Campbell, R. E., et al. A monomeric red fluorescent protein. Proceedings of the National Academy of Sciences of the United States of America. 99 (12), 7877-7882 (2002).
- Cutrale, F., et al. Using enhanced number and brightness to measure protein oligomerization dynamics in live cells. Nature Protocols. 14 (2), 616-638 (2019).
- Dunsing, V., Chiantia, S. A Fluorescence Fluctuation Spectroscopy Assay of Protein-Protein Interactions at Cell-Cell Contacts. Journal of Visualized Experiments. (142), (2018).

