Aanvulling van splicingactiviteit door een Galectin-3 - U1 snRNP-complex op kralen
Summary
Dit artikel beschrijft de experimentele procedures voor (a) depletie van U1 snRNP uit nucleaire extracten, met gelijktijdig verlies van splicingactiviteit; en (b) reconstitutie van splicingactiviteit in het U1-uitgeputte extract door galectine-3 – U1 snRNP-deeltjes gebonden aan kralen covalent gekoppeld aan anti-galectine-3-antilichamen.
Abstract
Klassieke depletie-reconstitutie-experimenten geven aan dat galectine-3 een vereiste splicingfactor is in nucleaire extracten. Het mechanisme van opname van galectine-3 in de splicingroute wordt in dit artikel behandeld. Sedimentatie van HeLa-celkernextracten op 12%-32% glycerolgradiënten levert fracties op die verrijkt zijn in een endogeen ~10S-deeltje dat galectine-3 en U1 snRNP bevat. We beschrijven nu een protocol om nucleaire extracten van U1 snRNP uit te putten met gelijktijdig verlies van splicingactiviteit. Splicing-activiteit in het U1-uitgeputte extract kan worden gereconstitueerd door het galectine-3 – U1 snRNP-deeltje gevangen op agarose-kralen covalent gekoppeld aan anti-galectine-3-antilichamen. De resultaten geven aan dat het galectine-3 – U1 snRNP – pre-mRNA ternair complex een functioneel E-complex is dat leidt tot tussenproducten en producten van de splicingreactie en dat galectine-3 de splicingroute binnenkomt door zijn associatie met U1 snRNP. Het schema van het gebruik van complexen affiniteit- of immuno-geselecteerd op kralen om splicing activiteit te reconstitueren in extracten uitgeput van een specifieke splicing factor kan algemeen toepasbaar zijn op andere systemen.
Introduction
De productie van de meeste eukaryote boodschapper-RNA’s (mRNA’s) omvat verwijdering van introns en ligatie van exonen in een nucleair proces dat pre-mRNA-splicing wordt genoemd1. Twee klassen van RNA-eiwitcomplexen (RRP’s) sturen de verwerking van pre-messenger RNA tot volwassen mRNA via spliceosomale complexen. Eén klasse, ontluikende pre-messenger RRP’s, wordt co-transcriptioneel gevormd door de binding van heterogene nucleaire RNP-eiwitten en andere RNA-bindende eiwitten, waaronder enkele leden van de SR-familie, die hnRNP-complexen opleveren2. De tweede klasse, uracil-rijke kleine nucleaire RRP’s (U snRNPs met U1, U2, U4, U5 en U6 snRNAs) is geassocieerd met U-specifieke en kerneiwitten3,4. De U snRNPs interageren op een geordende manier met specifieke regio’s van pre-messenger RRP’s in een dynamische remodelleringsroute, terwijl introns worden weggesneden en exonen worden geligeerd om volwassen mRNPs5 te produceren. Veel extra nucleaire eiwitten nemen deel aan deze verwerkingsgebeurtenissen6.
Galectine-1 (Gal1) en galectine-3 (Gal3) zijn twee eiwitten die vereiste factoren zijn in de splicingroute, zoals aangetoond door depletie-reconstitutiestudies7,8. Verwijdering van beide galectines uit het splitsen van competente nucleaire extracten (NE) elimineert spliceosoomassemblage en splicingactiviteit in een vroeg stadium. Toevoeging van beide galectine aan zo’n dubbel uitgeput NE herstelt beide activiteiten. Gal1 en Gal3 zijn componenten van actieve spliceosomen zoals blijkt uit specifieke immunoprecipitatie van pre-mRNA, splicing intermediates en volwassen mRNA door antiserum specifiek voor Gal1 of Gal39. Belangrijk is dat Gal3 zich associeert met endogene U-snRNA die deeltjes in het NE bevatten buiten de splicingroute, zoals blijkt uit de precipitatie van snRNPs door anti-Gal3 antisera10. Ten slotte verandert het uitschakelen van Gal3 in HeLa-cellen de splicingpatronen van talrijke genen11.
In NE voorgeïncubeerd om voorgevormde spliceosomen12 te demonteren, worden snRNPs gevonden in meerdere complexen die sedimenteren in glycerolgradiënten van 7S tot groter dan 60S. Hoewel glycerolgradiëntfractionering een veelgebruikte techniek is voor de isolatie van spliceosomale complexen en componenten (zie bijvoorbeeld referenties13,14,15), hebben we deze methode uitgebreid door specifieke fracties te karakteriseren met behulp van antilichaamimmunoprecipitaties. Een snRNP-sedimentering op 10S bevat alleen U1-snRNA samen met Gal3. Immunoprecipitatie van de 10S-fractie met antisera specifiek voor Gal3 of U1 snRNP co-precipiteert zowel U1 als Gal3, wat aangeeft dat sommige van de U1 snRNP-monodeeltjes gebonden zijn aan Gal310. Aangezien U1 snRNP het eerste complex is dat bindt aan pre-mRNP in spliceosomale assemblage1,5, vertegenwoordigt deze stap een potentiële ingangsplaats voor Gal3 in de splicingroute. Op basis hiervan toonden we aan dat 10S Gal3-U1 snRNP monodeeltjes gebonden aan anti-Gal3 bevattende kralen de splicingactiviteit herstelden tot een U1 snRNP uitgeput NE, waardoor dit complex werd vastgesteld als één mechanisme waarmee Gal3 wordt gerekruteerd in de spliceosomale route16. Dit in tegenstelling tot pogingen om spliceosomen te isoleren in specifieke stadia van de splicingreactie en het catalogiseren van de geassocieerde factoren17,18. In dergelijke studies wordt de aanwezigheid van bepaalde factoren op een bepaald moment vastgesteld, maar niet het mechanisme waarmee ze werden geladen.
We hadden eerder in detail de bereiding van NE, het splicing substraat, de assemblage van het splicing reactiemengsel en de analyse van producten beschreven in onze documentatie van de rol van galectinen in pre-mRNA splicing19. We beschrijven nu de experimentele procedures voor fractionering van nucleaire extracten om een fractie verrijkt in Gal3 – U1 snRNP-complex te verkrijgen en voor immunoselectie van het laatste complex om splicingactiviteit te reconstitueren in een U1-uitgeput nucleair extract.
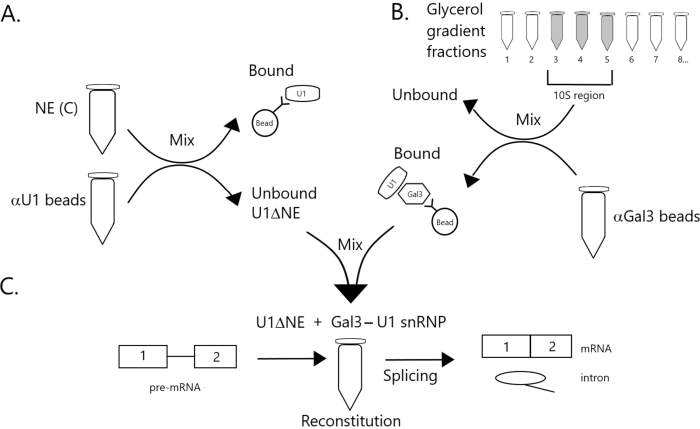
Figuur 1: Schematisch diagram ter illustratie van de complementatie van splicingactiviteit in nucleair extract uitgeput van U1 snRNP door een Gal3-U1 snRNP-complex op kralen. (A) NE in Buffer C (NE(C)) wordt geïncubeerd met eiwit A-Sepharose kralen covalent gekoppeld aan anti-U1 snRNP (αU1 kralen). De ongebonden fractie is uitgeput van U1 snRNP (U1ΔNE). (B) NE in Buffer D (NE(D)) wordt gefractioneerd over een glycerolgradiënt van 12%-32% door ultracentrifugatie. Fracties die overeenkomen met het 10S-gebied (fracties 3-5) worden gecombineerd en gemengd met kralen die covalent zijn gekoppeld aan anti-Gal3-antilichamen (αGal3-kralen). Het materiaal gebonden aan de kralen bevat een Gal3-U1 snRNP monodeeltje. (C) Het Gal3-U1 snRNP-complex uit deel (B) wordt gemengd met U1ΔNE uit deel (A) in een splicing-assay met behulp van 32P-gelabeld MINX pre-mRNA-substraat en de tussenproducten en producten van de splicingreactie worden geanalyseerd door gel-elektroforese en autoradiografie. Klik hier om een grotere versie van deze figuur te bekijken.
Protocol
Representative Results
Discussion
Dit rapport biedt de experimentele details die documenteren dat een Gal3 – U1 snRNP-complex gevangen op anti-Gal3-gecoate kralen kan binden aan pre-mRNA-substraat en dit ternaire complex kan splicingactiviteit herstellen tot een U1 snRNP-uitgeput NE. Gal3 is een lid van een familie van eiwitten die oorspronkelijk zijn geïsoleerd op basis van zijn galactose-specifieke koolhydraatbindende activiteit23 . Vroege immunofluorescentie- en subcellulaire fractioneringsstudies gaven de eerste hint van een …
Declarações
The authors have nothing to disclose.
Acknowledgements
Dit werk is ondersteund door National Science Foundation Grant MCB-0092919 en Michigan State University Intramural Research Grant 09-CDFP-2001 (aan RJP) en door National Institutes of Health Grant GM-38740 en Michigan AgBioResearch Project MICL02455 (aan JLW).
Het MINX pre-mRNA substraat dat werd gebruikt in de splicing assays was een vriendelijk geschenk van Dr. Susan Berget (Baylor College of Medicine, Houston, TX, VS).
Materials
| anti-U1 snRNP | The Binding Site | Hu ENA-RNP #33471 | human autoimmune serum specific for U1 snRNP |
| bottle top vacuum filter | Fisher Scientific | Corning 431153 (0.22 μm; PES 150 ml) | for filtering solutions containing Tris |
| centrifuge | International Equipment Company | IEC Model PR-6 | for pelletting Sepharose beads in immunoprecipitation |
| diethylpyrocarbonate (DEPC) | Sigma-Aldrich | 159220-5G | for treatment of water used in preparation of all solutions |
| dimethylpimelimidate (DMP) | Sigma-Aldrich | 80490-5G | for cross-linking antibody to Sepharose beads |
| electrophoresis cell | BioRad Laboratories, Inc | Mini-Protean II | for SDS-PAGE separation of proteins |
| ethanolamine | Sigma-Aldrich | 411000-100ml | for blocking after the cross-linking reaction |
| gel electrophoresis system | Hoefer, Inc | HSI SE 500 Series | for separating snRNAs by gel electrophoresis |
| gel slab dryer | BioRad | Model 224 | for drying gel slabs for autoradiography |
| Hybond ECL membrane | GE Healthcare | RPN3032D (0.2 μm; 30 cm x 3 m) | for immunoblotting of proteins on membrane |
| microdialyzer (12 x 100 μl sample capacity) | Pierce | Microdialyzer System 100 | for exchanging the buffer of nuclear extract |
| microdialyzer membranes (8K cutoff) | Pierce | 66310 | for exchanging the buffer of nuclear extract |
| non-fat dry milk | Spartan Stores | Spartan Instant Non-fat Dry Milk | |
| Protein A Sepharose CL-4B | Millipore-Sigma | GE 17-0780-01 | for coupling antibody to beads |
| Proteinase K | Millipore-Sigma | P2308-5mg | for stopping the splicing reaction to isolate the RNAs |
| RNasin | Promega | N2111 | for inhibiting ribonuclease activity |
| rocker/rotator | Lab Industries, Inc | Labquake Shaker 400-110 | for mixing protein solutions in coupling reactions and in immunoprecipitation |
| Safety-Solve | Research Products International Corp. | No. 111177 | scintillation counting cocktail for determination of radioactivity in splicing substrate |
| scintillation counter | Beckman Instruments | LS6000SC | scintillation counter for determination of radioactivity |
| speed vaccum concentrator | Savant | SVC 100H | for drying ethanol-precipitated RNA pellets |
| Transphor electrophoresis unit | Hoefer, Inc | Hoefer TE Series Transphor | for protein transfer from SDS-PAGE to blotting membrane |
Referências
- Hoskins, A. A., Moore, M. J. The spliceosome: a flexible, reversible macromolecular machine. Trends In Biochemical Sciences. 37, 179-188 (2012).
- Choi, Y. D., Grabowski, P., Sharp, P. A., Dreyfuss, G. Heterogeneous nuclear ribonucleoproteins: role in RNA splicing. Science. 231, 1534-1539 (1986).
- Lerner, M., Steitz, J. A. Snurps and scyrps. Cell. 25, 298-300 (1981).
- Maniatis, T., Reed, R. The role of small nuclear ribonucleoprotein particles in pre-mRNA splicing. Nature. 325, 673-678 (1987).
- Hoskins, A. A., et al. Ordered and dynamic assembly of single spliceosomes. Science. 331, 1289-1295 (2011).
- Coppin, L., Leclerc, J., Vincent, A., Porchet, N., Pigny, P. Messenger RNA life-cycle in cancer: emerging role of conventional and non-conventional RNA-binding proteins. International Journal of Molecular Sciences. 19, 650-676 (2018).
- Dagher, S. F., Wang, J. L., Patterson, R. J. Identification of galectin-3 as a factor in pre-mRNA splicing. Proceedings of the National Academy of Sciences of the United States of America. 92, 1213-1217 (1995).
- Vyakarnam, A., Dagher, S. F., Wang, J. L., Patterson, R. J. Evidence for a role for galectin-1 in pre-mRNA splicing. Molecular and Cellular Biology. 17, 4730-4737 (1997).
- Wang, W., Park, J. W., Wang, J. L., Patterson, R. J. Immunoprecipitation of spliceosomal RNAs by antisera to galectin-1 and galectin-3. Nucleic Acids Research. 34, 5166-5174 (2006).
- Haudek, K. C., Voss, P. G., Locascio, L. E., Wang, J. L., Patterson, R. J. A mechanism for incorporation of galectin-3 into the spliceosome through its association with U1 snRNP. Bioquímica. 48, 7705-7712 (2009).
- Fritsch, K., et al. Galectin-3 interacts with components of the nuclear ribonucleoprotein complex. BMC Cancer. 16, 502-511 (2016).
- Conway, G. C., Krainer, A. R., Spector, D. L., Roberts, R. J. Multiple splicing factors are released from endogenous complexes during in vitro pre-mRNA splicing. Molecular and Cellular Biology. 9, 5273-5280 (1989).
- Dery, K. J., Yean, S. L., Lin, R. J. Assembly and glycerol gradient isolation of yeast spliceosomes containing transcribed or synthetic U6 snRNA. Methods in Molecular Biology. 488, 41-63 (2008).
- Yoshimoto, R., Kataoka, N., Okawa, K., Ohno, M. Isolation and characterization of post-splicing lariat-intron complexes. Nucleic Acids Research. 37, 891-902 (2009).
- Malca, H., Shomron, N., Ast, G. The U1 snRNP base pairs with the 5′ splice site within a penta-snRNP complex. Molecular and Cellular Biology. 23, 3442-3455 (2003).
- Haudek, K. C., Voss, P. G., Wang, J. L., Patterson, R. J. A 10S galectin-3 – snRNP complex assembles into active spliceosomes. Nucleic Acids Research. 44, 6391-6397 (2016).
- Rappsilber, J., Ryder, U., Lamond, A. I., Mann, M. Large-scale proteomic analysis of the human spliceosome. Genome Research. 12, 1231-1245 (2002).
- Jurica, M. S., Moore, M. J. Capturing splicing complexes to study structure and mechanism. Methods. 28, 336-345 (2002).
- Patterson, R. J., Haudek, K. C., Voss, P. G., Wang, J. L. Examination of the role of galectins in pre-mRNA splicing. Methods in Molecular Biology. 1207, 431-449 (2015).
- Dignam, J. D., Lebovitz, R. M., Roeder, R. G. Accurate transcription initiation by RNA polymerase II in a soluble extract from isolated mammalian nuclei. Nucleic Acids Research. 11, 1475-1489 (1983).
- Agarwal, N., Sun, Q., Wang, S. Y., Wang, J. L. Carbohydrate-binding protein 35. I. Properties of the recombinant polypeptide and the individuality of the domains. Journal of Biological Chemistry. 268, 14932 (1993).
- Zillmann, M., Zapp, M. I., Berget, S. M. Gel electrophoretic isolation of splicing complexes containing U1 small nuclear ribonucleoprotein particles. Molecular and Cellular Biology. 8, 814-821 (1988).
- Barondes, S. H., et al. Galectins: a family of animal β-galactoside-binding proteins. Cell. 76, 597-598 (1994).
- Laing, J. G., Wang, J. L. Identification of carbohydrate binding protein 35 in heterogeneous nuclear ribonucleoprotein complex. Bioquímica. 27, 5329-5334 (1988).
- Vyakarnam, A., Lenneman, A. J., Lakkides, K. M., Patterson, R. J., Wang, J. L. A comparative nuclear localization study of galectin-1 with other splicing components. Experimental Cell Research. 242, 419-428 (1998).
- Michaud, S., Reed, R. An ATP-independent complex commits pre-mRNA to the mammalian spliceosome assembly pathway. Genes & Development. 5, 2534-2546 (1991).
- Chiu, Y. -. F., et al. Cwc25 is a novel splicing factor required after Prp2 and Yju2 to facilitate the first catalytic reaction. Molecular and Cellular Biology. 29, 5671-5678 (2009).
- Krishnan, R., et al. Biased Brownian ratcheting leads to pre-mRNA remodeling and capture prior to first-step splicing. Nature Structural and Molecular Biology. 20, 1450-1457 (2013).
- Gray, R. M., et al. Distinct effects on splicing of two monoclonal antibodies directed against the amino-terminal domain of galectin-3. Archives of Biochemistry and Biophysics. 475, 100-108 (2008).

