השלמת פעילות התרסיס על ידי מתחם גלקטין-3 - U1 snRNP על חרוזים
Summary
מאמר זה מתאר את ההליכים הניסיוניים עבור (א) דלדול של U1 snRNP מתמציות גרעיניות, עם אובדן במקביל של פעילות splicing; ותואר (ב) שחזור של פעילות ההצמדה בתמצית U1-מרוקנת על ידי חלקיקי גלקטין-3 – U1 snRNP הקשורים חרוזים בשילוב עם נוגדנים אנטי-גלקטין-3.
Abstract
ניסויים קלאסיים של דלדול-שחזור מצביעים על כך שגלקטין-3 הוא גורם שיתוף נדרש בתמציות גרעיניות. מנגנון ההשתלבות של גלקטין-3 במסלול ההשתלבות מטופל במאמר זה. משקעים של תמציות גרעיניות תא HeLa על 12%-32% שיפועי גליסול מניב שברים מועשרים חלקיק אנדוגני ~ 10S המכיל גלקטין-3 ו U1 snRNP. כעת אנו מתארים פרוטוקול לרוקן תמציות גרעיניות של U1 snRNP עם אובדן במקביל של פעילות חיבור. פעילות הצמדה בתמצית U1-מרוקנת ניתן ליישב מחדש על ידי חלקיק גלקטין-3 – U1 snRNP לכוד על חרוזי אגרוז covalented בשילוב עם נוגדנים אנטי גלקטין-3. התוצאות מצביעות על כך galectin-3 – U1 snRNP – מתחם טרנרי טרום mRNA הוא קומפלקס E פונקציונלי המוביל למתווכים ומוצרים של תגובת החיבור וכי galectin-3 נכנס למסלול החיבור באמצעות הקשר שלה עם U1 snRNP. התוכנית של שימוש במתחמים זיקה- או immuno-שנבחרו על חרוזים כדי לשחזר את פעילות ההשתתבות בתמציות מרוקן של גורם חרוז מסוים עשוי להיות ישים בדרך כלל למערכות אחרות.
Introduction
הייצור של רוב RNAs שליח eukaryotic (mRNAs) כרוך בהסרת introns וקשירה של exons בתהליך גרעיני המכונה חיבור לפני mRNA1. שני סוגים של מתחמי חלבון RNA (RNPs) מכוונים את העיבוד של RNA לפני השליח ל- mRNA בוגר באמצעות מתחמים spliceosomal. מחלקה אחת, RNPs טרום שליח המתהווה, נוצרת שיתוף תמלול על ידי כריכה של חלבונים גרעיניים הטרוגניים וחלבונים אחרים מחייבים RNA, כולל כמה מבני משפחת SR, מניב מתחמי hnRNP2. ה-RNPs הגרעיניים הקטנים העשירים בארסיל (U snRNPs עם U1, U2, U4, U5 ו-U6 snRNAs) קשורים לחלבונים ספציפיים ל-U ולחלבוני ליבה3,4. ה- snRNPs של ארה”ב מתקשרים באופן מסודר עם אזורים ספציפיים של RNPs לפני השליח במסלול שיפוץ דינמי כאשר אינטרונים נקטעים ואקסונים קשורים כדי לייצר mRNPs5 בוגרים. חלבונים גרעיניים רבים נוספים משתתפים באירועי עיבוד אלה6.
גלקטין-1 (Gal1) וגלקטין-3 (Gal3) הם שני חלבונים הנדרשים במסלול ההשתלבות כפי שמוצג על ידי מחקרי דלדול-שיקום7,8. הסרת שני הגלקטינים מליזוב תמציות גרעיניות מוסמכות (NE) מבטלת את ההרכבה המשולבת ואת פעילות ההשתלבות בשלב מוקדם. תוספת של כל גלקטין ל- NE מרוקן כפליים משחזרת את שתי הפעילויות. Gal1 ו- Gal3 הם מרכיבים של spliceosomes פעיל כפי שמעידים אימונופרציפיטציה ספציפית של טרום mRNA, שילוב ביניים, ו- mRNA בוגר על ידי אנטי-ספירום ספציפי עבור Gal1 או Gal39. חשוב לציין, Gal3 מקשר עם snRNA U אנדוגני המכיל חלקיקים ב- NE מחוץ למסלול ההשתלבות כפי שמוצג על ידי משקעים של snRNPs על ידי אנטי-Gal3 antisera10. לבסוף, השתקה של Gal3 בתאי HeLa משנה דפוסי שכפול של גנים רבים11.
ב- NE דגירה מראש כדי לפרק spliceosomes preformed12, snRNPs נמצאים במתחמים מרובים המשקעים בשיפועים גליצריל מ 7S ליותר מ 60S. למרות שבר הדרגתי גליצול היא טכניקה נפוצה לבידוד של מתחמים ורכיבים spliceosomal (ראה הפניות13,14,15 למשל), הרחבנו שיטה זו על ידי אפיון שברים ספציפיים באמצעות חיסונים נוגדנים. משקעים snRNP ב- 10S מכיל רק U1 snRNA יחד עם Gal3. אימונופרציפיטציה של שבר 10S עם אנטי-רה ספציפית עבור Gal3 או U1 snRNP שיתוף מזרז הן U1 והן Gal3 המציין כמה חלקיקים U1 snRNP קשורים Gal310. מכיוון ש- U1 snRNP הוא המתחם הראשון שנקשר ל- pre-mRNP בהרכבה spliceosomal1,5, שלב זה מייצג אתר כניסה פוטנציאלי עבור Gal3 למסלול ההשתתפות. על בסיס זה, הראינו כי 10S Gal3-U1 snRNP מונו-חלקיקים הקשורים אנטי-Gal3 המכיל חרוזים משוחזרים פעילות splicing ל- U1 snRNP מרוקן NE, הקמת מורכב זה כמנגנון אחד שבאמצעותו Gal3 מגויס לתוך המסלול spliceosomal16. זאת בניגוד לניסיונות לבודד את התרסיסים בשלבים ספציפיים בתגובת ההשתלבות ולקטלג את הגורמים הקשורים 17,18. במחקרים כאלה, נוכחותם של גורמים מסוימים בנקודת זמן כלשהי מובהר אך לא המנגנון שבאמצעותו הם נטענו.
תיארנו בעבר בפירוט את הכנת NE, את מצע ההשתלבות, את ההרכבה של תערובת התגובה המשתלבת, ואת הניתוח של מוצרים בתיעוד שלנו של תפקיד הגלקטינים ב- splicing לפני mRNA19. כעת אנו מתארים את ההליכים הניסיוניים לפירוק תמציות גרעיניות כדי להשיג שבר מועשר במתחם Gal3 – U1 snRNP ולבחירה חיסונית של המתחם האחרון כדי לחדש את פעילות ההכלאה בתמצית גרעינית מרוקנת U1.
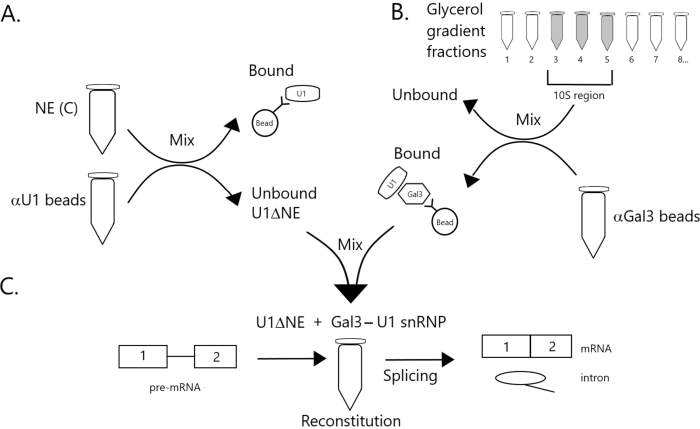
איור 1: תרשים סכמטי הממחיש את ההשלמה של פעילות ההצמדה בתמצית גרעינית מרוקנת מ- U1 snRNP על ידי קומפלקס Gal3-U1 snRNP על חרוזים. (A) NE במאגר C (NE(C)) הוא דגירה עם חרוזי חלבון A-Sepharose בשילוב עם נוגדי U1 snRNP (חרוזים αU1). השבר הלא מאוגד מתרוקן מ- U1 snRNP (U1ΔNE). (B) NE במאגר D (NE(D)) מחולק מעבר הדרגתי של 12%-32% גליצריל על-ידי אולטרה-צנטריפוגה. שברים המתאימים לאזור 10S (שברים 3-5) משולבים ומעורבבים עם חרוזים בשילוב עם נוגדנים נגד Gal3 (חרוזים αGal3). החומר הקשור החרוזים מכיל מונופרקל SnRNP 3-U1. (ג) מתחם Gal3-U1 snRNP מחלק (B) מעורבב עם U1ΔNE מחלק (A) בבדיקה משולבת באמצעות מצע קדם-mRNA של MINX עם תווית 32P והמתווכים והמוצרים של תגובת ההצמדה מנותחים על ידי אלקטרופורזה ג’ל ואוטרדיוגרפיה. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
Protocol
Representative Results
Discussion
דו”ח זה מספק את הפרטים הניסיוניים המתעדים מתחם Gal3 – U1 snRNP הלכוד על חרוזים מצופים אנטי-Gal3 יכול להיקשר למצע טרום mRNA ומתחם טרנרי זה יכול לשחזר את פעילות החיבור ל- NE מרוקן U1 snRNP. Gal3 הוא חבר במשפחת חלבונים מבודדים במקור על בסיס פעילות מחייבת הפחמימות הספציפית לגלקטוז23 . מחקרי אימונופל?…
Declarações
The authors have nothing to disclose.
Acknowledgements
עבודה זו נתמכה על ידי הקרן הלאומית למדע גרנט MCB-0092919 ומענק מחקר פנים-ארצי של אוניברסיטת מישיגן 09-CDFP-2001 (ל- RJP) ועל ידי המכונים הלאומיים לבריאות גרנט GM-38740 ופרויקט MICL02455 של מישיגן AgBioResearch (ל- JLW).
מצע הקדם-mRNA של MINX ששימש בבדיקות ההסתבכות היה מתנה חביבה מד”ר סוזן ברגט (מכללת ביילור לרפואה, יוסטון, TX, ארה”ב).
Materials
| anti-U1 snRNP | The Binding Site | Hu ENA-RNP #33471 | human autoimmune serum specific for U1 snRNP |
| bottle top vacuum filter | Fisher Scientific | Corning 431153 (0.22 μm; PES 150 ml) | for filtering solutions containing Tris |
| centrifuge | International Equipment Company | IEC Model PR-6 | for pelletting Sepharose beads in immunoprecipitation |
| diethylpyrocarbonate (DEPC) | Sigma-Aldrich | 159220-5G | for treatment of water used in preparation of all solutions |
| dimethylpimelimidate (DMP) | Sigma-Aldrich | 80490-5G | for cross-linking antibody to Sepharose beads |
| electrophoresis cell | BioRad Laboratories, Inc | Mini-Protean II | for SDS-PAGE separation of proteins |
| ethanolamine | Sigma-Aldrich | 411000-100ml | for blocking after the cross-linking reaction |
| gel electrophoresis system | Hoefer, Inc | HSI SE 500 Series | for separating snRNAs by gel electrophoresis |
| gel slab dryer | BioRad | Model 224 | for drying gel slabs for autoradiography |
| Hybond ECL membrane | GE Healthcare | RPN3032D (0.2 μm; 30 cm x 3 m) | for immunoblotting of proteins on membrane |
| microdialyzer (12 x 100 μl sample capacity) | Pierce | Microdialyzer System 100 | for exchanging the buffer of nuclear extract |
| microdialyzer membranes (8K cutoff) | Pierce | 66310 | for exchanging the buffer of nuclear extract |
| non-fat dry milk | Spartan Stores | Spartan Instant Non-fat Dry Milk | |
| Protein A Sepharose CL-4B | Millipore-Sigma | GE 17-0780-01 | for coupling antibody to beads |
| Proteinase K | Millipore-Sigma | P2308-5mg | for stopping the splicing reaction to isolate the RNAs |
| RNasin | Promega | N2111 | for inhibiting ribonuclease activity |
| rocker/rotator | Lab Industries, Inc | Labquake Shaker 400-110 | for mixing protein solutions in coupling reactions and in immunoprecipitation |
| Safety-Solve | Research Products International Corp. | No. 111177 | scintillation counting cocktail for determination of radioactivity in splicing substrate |
| scintillation counter | Beckman Instruments | LS6000SC | scintillation counter for determination of radioactivity |
| speed vaccum concentrator | Savant | SVC 100H | for drying ethanol-precipitated RNA pellets |
| Transphor electrophoresis unit | Hoefer, Inc | Hoefer TE Series Transphor | for protein transfer from SDS-PAGE to blotting membrane |
Referências
- Hoskins, A. A., Moore, M. J. The spliceosome: a flexible, reversible macromolecular machine. Trends In Biochemical Sciences. 37, 179-188 (2012).
- Choi, Y. D., Grabowski, P., Sharp, P. A., Dreyfuss, G. Heterogeneous nuclear ribonucleoproteins: role in RNA splicing. Science. 231, 1534-1539 (1986).
- Lerner, M., Steitz, J. A. Snurps and scyrps. Cell. 25, 298-300 (1981).
- Maniatis, T., Reed, R. The role of small nuclear ribonucleoprotein particles in pre-mRNA splicing. Nature. 325, 673-678 (1987).
- Hoskins, A. A., et al. Ordered and dynamic assembly of single spliceosomes. Science. 331, 1289-1295 (2011).
- Coppin, L., Leclerc, J., Vincent, A., Porchet, N., Pigny, P. Messenger RNA life-cycle in cancer: emerging role of conventional and non-conventional RNA-binding proteins. International Journal of Molecular Sciences. 19, 650-676 (2018).
- Dagher, S. F., Wang, J. L., Patterson, R. J. Identification of galectin-3 as a factor in pre-mRNA splicing. Proceedings of the National Academy of Sciences of the United States of America. 92, 1213-1217 (1995).
- Vyakarnam, A., Dagher, S. F., Wang, J. L., Patterson, R. J. Evidence for a role for galectin-1 in pre-mRNA splicing. Molecular and Cellular Biology. 17, 4730-4737 (1997).
- Wang, W., Park, J. W., Wang, J. L., Patterson, R. J. Immunoprecipitation of spliceosomal RNAs by antisera to galectin-1 and galectin-3. Nucleic Acids Research. 34, 5166-5174 (2006).
- Haudek, K. C., Voss, P. G., Locascio, L. E., Wang, J. L., Patterson, R. J. A mechanism for incorporation of galectin-3 into the spliceosome through its association with U1 snRNP. Bioquímica. 48, 7705-7712 (2009).
- Fritsch, K., et al. Galectin-3 interacts with components of the nuclear ribonucleoprotein complex. BMC Cancer. 16, 502-511 (2016).
- Conway, G. C., Krainer, A. R., Spector, D. L., Roberts, R. J. Multiple splicing factors are released from endogenous complexes during in vitro pre-mRNA splicing. Molecular and Cellular Biology. 9, 5273-5280 (1989).
- Dery, K. J., Yean, S. L., Lin, R. J. Assembly and glycerol gradient isolation of yeast spliceosomes containing transcribed or synthetic U6 snRNA. Methods in Molecular Biology. 488, 41-63 (2008).
- Yoshimoto, R., Kataoka, N., Okawa, K., Ohno, M. Isolation and characterization of post-splicing lariat-intron complexes. Nucleic Acids Research. 37, 891-902 (2009).
- Malca, H., Shomron, N., Ast, G. The U1 snRNP base pairs with the 5′ splice site within a penta-snRNP complex. Molecular and Cellular Biology. 23, 3442-3455 (2003).
- Haudek, K. C., Voss, P. G., Wang, J. L., Patterson, R. J. A 10S galectin-3 – snRNP complex assembles into active spliceosomes. Nucleic Acids Research. 44, 6391-6397 (2016).
- Rappsilber, J., Ryder, U., Lamond, A. I., Mann, M. Large-scale proteomic analysis of the human spliceosome. Genome Research. 12, 1231-1245 (2002).
- Jurica, M. S., Moore, M. J. Capturing splicing complexes to study structure and mechanism. Methods. 28, 336-345 (2002).
- Patterson, R. J., Haudek, K. C., Voss, P. G., Wang, J. L. Examination of the role of galectins in pre-mRNA splicing. Methods in Molecular Biology. 1207, 431-449 (2015).
- Dignam, J. D., Lebovitz, R. M., Roeder, R. G. Accurate transcription initiation by RNA polymerase II in a soluble extract from isolated mammalian nuclei. Nucleic Acids Research. 11, 1475-1489 (1983).
- Agarwal, N., Sun, Q., Wang, S. Y., Wang, J. L. Carbohydrate-binding protein 35. I. Properties of the recombinant polypeptide and the individuality of the domains. Journal of Biological Chemistry. 268, 14932 (1993).
- Zillmann, M., Zapp, M. I., Berget, S. M. Gel electrophoretic isolation of splicing complexes containing U1 small nuclear ribonucleoprotein particles. Molecular and Cellular Biology. 8, 814-821 (1988).
- Barondes, S. H., et al. Galectins: a family of animal β-galactoside-binding proteins. Cell. 76, 597-598 (1994).
- Laing, J. G., Wang, J. L. Identification of carbohydrate binding protein 35 in heterogeneous nuclear ribonucleoprotein complex. Bioquímica. 27, 5329-5334 (1988).
- Vyakarnam, A., Lenneman, A. J., Lakkides, K. M., Patterson, R. J., Wang, J. L. A comparative nuclear localization study of galectin-1 with other splicing components. Experimental Cell Research. 242, 419-428 (1998).
- Michaud, S., Reed, R. An ATP-independent complex commits pre-mRNA to the mammalian spliceosome assembly pathway. Genes & Development. 5, 2534-2546 (1991).
- Chiu, Y. -. F., et al. Cwc25 is a novel splicing factor required after Prp2 and Yju2 to facilitate the first catalytic reaction. Molecular and Cellular Biology. 29, 5671-5678 (2009).
- Krishnan, R., et al. Biased Brownian ratcheting leads to pre-mRNA remodeling and capture prior to first-step splicing. Nature Structural and Molecular Biology. 20, 1450-1457 (2013).
- Gray, R. M., et al. Distinct effects on splicing of two monoclonal antibodies directed against the amino-terminal domain of galectin-3. Archives of Biochemistry and Biophysics. 475, 100-108 (2008).

