Komplementering av skjøteaktivitet av et Galectin-3 - U1 snRNP-kompleks på perler
Summary
Denne artikkelen beskriver de eksperimentelle prosedyrene for (a) uttømming av U1 snRNP fra kjernefysiske ekstrakter, med samtidig tap av skjøteaktivitet; og (b) rekonstituering av skjøteaktivitet i U1-utarmet ekstrakt ved galectin-3 – U1 snRNP partikler bundet til perler kovalent kombinert med anti-galectin-3 antistoffer.
Abstract
Klassiske uttømmingsrekonstitueringsforsøk indikerer at galectin-3 er en nødvendig skjøtefaktor i atomekstrakter. Mekanismen for inkorporering av galectin-3 i skjøtebanen er adressert i dette papiret. Sedimentering av HeLa celle kjernefysiske ekstrakter på 12%-32% glyserol gradienter gir fraksjoner beriket i en endogen ~ 10S partikkel som inneholder galectin-3 og U1 snRNP. Vi beskriver nå en protokoll for å tømme kjernefysiske ekstrakter av U1 snRNP med samtidig tap av skjøteaktivitet. Skjøteaktivitet i U1-utarmet ekstrakt kan rekonstitueres av galectin-3 – U1 snRNP partikkel fanget på agarose perler kovalent kombinert med anti-galectin-3 antistoffer. Resultatene indikerer at galectin-3 – U1 snRNP – pre-mRNA ternary komplekset er et funksjonelt E-kompleks som fører til mellomprodukter og produkter av skjøtereaksjonen, og at galectin-3 kommer inn i skjøteveien gjennom sin tilknytning til U1 snRNP. Ordningen med å bruke kompleks affinitet- eller immuno-valgt på perler for å rekonstituere skjøteaktivitet i ekstrakter utarmet av en bestemt skjøtefaktor kan generelt gjelde for andre systemer.
Introduction
Produksjon av de fleste eukaryote budbringere RNAer (mRNAer) innebærer fjerning av introner og ligasjon av eksoner i en kjernefysisk prosess kalt pre-mRNA skjøting1. To klasser av RNA-proteinkomplekser (RNPer) styrer behandlingen av pre-messenger RNA til moden mRNA via spleiseosomale komplekser. En klasse, nascent pre-messenger RNPs, dannes co-transkripsjonelt ved binding av heterogene kjernefysiske RNP-proteiner og andre RNA-bindende proteiner, inkludert noen medlemmer av SR-familien, noe som gir hnRNP-komplekser2. Den andre klassen, uracil-rike små kjernefysiske RNPer (U snRNPer med U1, U2, U4, U5 og U6 snRNAs) er forbundet med U-spesifikke og kjerneproteiner3,4. De amerikanske snRNPene samhandler på en ordnet måte med spesifikke regioner av pre-messenger RNPer i en dynamisk ombyggingsvei som introner utskilles og exons er ligated å produsere modne mRNPs5. Mange andre kjerneproteiner deltar i disse behandlingshendelsene6.
Galectin-1 (Gal1) og galectin-3 (Gal3) er to proteiner som er nødvendige faktorer i skjøteveien som vist ved uttømming-rekonstitueringsstudier7,8. Fjerning av begge galectins fra skjøting kompetente kjernefysiske ekstrakter (NE) avskaffer skjøteosom montering og skjøting aktivitet på et tidlig trinn. Tillegg av begge galectin til en slik dobbelt utarmet NE gjenoppretter begge aktivitetene. Gal1 og Gal3 er komponenter av aktive skjøteosomer som det fremgår av spesifikk immunoprecipitation av pre-mRNA, skjøting mellomprodukter, og moden mRNA ved antiserum spesifikk for enten Gal1 eller Gal39. Viktigst, Gal3 forbinder med endogene U snRNA inneholder partikler i NE utenfor skjøteveien som vist ved nedbør av snRNPs av anti-Gal3 antisera10. Til slutt endrer silencing av Gal3 i HeLa-celler skjøtemønstre av mange gener11.
I NE pre-inkubert for å demontere preformede skjøteosomer12, snRNPs finnes i flere komplekser sedimentering i glyserol gradienter fra 7S til større enn 60S. Selv om glyserol gradient fraksjonering er en vanlig teknikk for isolering av spleiseosomale komplekser og komponenter (se referanser13,14,15 for eksempel), har vi utvidet denne metoden ved å karakterisere spesifikke fraksjoner ved hjelp av antistoff immunoprecipitations. En snRNP sedimentering ved 10S inneholder bare U1 snRNA sammen med Gal3. Immunoprecipitation av 10S fraksjonen med antisera spesifikk for Gal3 eller U1 snRNP co-utfelling både U1 og Gal3 indikerer noen av U1 snRNP monopartikler er bundet til Gal310. Siden U1 snRNP er det første komplekset som binder seg til pre-mRNP i spleiseosomal montering1,5, representerer dette trinnet et potensielt inngangssted for Gal3 inn i skjøtebanen. På dette grunnlaget viste vi at 10S Gal3-U1 snRNP monopartikler bundet til anti-Gal3 som inneholder perler restaurert skjøteaktivitet til en U1 snRNP utarmet NE, og etablerer dette komplekset som en mekanisme som Gal3 rekrutteres til den spleiseosomale banen16. Dette står i kontrast til forsøk på å isolere skjøteosomer på bestemte stadier i skjøtereaksjonen og katalogisere de tilhørende faktorene17,18. I slike studier er tilstedeværelsen av visse faktorer på et tidspunkt fastslått, men ikke mekanismen de ble lastet på.
Vi hadde tidligere beskrevet i detalj utarbeidelsen av NE, skjøteunderlaget, monteringen av skjøtereaksjonsblandingen og analysen av produkter i vår dokumentasjon av galektinenes rolle i pre-mRNA skjøting19. Vi beskriver nå de eksperimentelle prosedyrene for fraksjonering av atomekstrakter for å oppnå en brøkdel beriket i Gal3 – U1 snRNP-kompleks og for immunvalg av sistnevnte kompleks for å rekonstituere skjøteaktivitet i et U1-utarmet atomekstrakt.
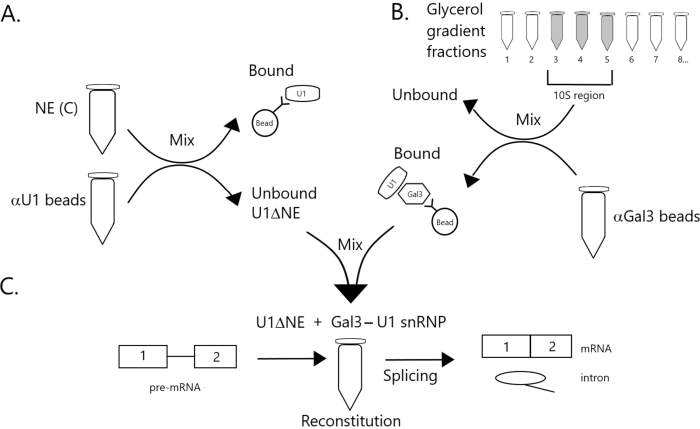
Figur 1: Skjematisk diagram som illustrerer komplementering av skjøteaktivitet i kjernefysisk ekstrakt utarmet av U1 snRNP av et Gal3-U1 snRNP-kompleks på perler. (A) NE i Buffer C (NE(C)) inkuberes med Protein A-Sepharose perler kovalent kombinert med anti-U1 snRNP (αU1 perler). Den ubundne brøkdelen er utarmet av U1 snRNP (U1ΔNE). (B) NE i buffer D (NE(D)) er fraksjonert over en 12%-32% glyserol gradient ved ultracentrifugation. Fraksjoner som tilsvarer 10S-regionen (brøker 3-5) kombineres og blandes med perler kovalent kombinert med anti-Gal3 antistoffer (αGal3 perler). Materialet bundet til perlene inneholder en Gal3-U1 snRNP monopartikkel. (C) Gal3-U1 snRNP-komplekset fra part (B) blandes med U1ΔNE fra del (A) i en skjøteanalyse ved hjelp av 32P-merket MINX pre-mRNA-substrat og mellomprodukter og produkter av skjøtereaksjonen analyseres ved gelelektroforese og autoradiografi. Klikk her for å se en større versjon av denne figuren.
Protocol
Representative Results
Discussion
Denne rapporten gir de eksperimentelle detaljene som dokumenterer et Gal3 – U1 snRNP-kompleks fanget på anti-Gal3-belagte perler kan binde seg til pre-mRNA-substrat, og dette ternære komplekset kan gjenopprette skjøteaktivitet til en U1 snRNP-utarmet NE. Gal3 er et medlem av en familie av proteiner som opprinnelig var isolert på grunnlag av sin galaktosespesifikke karbohydratbindingsaktivitet23 . Tidlig immunfluorescens og subcellulære fraksjoneringsstudier ga det første hintet om en assosia…
Declarações
The authors have nothing to disclose.
Acknowledgements
Dette arbeidet har blitt støttet av National Science Foundation Grant MCB-0092919 og Michigan State University Intramural Research Grant 09-CDFP-2001 (til RJP) og av National Institutes of Health Grant GM-38740 og Michigan AgBioResearch Project MICL02455 (til JLW).
MINX pre-mRNA-substratet som ble brukt i skjøteanalysene var en snill gave fra Dr. Susan Berget (Baylor College of Medicine, Houston, TX, USA).
Materials
| anti-U1 snRNP | The Binding Site | Hu ENA-RNP #33471 | human autoimmune serum specific for U1 snRNP |
| bottle top vacuum filter | Fisher Scientific | Corning 431153 (0.22 μm; PES 150 ml) | for filtering solutions containing Tris |
| centrifuge | International Equipment Company | IEC Model PR-6 | for pelletting Sepharose beads in immunoprecipitation |
| diethylpyrocarbonate (DEPC) | Sigma-Aldrich | 159220-5G | for treatment of water used in preparation of all solutions |
| dimethylpimelimidate (DMP) | Sigma-Aldrich | 80490-5G | for cross-linking antibody to Sepharose beads |
| electrophoresis cell | BioRad Laboratories, Inc | Mini-Protean II | for SDS-PAGE separation of proteins |
| ethanolamine | Sigma-Aldrich | 411000-100ml | for blocking after the cross-linking reaction |
| gel electrophoresis system | Hoefer, Inc | HSI SE 500 Series | for separating snRNAs by gel electrophoresis |
| gel slab dryer | BioRad | Model 224 | for drying gel slabs for autoradiography |
| Hybond ECL membrane | GE Healthcare | RPN3032D (0.2 μm; 30 cm x 3 m) | for immunoblotting of proteins on membrane |
| microdialyzer (12 x 100 μl sample capacity) | Pierce | Microdialyzer System 100 | for exchanging the buffer of nuclear extract |
| microdialyzer membranes (8K cutoff) | Pierce | 66310 | for exchanging the buffer of nuclear extract |
| non-fat dry milk | Spartan Stores | Spartan Instant Non-fat Dry Milk | |
| Protein A Sepharose CL-4B | Millipore-Sigma | GE 17-0780-01 | for coupling antibody to beads |
| Proteinase K | Millipore-Sigma | P2308-5mg | for stopping the splicing reaction to isolate the RNAs |
| RNasin | Promega | N2111 | for inhibiting ribonuclease activity |
| rocker/rotator | Lab Industries, Inc | Labquake Shaker 400-110 | for mixing protein solutions in coupling reactions and in immunoprecipitation |
| Safety-Solve | Research Products International Corp. | No. 111177 | scintillation counting cocktail for determination of radioactivity in splicing substrate |
| scintillation counter | Beckman Instruments | LS6000SC | scintillation counter for determination of radioactivity |
| speed vaccum concentrator | Savant | SVC 100H | for drying ethanol-precipitated RNA pellets |
| Transphor electrophoresis unit | Hoefer, Inc | Hoefer TE Series Transphor | for protein transfer from SDS-PAGE to blotting membrane |
Referências
- Hoskins, A. A., Moore, M. J. The spliceosome: a flexible, reversible macromolecular machine. Trends In Biochemical Sciences. 37, 179-188 (2012).
- Choi, Y. D., Grabowski, P., Sharp, P. A., Dreyfuss, G. Heterogeneous nuclear ribonucleoproteins: role in RNA splicing. Science. 231, 1534-1539 (1986).
- Lerner, M., Steitz, J. A. Snurps and scyrps. Cell. 25, 298-300 (1981).
- Maniatis, T., Reed, R. The role of small nuclear ribonucleoprotein particles in pre-mRNA splicing. Nature. 325, 673-678 (1987).
- Hoskins, A. A., et al. Ordered and dynamic assembly of single spliceosomes. Science. 331, 1289-1295 (2011).
- Coppin, L., Leclerc, J., Vincent, A., Porchet, N., Pigny, P. Messenger RNA life-cycle in cancer: emerging role of conventional and non-conventional RNA-binding proteins. International Journal of Molecular Sciences. 19, 650-676 (2018).
- Dagher, S. F., Wang, J. L., Patterson, R. J. Identification of galectin-3 as a factor in pre-mRNA splicing. Proceedings of the National Academy of Sciences of the United States of America. 92, 1213-1217 (1995).
- Vyakarnam, A., Dagher, S. F., Wang, J. L., Patterson, R. J. Evidence for a role for galectin-1 in pre-mRNA splicing. Molecular and Cellular Biology. 17, 4730-4737 (1997).
- Wang, W., Park, J. W., Wang, J. L., Patterson, R. J. Immunoprecipitation of spliceosomal RNAs by antisera to galectin-1 and galectin-3. Nucleic Acids Research. 34, 5166-5174 (2006).
- Haudek, K. C., Voss, P. G., Locascio, L. E., Wang, J. L., Patterson, R. J. A mechanism for incorporation of galectin-3 into the spliceosome through its association with U1 snRNP. Bioquímica. 48, 7705-7712 (2009).
- Fritsch, K., et al. Galectin-3 interacts with components of the nuclear ribonucleoprotein complex. BMC Cancer. 16, 502-511 (2016).
- Conway, G. C., Krainer, A. R., Spector, D. L., Roberts, R. J. Multiple splicing factors are released from endogenous complexes during in vitro pre-mRNA splicing. Molecular and Cellular Biology. 9, 5273-5280 (1989).
- Dery, K. J., Yean, S. L., Lin, R. J. Assembly and glycerol gradient isolation of yeast spliceosomes containing transcribed or synthetic U6 snRNA. Methods in Molecular Biology. 488, 41-63 (2008).
- Yoshimoto, R., Kataoka, N., Okawa, K., Ohno, M. Isolation and characterization of post-splicing lariat-intron complexes. Nucleic Acids Research. 37, 891-902 (2009).
- Malca, H., Shomron, N., Ast, G. The U1 snRNP base pairs with the 5′ splice site within a penta-snRNP complex. Molecular and Cellular Biology. 23, 3442-3455 (2003).
- Haudek, K. C., Voss, P. G., Wang, J. L., Patterson, R. J. A 10S galectin-3 – snRNP complex assembles into active spliceosomes. Nucleic Acids Research. 44, 6391-6397 (2016).
- Rappsilber, J., Ryder, U., Lamond, A. I., Mann, M. Large-scale proteomic analysis of the human spliceosome. Genome Research. 12, 1231-1245 (2002).
- Jurica, M. S., Moore, M. J. Capturing splicing complexes to study structure and mechanism. Methods. 28, 336-345 (2002).
- Patterson, R. J., Haudek, K. C., Voss, P. G., Wang, J. L. Examination of the role of galectins in pre-mRNA splicing. Methods in Molecular Biology. 1207, 431-449 (2015).
- Dignam, J. D., Lebovitz, R. M., Roeder, R. G. Accurate transcription initiation by RNA polymerase II in a soluble extract from isolated mammalian nuclei. Nucleic Acids Research. 11, 1475-1489 (1983).
- Agarwal, N., Sun, Q., Wang, S. Y., Wang, J. L. Carbohydrate-binding protein 35. I. Properties of the recombinant polypeptide and the individuality of the domains. Journal of Biological Chemistry. 268, 14932 (1993).
- Zillmann, M., Zapp, M. I., Berget, S. M. Gel electrophoretic isolation of splicing complexes containing U1 small nuclear ribonucleoprotein particles. Molecular and Cellular Biology. 8, 814-821 (1988).
- Barondes, S. H., et al. Galectins: a family of animal β-galactoside-binding proteins. Cell. 76, 597-598 (1994).
- Laing, J. G., Wang, J. L. Identification of carbohydrate binding protein 35 in heterogeneous nuclear ribonucleoprotein complex. Bioquímica. 27, 5329-5334 (1988).
- Vyakarnam, A., Lenneman, A. J., Lakkides, K. M., Patterson, R. J., Wang, J. L. A comparative nuclear localization study of galectin-1 with other splicing components. Experimental Cell Research. 242, 419-428 (1998).
- Michaud, S., Reed, R. An ATP-independent complex commits pre-mRNA to the mammalian spliceosome assembly pathway. Genes & Development. 5, 2534-2546 (1991).
- Chiu, Y. -. F., et al. Cwc25 is a novel splicing factor required after Prp2 and Yju2 to facilitate the first catalytic reaction. Molecular and Cellular Biology. 29, 5671-5678 (2009).
- Krishnan, R., et al. Biased Brownian ratcheting leads to pre-mRNA remodeling and capture prior to first-step splicing. Nature Structural and Molecular Biology. 20, 1450-1457 (2013).
- Gray, R. M., et al. Distinct effects on splicing of two monoclonal antibodies directed against the amino-terminal domain of galectin-3. Archives of Biochemistry and Biophysics. 475, 100-108 (2008).

