DNA-spanningsondes om de voorbijgaande piconewtonreceptorkrachten door immuuncellen in kaart te brengen
Summary
Dit artikel beschrijft een gedetailleerd protocol voor het gebruik van DNA-gebaseerde spanningsondes om de receptorkrachten in beeld te brengen die door immuuncellen worden toegepast. Deze aanpak kan receptorkrachten >4,7pN in realtime in kaart brengen en krachten in de loop van de tijd integreren.
Abstract
Mechanische krachten die worden overgedragen op de kruising tussen twee naburige cellen en op de kruising tussen cellen en de extracellulaire matrix zijn van cruciaal belang voor het reguleren van vele processen, variërend van ontwikkeling tot immunologie. Daarom is het ontwikkelen van de tools om deze krachten op moleculaire schaal te bestuderen van cruciaal belang. Onze groep ontwikkelde een reeks moleculaire spanningssensoren om de krachten te kwantificeren en te visualiseren die door cellen worden gegenereerd en naar specifieke liganden worden verzonden. De meest gevoelige klasse van moleculaire spanningssensoren bestaat uit nucleïnezuur stam-loop haarspelden. Deze sensoren gebruiken fluorofoor-quencher-paren om te rapporteren over de mechanische uitbreiding en ontvouwing van DNA-haarspelden onder kracht. Een uitdaging met DNA-haarspeldspanningssensoren is dat ze omkeerbaar zijn met snelle haarspeldhervouwing bij beëindiging van de spanning en dus zijn voorbijgaande krachten moeilijk te registreren. In dit artikel beschrijven we de protocollen voor het voorbereiden van DNA-spanningssensoren die kunnen worden “vergrendeld” en voorkomen dat ze opnieuw worden gevouwen om “opslag” van mechanische informatie mogelijk te maken. Dit maakt het mogelijk om zeer voorbijgaande piconewtonkrachten te registreren, die vervolgens kunnen worden “gewist” door de toevoeging van complementaire nucleïnezuren die het slot verwijderen. Dit vermogen om te schakelen tussen real-time spanningskartering en mechanische informatieopslag onthult zwakke, kortstondige en minder overvloedige krachten, die vaak worden gebruikt door T-cellen als onderdeel van hun immuunfuncties.
Introduction
Immuuncellen verdedigen zich tegen ziekteverwekkers en kankercellen door voortdurend de oppervlakken van doelcellen te kruipen en te scannen op antigenen, waarbij hun oppervlak 1,2 wordt gestudeerd. Antigeenherkenning wordt geïnitieerd na binding tussen de T-celreceptor (TCR) en het peptide-major histocompatibiliteitscomplex MHC (pMHC) -complex dat tot expressie komt op het oppervlak van doelcellen. Omdat TCR-pMHC-herkenning plaatsvindt op de kruising tussen twee mobiele cellen, wordt al lang vermoed dat het mechanische krachten ervaart. Bovendien leidde dit tot het mechanosensormodel van TCR-activering, dat suggereert dat TCR-krachten bijdragen aan zijn functie 3,4. Om te begrijpen wanneer, waar en hoe mechanische krachten bijdragen aan de T-celfunctie, is het noodzakelijk om hulpmiddelen te ontwikkelen om de moleculaire krachten die door T-cellen worden overgedragen te visualiseren. Traditioneel worden methoden zoals tractiekrachtmicroscopie (TFM) en micropillar arrays gebruikt om cellulaire krachten 5,6 te onderzoeken. De krachtgevoeligheid van TFM en micropilaire arrays bevindt zich echter op de nanonewton (nN) schaal en is dus vaak onvoldoende om de moleculaire piconewton (pN) krachten te bestuderen die door celreceptorenworden overgedragen 7. Om de kracht en ruimtelijke resolutie voor detectie te verbeteren, pionierde ons laboratorium met de ontwikkeling van moleculaire spanningsondes, die aanvankelijk werden gesynthetiseerd met behulp van polyethyleenglycol (PEG) polymeren7. Moleculaire spanningsondes bestaan uit een uitschuifbare moleculaire “veer” (PEG, eiwit, DNA) geflankeerd door een fluorofoor en quencher en zijn verankerd op een oppervlak. Krachten die op het eindpunt van de sonde worden uitgeoefend, leiden tot de uitbreiding ervan, waardoor de fluorofoor en de quencher worden gescheiden en zo een sterk fluorescentiesignaal wordt gegenereerd (figuur 1A)8,9,10.
In het afgelopen decennium hebben we een bibliotheek ontwikkeld van verschillende klassen van moleculaire spanningsondes met veerelementen gemaakt van nucleïnezuren11, eiwitten10 en polymeren8. Onder deze bieden de op DNA gebaseerde spanningssondes de hoogste signaal-ruisverhouding en de grootste krachtgevoeligheid, die gemakkelijk kan worden afgestemd van een paar pN tot ~ 20 pN11. We hebben deze real-time DNA-spanningsondes gebruikt om de moleculaire krachten te bestuderen die worden gegenereerd door vele verschillende celtypen, waaronder fibroblasten, kankercellen, bloedplaatjes en immuuncellen11,12,13. Dit manuscript zal protocollen beschrijven voor het synthetiseren en assembleren van DNA-spanningsondes op een oppervlak om moleculaire receptorkrachten in kaart te brengen met pN-krachtresolutie met behulp van een conventionele fluorescentiemicroscoop. Hoewel de huidige procedure chemische modificaties aan het nucleïnezuur omvat om de fluorescerende reporter te introduceren (figuur 1B), is het belangrijk op te merken dat veel van de modificatie- en zuiveringsstappen kunnen worden uitbesteed aan aangepaste DNA-synthesebedrijven. Daarom is de technologie van DNA-spanningsondes gemakkelijk en toegankelijk voor de bredere celbiologie en mechanobiologiegemeenschappen.
Kortom, om DNA-spanningssensoren samen te stellen, wordt een DNA-haarspeld gehybridiseerd tot een fluorescerende ligandstreng op de ene arm en een quencher-ankerstreng op de andere arm en vervolgens geïmmobiliseerd op een glazen substraat (figuur 1C, real-time spanning). Bij afwezigheid van mechanische kracht wordt de haarspeld gesloten en wordt dus de fluorescentie geblust. Wanneer de uitgeoefende mechanische kracht echter groter is dan de F1/2 (de kracht bij evenwicht die leidt tot een kans van 50% om zich te ontvouwen), smelt de haarspeld mechanisch en wordt een fluorescerend signaal gegenereerd.
Voortbouwend op de real-time DNA-spanningssensor beschrijven we ook protocollen om geaccumuleerde krachten in kaart te brengen, wat vooral nuttig is voor het bestuderen van interacties tussen receptoren op immuuncellen en hun natuurlijke ligand. Dit komt omdat immuunreceptoren vaak kortstondige bindingen vertonen 3,14. Geaccumuleerde krachten worden in beeld gebracht met behulp van een “vergrendelingsstreng” die bij voorkeur bindt aan open DNA-haarspelden en de opslag van fluorescentiesignalen mogelijk maakt die verband houden met mechanische trekgebeurtenissen (figuur 1C, vergrendelde spanning). De vergrendelingsstreng is ontworpen om een cryptische bindingsplaats te binden die wordt blootgesteld aan mechanisch geïnduceerd smelten van de haarspeld en de haarspeld in de open toestand te vergrendelen door het opnieuw vouwen van de haarspeld te blokkeren, waardoor het spanningssignaal wordt opgeslagen en een geaccumuleerde spanningskaart wordt gegenereerd. Bovendien is de vergrendelingsstreng ontworpen met een acht-nucleotide teengreep, die een teengreep-gemedieerde strengverplaatsingsreactie mogelijk maakt met zijn volledige complement, de “ontgrendelende” streng. Met de toevoeging van de ontgrendelingsstreng wordt de gebonden vergrendelingsstreng van de haarspeldconstructie gestript, waardoor het opgeslagen spanningssignaal wordt gewist en de haarspeld wordt teruggezet naar de real-time status.
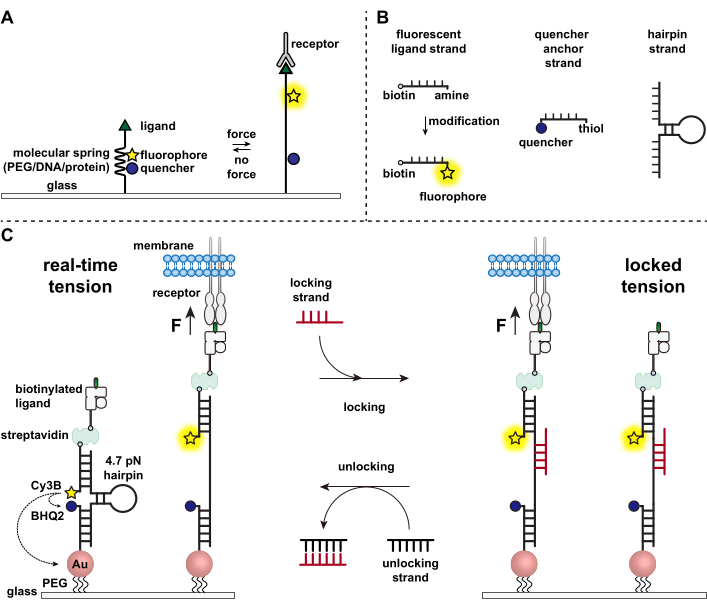
Figuur 1: Schema van de state-of-art moleculaire spanningsondes . (A) Algemeen ontwerp van real-time moleculaire spanningsonde, (B) Strengen voor de DNA-gebaseerde spanningsondeconstructie, en (C) gemanipuleerde DNA-gebaseerde spanningsondes en hun schakelen tussen real-time toestand en vergrendelde toestand. Klik hier om een grotere versie van deze figuur te bekijken.
Het hoofdprotocol bestaat uit vier hoofdsecties – oligonucleotidepreparaat, oppervlaktevoorbereiding, beeldvorming en gegevensanalyse. Dit protocol is met succes aangetoond door ons laboratorium en anderen in naïeve en geactiveerde OT-1 CD8 + T-cellen, OT-II CD4 + -cellen, evenals hybridomen, en kan worden toegepast om verschillende immuuncelreceptoren te ondervragen, waaronder T-celreceptor, geprogrammeerde celdoodreceptor (PD1) en lymfocytfunctie-geassocieerde antigeen 1 (LFA-1) krachten. OT-1 CD8+ naïeve T-cellen worden in dit artikel als voorbeeldcellijn gebruikt.
Protocol
Representative Results
Discussion
Met de gedetailleerde procedures die hier worden gegeven, kan men DNA-haarspeldspanningsondesubstraten voorbereiden om de receptorspanning geproduceerd door immuuncellen in kaart te brengen en te kwantificeren. Wanneer cellen op het substraat van de DNA-haarspeldspanningsonde worden geplaatst, landen, hechten en verspreiden ze zich terwijl de receptoren de liganden zowel chemisch als mechanisch waarnemen, waarvan de laatste wordt gedetecteerd door onze sondes. In sommige gevallen kunnen cellen zich echter niet verspreide…
Declarações
The authors have nothing to disclose.
Acknowledgements
Dit werk werd ondersteund door NIH Grants R01GM131099, NIH R01GM124472 en NSF CAREER 1350829. We danken de NIH Tetramer Facility voor pMHC-liganden. Deze studie werd gedeeltelijk ondersteund door de Emory Comprehensive Glycomics Core.
Materials
| 3-hydroxypicolinic acid (3-HPA) | Sigma | 56197 | maldi-TOF-MS matrix |
| mPEG-SC | Biochempeg | MF001023-2K | surface prep |
| (3-Aminopropyl)triethoxysilane | Acros | AC430941000 | surface prep |
| 10x Red blood cell lysis buffer | Biolegend | 00-4333-57 | buffer |
| 8.8 nm gold nanoparticles, tannic acid | Nanocomposix | customized order | surface prep |
| Atto647N NHS ester | Sigma | 18373-1MG-F | fluorophore, oligo prep |
| Attofluor Cell Chamber, for microscopy | Thermo Fisher Scientific | A7816 | imaging |
| BD Syringes only with Luer-Lok | BD bioscience | 309657 | cells |
| biotinylated anti-mouse CD3e | ebioscience | 13-0031-82 | antibody/ligand |
| Biotinylated pMHC ovalbumin (SIINFEKL) | NIH Tetramer Core Facility at Emory University | NA | antibody/ligand |
| bovine serum albumin | Sigma | 735078001 | block non-specific interactions |
| Cell strainers | Biologix | 15-1100 | cells |
| Coverslip Mini-Rack, teflon | Thermo Fisher Scientific | C14784 | surface prep |
| Cy3B NHS ester | GE Healthcare | PA63101 | fluorophore, oligo prep |
| Dulbecco's phosphate-buffered saline (DPBS) | Corning | 21-031-CM | buffer |
| ethanol | Sigma | 459836 | surface prep |
| Hank’s balanced salts (HBSS) | Sigma | H8264 | buffer |
| hydrogen peroxide | Sigma | H1009 | surface prep |
| LA-PEG-SC | Biochempeg | HE039023-3.4K | surface prep |
| Midi MACS (LS) startup kit | Miltenyi Biotec | 130-042-301 | cells |
| mouse CD8+ T cell isolation kit | Miltenyi Biotec | 130-104-075 | cells |
| Nanosep MF centrifugal devices | Pall laboratory | ODM02C35 | oligo prep |
| No. 2 round glass coverslips | VWR | 48382-085 | surface prep |
| NTA-SAM | Dojindo Molecular Technologies | N475-10 | surface prep |
| P2 gel | Bio-rad | 1504118 | oligo prep |
| sufuric acid | EMD Millipore Corporation | SX1244-6 | surface prep |
| Sulfo-NHS acetate | Thermo Fisher Scientific | 26777 | surface prep |
| Equipment | |||
| Agilent AdvanceBio Oligonucleotide C18 column, 4.6 x 150 mm, 2.7 μm | 653950-702 | oligonucleotide preparation | |
| Barnstead Nanopure water purifying system | Thermo Fisher | water | |
| CFI Apo 100× NA 1.49 objective | Nikon | Microscopy | |
| Cy5 cube | CHROMA | Microscopy | |
| evolve electron multiplying charge coupled device (EMCCD) | Photometrics | Microscopy | |
| High-performance liquid chromatography | Agilent 1100 | oligonucleotide preparation | |
| Intensilight epifluorescence source | Nikon | Microscopy | |
| Matrix-assisted laser desorption/ionization time-of-flight mass spectrometer (MALDI-TOF-MS) | Voyager STR | oligonucleotide preparation | |
| Nanodrop 2000 UV-Vis Spectrophotometer | Thermo Fisher | oligonucleotide preparation | |
| Nikon Eclipse Ti inverted microscope | Nikon | Microscopy | |
| Nikon Perfect Focus System | Nikon | Microscopy | |
| NIS Elements software | Nikon | Microscopy | |
| quad band TIRF 405/488/561/647 cube | CHROMA | Microscopy | |
| RICM cube | CHROMA | Microscopy | |
| TIRF launcher with 488 nm (50 mW), 561 nm (50 mW), and 640 nm | Coherent | Microscopy | |
| TRITC cube | CHROMA | Microscopy | |
| oligo name | 5' modification / 3' modification | sequence (5' to 3') | Use |
| 15mer amine locking strand | 5' modification: no modification 3' modification: /3AmMO/ |
AAA AAA CAT TTA TAC CCT ACC TA | locking real-time tension signal |
| 15mer Atto647N locking strand | 5' modification: Atto647N 3' modification: /3AmMO/ |
AAA AAA CAT TTA TAC CCT ACC TA | locking real-time tension signal |
| 15mer non-fluoresccent locking strand | 5' modification: no modification 3' modification: no modification |
A AAA AAC ATT TAT AC | locking real-time tension signal for quantitative analysis |
| 4.7 pN hairpin strand | 5' modification: no modification 3' modification: no modification |
GTGAAATACCGCACAGATGCGT TTGTATAAATGTTTTTTTCATTTAT ACTTTAAGAGCGCCACGTAGCC CAGC |
hairpin probe |
| amine ligand strand | 5' modification: /5AmMC6/ 3' modification: /3Bio/ |
CGCATCTGTGCG GTA TTT CAC TTT | hairpin probe |
| BHQ2 anchor strand | 5' modification: /5ThiolMC6-D/ 3' modification: /3BHQ_2/ |
TTTGCTGGGCTACGTGGCGCTCTT | hairpin probe |
| Cy3B ligand strand | 5' modification: Cy3B 3' modification: /3Bio/ |
CGCATCTGTGCG GTA TTT CAC TTT | hairpin probe |
| unlocking strand | 5' modification: no modification 3' modification: no modification |
TAG GTA GGG TAT AAA TGT TTT TTT C | unlocking accumulated tension signal |
Referências
- Dustin, M. L. T-cell activation through immunological synapses and kinapses. Immunological Reviews. 221 (1), 77-89 (2008).
- Spillane, K. M., Tolar, P. B cell antigen extraction is regulated by physical properties of antigen-presenting cells. Journal of Cell Biology. 216 (1), 217-230 (2017).
- Feng, Y., et al. Mechanosensing drives acuity of αβ T-cell recognition. Proceedings of the National Academy of Sciences. 114 (39), 8204-8213 (2017).
- Hong, J., et al. A TCR mechanotransduction signaling loop induces negative selection in the thymus. Nature Immunology. 19 (12), 1379-1390 (2018).
- Basu, R., et al. Cytotoxic T cells use mechanical force to potentiate target cell killing. Cell. 165 (1), 100-110 (2016).
- Bashour, K. T., et al. CD28 and CD3 have complementary roles in T-cell traction forces. Proceedings of the National Academy of Sciences. 111 (6), 2241-2246 (2014).
- Ma, V. P. Y., Salaita, K. DNA nanotechnology as an emerging tool to study mechanotransduction in living systems. Small. 15 (26), 1900961 (2019).
- Liu, Y., Yehl, K., Narui, Y., Salaita, K. Tension sensing nanoparticles for mechano-imaging at the living/nonliving interface. Journal of the American Chemical Society. 135 (14), 5320-5323 (2013).
- Glazier, R., et al. DNA mechanotechnology reveals that integrin receptors apply pN forces in podosomes on fluid substrates. Nature Communications. 10 (1), 1-13 (2019).
- Galior, K., Liu, Y., Yehl, K., Vivek, S., Salaita, K. Titin-based nanoparticle tension sensors map high-magnitude integrin forces within focal adhesions. Nano Letters. 16 (1), 341-348 (2016).
- Zhang, Y., Ge, C., Zhu, C., Salaita, K. DNA-based digital tension probes reveal integrin forces during early cell adhesion. Nature Communications. 5, 5167 (2014).
- Liu, Y., et al. DNA-based nanoparticle tension sensors reveal that T-cell receptors transmit defined pN forces to their antigens for enhanced fidelity. Proceedings of the National Academy of Sciences. 113 (20), 5610-5615 (2016).
- Zhang, Y., et al. Platelet integrins exhibit anisotropic mechanosensing and harness piconewton forces to mediate platelet aggregation. Proceedings of the National Academy of Sciences. 115 (2), 325-330 (2018).
- Huang, J., et al. The kinetics of two-dimensional TCR and pMHC interactions determine T-cell responsiveness. Nature. 464 (7290), 932-936 (2010).
- Ma, R., et al. DNA probes that store mechanical information reveal transient piconewton forces applied by T cells. Proceedings of the National Academy of Sciences. 116 (34), 16949-16954 (2019).
- Hui, E., et al. T cell costimulatory receptor CD28 is a primary target for PD-1-mediated inhibition. Science. 355 (6332), 1428-1433 (2017).
- Whitley, K. D., Comstock, M. J., Chemla, Y. R. Elasticity of the transition state for oligonucleotide hybridization. Nucleic Acids Research. 45 (2), 547-555 (2016).
- Brockman, J. M., et al. Live-cell super-resolved PAINT imaging of piconewton cellular traction forces. Nature Methods. 17 (10), 1018-1024 (2020).

