Bağışıklık Hücreleri Tarafından Geçici Piconewton Reseptör Kuvvetlerini Haritalamak için DNA Gerilim Probları
Summary
Bu makalede, bağışıklık hücreleri tarafından uygulanan reseptör kuvvetlerini görüntülemek için DNA tabanlı gerilim problarını kullanmak için ayrıntılı bir protokol açıklanmaktadır. Bu yaklaşım, reseptör kuvvetlerini gerçek zamanlı olarak >4.7pN’yi haritalayabilir ve zaman içinde kuvvetleri entegre edebilir.
Abstract
İki komşu hücre arasındaki kavşakta ve hücreler ile hücre dışı matris arasındaki kavşakta iletilen mekanik kuvvetler, gelişimden immünolojiye kadar birçok süreci düzenlemek için kritik öneme sahiptir. Bu nedenle, bu kuvvetleri moleküler ölçekte incelemek için araçlar geliştirmek kritik öneme sahiptir. Grubumuz, hücreler tarafından üretilen ve belirli ligandlara iletilen kuvvetleri ölçmek ve görselleştirmek için bir dizi moleküler gerilim sensörü geliştirdi. Moleküler gerilim sensörlerinin en hassas sınıfı, nükleik asit sap-döngü saç tokalarından oluşur. Bu sensörler, DNA saç tokalarının kuvvet altında mekanik olarak uzamasını ve açılmasını bildirmek için florofor-söndürücü çiftleri kullanır. DNA saç tokası gerginlik sensörleri ile ilgili bir zorluk, gerginliğin sona ermesi üzerine hızlı saç tokası ile geri dönüşümlü olmaları ve bu nedenle geçici kuvvetlerin kaydedilmesinin zor olmasıdır. Bu makalede, mekanik bilginin “depolanmasını” sağlamak için “kilitlenebilen” ve yeniden katlanması önlenebilen DNA gerilim sensörlerini hazırlama protokollerini açıklayacağız. Bu, kilidi kaldıran tamamlayıcı nükleik asitlerin eklenmesiyle daha sonra “silinebilen” oldukça geçici pikonewton kuvvetlerinin kaydedilmesine izin verir. Gerçek zamanlı gerilim haritalaması ve mekanik bilgi depolama arasında geçiş yapma yeteneği, T hücreleri tarafından bağışıklık fonksiyonlarının bir parçası olarak yaygın olarak kullanılan zayıf, kısa ömürlü ve daha az bol miktarda kuvvet ortaya çıkarır.
Introduction
Bağışıklık hücreleri, hedef hücrelerin yüzeylerini antijenler için sürekli tarayarak ve tarayarak, yüzeylerini 1,2 saplayarak patojenlere ve kanser hücrelerine karşı savunurlar. Antijen tanıma, hedef hücrelerin yüzeyinde eksprese edilen T hücre reseptörü (TCR) ile peptid-majör histokompatibilite kompleksi MHC (pMHC) kompleksi arasındaki bağlanma ile başlatılır. TCR-pMHC tanıma iki mobil hücre arasındaki kavşakta meydana geldiğinden, uzun zamandır mekanik kuvvetler yaşadığından şüphelenilmektedir. Dahası, bu, TCR kuvvetlerinin 3,4 işlevine katkıda bulunduğunu öne süren TCR aktivasyonunun mekanosensör modeline yol açtı. Mekanik kuvvetlerin T hücresi fonksiyonuna ne zaman, nerede ve nasıl katkıda bulunduğunu anlamak için, T hücreleri tarafından iletilen moleküler kuvvetleri görselleştirmek için araçlar geliştirmek zorunludur. Geleneksel olarak, çekiş kuvveti mikroskobu (TFM) ve mikrosütun dizileri gibi yöntemler, hücresel kuvvetleri araştırmak için kullanılır 5,6. Bununla birlikte, TFM ve mikrosütun dizilerinin kuvvet duyarlılığı nanonewton (nN) ölçeğindedir ve bu nedenle hücre reseptörleri tarafından iletilen moleküler pikonewton (pN) kuvvetlerini incelemek için genellikle yetersizdir7. Tespit için kuvveti ve uzamsal çözünürlüğü iyileştirmek için laboratuvarımız başlangıçta polietilen glikol (PEG) polimerleri7 kullanılarak sentezlenen moleküler gerilim problarının geliştirilmesine öncülük etti. Moleküler gerilim probları, bir florofor ve söndürücü ile çevrili uzatılabilir bir moleküler “yay” dan (PEG, protein, DNA) oluşur ve bir yüzeye sabitlenir. Probun ucuna uygulanan kuvvetler, florofor ve söndürücüyü ayırarak genişlemesine yol açar ve böylece güçlü bir floresan sinyali üretir (Şekil 1A)8,9,10.
Son on yılda, nükleik asitler11, proteinler10 vepolimerler 8’den yapılmış yay elementleri ile farklı moleküler gerilim probları sınıflarından oluşan bir kütüphane geliştirdik. Bunlar arasında, DNA tabanlı gerilim probları, birkaç pN’den ~ 20 pN11’e kadar kolayca ayarlanabilen en yüksek sinyal-gürültü oranını ve en yüksek kuvvet hassasiyetini sağlar. Bu gerçek zamanlı DNA gerilim problarını, fibroblastlar, kanser hücreleri, trombositler ve bağışıklık hücreleri11,12,13 dahil olmak üzere birçok farklı hücre tipi tarafından üretilen moleküler kuvvetleri incelemek için kullandık. Bu makalede, geleneksel floresan mikroskobu kullanılarak moleküler reseptör kuvvetlerini pN kuvvet çözünürlüğü ile haritalamak için DNA gerilim problarını bir yüzey üzerinde sentezlemek ve birleştirmek için kullanılan protokoller anlatılacaktır. Mevcut prosedür, floresan muhabiri tanıtmak için nükleik asitte kimyasal modifikasyonlar içerirken (Şekil 1B), modifikasyon ve saflaştırma adımlarının çoğunun özel DNA sentez şirketlerine dış kaynaklı olabileceğini belirtmek önemlidir. Bu nedenle, DNA gerilim probları teknolojisi kolaydır ve daha geniş hücre biyolojisi ve mekanobiyoloji toplulukları için erişilebilirdir.
Kısaca, DNA gerilim sensörlerini monte etmek için, bir DNA saç tokası bir koldaki floresan ligand ipliğine ve diğer koldaki bir söndürücü çapa ipliğine hibridize edilir ve daha sonra bir cam substrat üzerinde hareketsiz hale getirilir (Şekil 1C, gerçek zamanlı gerginlik). Mekanik kuvvetin yokluğunda, saç tokası kapatılır ve böylece floresan söndürülür. Bununla birlikte, uygulanan mekanik kuvvet F1/2’den (% 50’lik bir açılma olasılığına yol açan dengedeki kuvvet) daha büyük olduğunda, saç tokası mekanik olarak erir ve bir floresan sinyali üretilir.
Gerçek zamanlı DNA gerilim sensörüne dayanarak, birikmiş kuvvetleri haritalamak için protokolleri de açıklıyoruz; bu, bağışıklık hücreleri üzerindeki reseptörler ile doğal ligandları arasındaki etkileşimleri incelemek için özellikle yararlıdır. Bunun nedeni, bağışıklık reseptörlerinin sıklıkla kısa ömürlü bağlar göstermesidir 3,14. Biriken kuvvetler, tercihen açık DNA saç tokalarına bağlanan ve mekanik çekme olaylarıyla ilişkili floresan sinyallerinin depolanmasına izin veren bir “kilitleme” ipliği kullanılarak görüntülenir (Şekil 1C, kilitli gerilim). Kilitleme ipliği, saç tokasının mekanik olarak indüklenen erimesi üzerine açığa çıkan şifreli bir bağlama bölgesini bağlamak ve saç tokasının yeniden katlanmasını engelleyerek saç tokasını açık durumda kilitlemek, böylece gerginlik sinyalini depolamak ve birikmiş bir gerilim haritası oluşturmak için tasarlanmıştır. Dahası, kilitleme ipliği, tam tamamlayıcısı olan “kilit açma” ipliği ile ayak parmağı aracılı bir iplik yer değiştirme reaksiyonu sağlayan sekiz nükleotid ayak parmağı ile tasarlanmıştır. Kilit açma telinin eklenmesiyle, bağlı kilitleme teli saç tokası yapısından sıyrılır, depolanan gerginlik sinyali silinir ve saç tokasını gerçek zamanlı duruma geri döndürür.
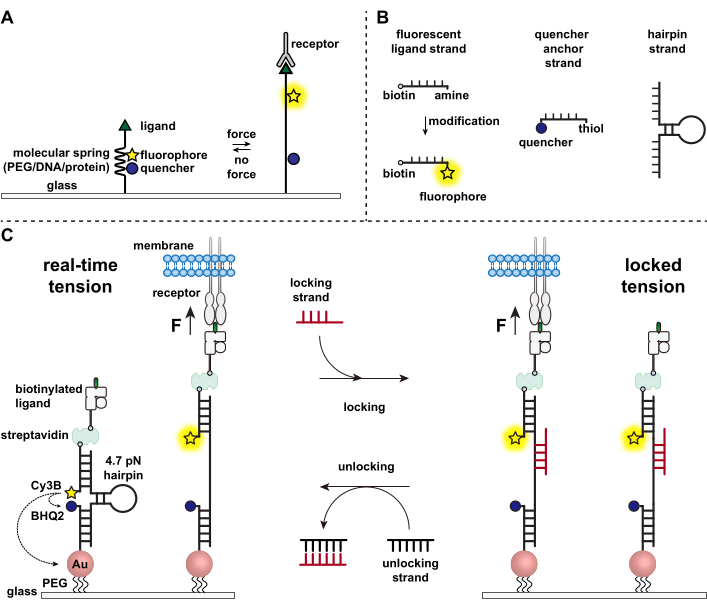
Resim 1: Son teknoloji moleküler gerilim problarının şeması . (A) Gerçek zamanlı moleküler gerilim probunun genel tasarımı, (B) DNA tabanlı gerilim probu yapısı için iplikçikler ve (C) DNA tabanlı gerilim probları ve bunların gerçek zamanlı durum ile kilitli durum arasında geçiş yapması. Bu şeklin daha büyük bir versiyonunu görmek için lütfen buraya tıklayın.
Ana protokol dört ana bölümden oluşur – oligonükleotid hazırlama, yüzey hazırlama, görüntüleme ve veri analizi. Bu protokol, laboratuvarımız ve diğerleri tarafından naif ve aktive edilmiş OT-1 CD8 + T hücreleri, OT-II CD4 + hücreleri ve hibridomlarda başarıyla gösterilmiştir ve T hücre reseptörü, programlanmış hücre ölüm reseptörü (PD1) ve lenfosit fonksiyonu ile ilişkili antijen 1 (LFA-1) kuvvetleri dahil olmak üzere farklı bağışıklık hücresi reseptörlerini sorgulamak için uygulanabilir. OT-1 CD8+ naif T hücreleri bu yazıda örnek hücre çizgisi olarak kullanılmıştır.
Protocol
Representative Results
Discussion
Burada verilen ayrıntılı prosedürlerle, bağışıklık hücreleri tarafından üretilen reseptör gerginliğini haritalamak ve ölçmek için DNA saç tokası gerginlik probu substratları hazırlanabilir. Hücreler DNA saç tokası gerilim probu substratına kaplandığında, reseptörler ligandları hem kimyasal hem de mekanik olarak algıladıkça iner, bağlanır ve yayılırlar, ikincisi problarımız tarafından tespit edilir. Bununla birlikte, bazı durumlarda hücreler yayılmayabilir (?…
Declarações
The authors have nothing to disclose.
Acknowledgements
Bu çalışma NIH Grants R01GM131099, NIH R01GM124472 ve NSF CAREER 1350829 tarafından desteklenmiştir. pMHC ligandları için NIH Tetramer Tesisi’ne teşekkür ederiz. Bu çalışma, kısmen, Emory Comprehensive Glycomics Core tarafından desteklenmiştir.
Materials
| 3-hydroxypicolinic acid (3-HPA) | Sigma | 56197 | maldi-TOF-MS matrix |
| mPEG-SC | Biochempeg | MF001023-2K | surface prep |
| (3-Aminopropyl)triethoxysilane | Acros | AC430941000 | surface prep |
| 10x Red blood cell lysis buffer | Biolegend | 00-4333-57 | buffer |
| 8.8 nm gold nanoparticles, tannic acid | Nanocomposix | customized order | surface prep |
| Atto647N NHS ester | Sigma | 18373-1MG-F | fluorophore, oligo prep |
| Attofluor Cell Chamber, for microscopy | Thermo Fisher Scientific | A7816 | imaging |
| BD Syringes only with Luer-Lok | BD bioscience | 309657 | cells |
| biotinylated anti-mouse CD3e | ebioscience | 13-0031-82 | antibody/ligand |
| Biotinylated pMHC ovalbumin (SIINFEKL) | NIH Tetramer Core Facility at Emory University | NA | antibody/ligand |
| bovine serum albumin | Sigma | 735078001 | block non-specific interactions |
| Cell strainers | Biologix | 15-1100 | cells |
| Coverslip Mini-Rack, teflon | Thermo Fisher Scientific | C14784 | surface prep |
| Cy3B NHS ester | GE Healthcare | PA63101 | fluorophore, oligo prep |
| Dulbecco's phosphate-buffered saline (DPBS) | Corning | 21-031-CM | buffer |
| ethanol | Sigma | 459836 | surface prep |
| Hank’s balanced salts (HBSS) | Sigma | H8264 | buffer |
| hydrogen peroxide | Sigma | H1009 | surface prep |
| LA-PEG-SC | Biochempeg | HE039023-3.4K | surface prep |
| Midi MACS (LS) startup kit | Miltenyi Biotec | 130-042-301 | cells |
| mouse CD8+ T cell isolation kit | Miltenyi Biotec | 130-104-075 | cells |
| Nanosep MF centrifugal devices | Pall laboratory | ODM02C35 | oligo prep |
| No. 2 round glass coverslips | VWR | 48382-085 | surface prep |
| NTA-SAM | Dojindo Molecular Technologies | N475-10 | surface prep |
| P2 gel | Bio-rad | 1504118 | oligo prep |
| sufuric acid | EMD Millipore Corporation | SX1244-6 | surface prep |
| Sulfo-NHS acetate | Thermo Fisher Scientific | 26777 | surface prep |
| Equipment | |||
| Agilent AdvanceBio Oligonucleotide C18 column, 4.6 x 150 mm, 2.7 μm | 653950-702 | oligonucleotide preparation | |
| Barnstead Nanopure water purifying system | Thermo Fisher | water | |
| CFI Apo 100× NA 1.49 objective | Nikon | Microscopy | |
| Cy5 cube | CHROMA | Microscopy | |
| evolve electron multiplying charge coupled device (EMCCD) | Photometrics | Microscopy | |
| High-performance liquid chromatography | Agilent 1100 | oligonucleotide preparation | |
| Intensilight epifluorescence source | Nikon | Microscopy | |
| Matrix-assisted laser desorption/ionization time-of-flight mass spectrometer (MALDI-TOF-MS) | Voyager STR | oligonucleotide preparation | |
| Nanodrop 2000 UV-Vis Spectrophotometer | Thermo Fisher | oligonucleotide preparation | |
| Nikon Eclipse Ti inverted microscope | Nikon | Microscopy | |
| Nikon Perfect Focus System | Nikon | Microscopy | |
| NIS Elements software | Nikon | Microscopy | |
| quad band TIRF 405/488/561/647 cube | CHROMA | Microscopy | |
| RICM cube | CHROMA | Microscopy | |
| TIRF launcher with 488 nm (50 mW), 561 nm (50 mW), and 640 nm | Coherent | Microscopy | |
| TRITC cube | CHROMA | Microscopy | |
| oligo name | 5' modification / 3' modification | sequence (5' to 3') | Use |
| 15mer amine locking strand | 5' modification: no modification 3' modification: /3AmMO/ |
AAA AAA CAT TTA TAC CCT ACC TA | locking real-time tension signal |
| 15mer Atto647N locking strand | 5' modification: Atto647N 3' modification: /3AmMO/ |
AAA AAA CAT TTA TAC CCT ACC TA | locking real-time tension signal |
| 15mer non-fluoresccent locking strand | 5' modification: no modification 3' modification: no modification |
A AAA AAC ATT TAT AC | locking real-time tension signal for quantitative analysis |
| 4.7 pN hairpin strand | 5' modification: no modification 3' modification: no modification |
GTGAAATACCGCACAGATGCGT TTGTATAAATGTTTTTTTCATTTAT ACTTTAAGAGCGCCACGTAGCC CAGC |
hairpin probe |
| amine ligand strand | 5' modification: /5AmMC6/ 3' modification: /3Bio/ |
CGCATCTGTGCG GTA TTT CAC TTT | hairpin probe |
| BHQ2 anchor strand | 5' modification: /5ThiolMC6-D/ 3' modification: /3BHQ_2/ |
TTTGCTGGGCTACGTGGCGCTCTT | hairpin probe |
| Cy3B ligand strand | 5' modification: Cy3B 3' modification: /3Bio/ |
CGCATCTGTGCG GTA TTT CAC TTT | hairpin probe |
| unlocking strand | 5' modification: no modification 3' modification: no modification |
TAG GTA GGG TAT AAA TGT TTT TTT C | unlocking accumulated tension signal |
Referências
- Dustin, M. L. T-cell activation through immunological synapses and kinapses. Immunological Reviews. 221 (1), 77-89 (2008).
- Spillane, K. M., Tolar, P. B cell antigen extraction is regulated by physical properties of antigen-presenting cells. Journal of Cell Biology. 216 (1), 217-230 (2017).
- Feng, Y., et al. Mechanosensing drives acuity of αβ T-cell recognition. Proceedings of the National Academy of Sciences. 114 (39), 8204-8213 (2017).
- Hong, J., et al. A TCR mechanotransduction signaling loop induces negative selection in the thymus. Nature Immunology. 19 (12), 1379-1390 (2018).
- Basu, R., et al. Cytotoxic T cells use mechanical force to potentiate target cell killing. Cell. 165 (1), 100-110 (2016).
- Bashour, K. T., et al. CD28 and CD3 have complementary roles in T-cell traction forces. Proceedings of the National Academy of Sciences. 111 (6), 2241-2246 (2014).
- Ma, V. P. Y., Salaita, K. DNA nanotechnology as an emerging tool to study mechanotransduction in living systems. Small. 15 (26), 1900961 (2019).
- Liu, Y., Yehl, K., Narui, Y., Salaita, K. Tension sensing nanoparticles for mechano-imaging at the living/nonliving interface. Journal of the American Chemical Society. 135 (14), 5320-5323 (2013).
- Glazier, R., et al. DNA mechanotechnology reveals that integrin receptors apply pN forces in podosomes on fluid substrates. Nature Communications. 10 (1), 1-13 (2019).
- Galior, K., Liu, Y., Yehl, K., Vivek, S., Salaita, K. Titin-based nanoparticle tension sensors map high-magnitude integrin forces within focal adhesions. Nano Letters. 16 (1), 341-348 (2016).
- Zhang, Y., Ge, C., Zhu, C., Salaita, K. DNA-based digital tension probes reveal integrin forces during early cell adhesion. Nature Communications. 5, 5167 (2014).
- Liu, Y., et al. DNA-based nanoparticle tension sensors reveal that T-cell receptors transmit defined pN forces to their antigens for enhanced fidelity. Proceedings of the National Academy of Sciences. 113 (20), 5610-5615 (2016).
- Zhang, Y., et al. Platelet integrins exhibit anisotropic mechanosensing and harness piconewton forces to mediate platelet aggregation. Proceedings of the National Academy of Sciences. 115 (2), 325-330 (2018).
- Huang, J., et al. The kinetics of two-dimensional TCR and pMHC interactions determine T-cell responsiveness. Nature. 464 (7290), 932-936 (2010).
- Ma, R., et al. DNA probes that store mechanical information reveal transient piconewton forces applied by T cells. Proceedings of the National Academy of Sciences. 116 (34), 16949-16954 (2019).
- Hui, E., et al. T cell costimulatory receptor CD28 is a primary target for PD-1-mediated inhibition. Science. 355 (6332), 1428-1433 (2017).
- Whitley, K. D., Comstock, M. J., Chemla, Y. R. Elasticity of the transition state for oligonucleotide hybridization. Nucleic Acids Research. 45 (2), 547-555 (2016).
- Brockman, J. M., et al. Live-cell super-resolved PAINT imaging of piconewton cellular traction forces. Nature Methods. 17 (10), 1018-1024 (2020).

