Sonde di tensione del DNA per mappare le forze transitorie del recettore di Piconewton da parte delle cellule immunitarie
Summary
Questo documento descrive un protocollo dettagliato per l’utilizzo di sonde di tensione basate sul DNA per visualizzare le forze recettoriali applicate dalle cellule immunitarie. Questo approccio può mappare le forze recettoriali >4.7pN in tempo reale e può integrare le forze nel tempo.
Abstract
Le forze meccaniche trasmesse alla giunzione tra due cellule vicine e alla giunzione tra le cellule e la matrice extracellulare sono fondamentali per regolare molti processi che vanno dallo sviluppo all’immunologia. Pertanto, lo sviluppo di strumenti per studiare queste forze su scala molecolare è fondamentale. Il nostro gruppo ha sviluppato una suite di sensori di tensione molecolare per quantificare e visualizzare le forze generate dalle cellule e trasmesse a ligandi specifici. La classe più sensibile di sensori di tensione molecolare è costituita da forcine ad anello stelo di acido nucleico. Questi sensori utilizzano coppie fluoroforo-quencher per segnalare l’estensione meccanica e lo sviluppo delle forcine del DNA sotto forza. Una sfida con i sensori di tensione a forcina del DNA è che sono reversibili con un rapido ripiegamento della forcina al termine della tensione e quindi le forze transitorie sono difficili da registrare. In questo articolo, descriviamo i protocolli per la preparazione di sensori di tensione del DNA che possono essere “bloccati” e impediti di ripiegarsi per consentire la “memorizzazione” di informazioni meccaniche. Ciò consente la registrazione di forze piconewton altamente transitorie, che possono essere successivamente “cancellate” dall’aggiunta di acidi nucleici complementari che rimuovono la serratura. Questa capacità di alternare tra la mappatura della tensione in tempo reale e la memorizzazione di informazioni meccaniche rivela forze deboli, di breve durata e meno abbondanti, che sono comunemente impiegate dalle cellule T come parte delle loro funzioni immunitarie.
Introduction
Le cellule immunitarie difendono dagli agenti patogeni e dalle cellule tumorali strisciando e scansionando continuamente le superfici delle cellule bersaglio alla ricerca di antigeni, costellando la loro superficie 1,2. Il riconoscimento dell’antigene viene avviato al momento del legame tra il recettore delle cellule T (TCR) e il complesso di istocompatibilità peptide-maggiore MHC (pMHC) espresso sulla superficie delle cellule bersaglio. Poiché il riconoscimento TCR-pMHC avviene alla giunzione tra due cellule mobili, è stato a lungo sospettato di sperimentare forze meccaniche. Inoltre, questo ha portato al modello meccanosensore di attivazione TCR, che suggerisce che le forze TCR contribuiscono alla sua funzione 3,4. Per capire quando, dove e come le forze meccaniche contribuiscono alla funzione delle cellule T, è imperativo sviluppare strumenti per visualizzare le forze molecolari trasmesse dalle cellule T. Tradizionalmente, metodi come la microscopia della forza di trazione (TFM) e gli array di micropilastri sono usati per studiare le forze cellulari 5,6. Tuttavia, la sensibilità alla forza degli array di TFM e micropilastri è alla scala nanonewton (nN) e quindi è spesso insufficiente per studiare le forze molecolari di piconewton (pN) trasmesse dai recettori cellulari7. Per migliorare la forza e la risoluzione spaziale per il rilevamento, il nostro laboratorio ha aperto la strada allo sviluppo di sonde di tensione molecolare, che sono state inizialmente sintetizzate utilizzando polimeri di polietilenglicole (PEG)7. Le sonde di tensione molecolare sono costituite da una “molla” molecolare estensibile (PEG, proteina, DNA) affiancata da un fluoroforo e un quencher e sono ancorate su una superficie. Le forze applicate al terminale della sonda portano alla sua estensione, separando il fluoroforo e il quencher, e generando così un forte segnale di fluorescenza (Figura 1A)8,9,10.
Negli ultimi dieci anni abbiamo sviluppato una libreria di diverse classi di sonde di tensione molecolari con elementi a molla costituiti da acidi nucleici11, proteine10 e polimeri8. Tra questi, le sonde di tensione basate sul DNA forniscono il più alto rapporto segnale/rumore e la massima sensibilità alla forza, che è facilmente sintonizzata da pochi pN fino a ~20 pN11. Abbiamo utilizzato queste sonde di tensione del DNA in tempo reale per studiare le forze molecolari generate da molti diversi tipi di cellule, tra cui fibroblasti, cellule tumorali, piastrine e cellule immunitarie11,12,13. Questo manoscritto descriverà i protocolli per sintetizzare e assemblare sonde di tensione del DNA su una superficie per mappare le forze dei recettori molecolari con risoluzione della forza pN utilizzando un microscopio a fluorescenza convenzionale. Mentre l’attuale procedura include modifiche chimiche all’acido nucleico per introdurre il reporter fluorescente (Figura 1B), è importante notare che molte delle fasi di modifica e purificazione possono essere esternalizzate a società di sintesi del DNA personalizzate. Pertanto, la tecnologia delle sonde di tensione del DNA è facile e accessibile alle più ampie comunità di biologia cellulare e meccanobiologia.
In breve, per assemblare i sensori di tensione del DNA, una forcina di DNA viene ibridata con un filamento di ligando fluorescente su un braccio e un filamento di ancoraggio di quencher sull’altro braccio e quindi immobilizzato su un substrato di vetro (Figura 1C, tensione in tempo reale). In assenza di forza meccanica, la forcina viene chiusa e quindi la fluorescenza viene spenta. Tuttavia, quando la forza meccanica applicata è maggiore di F1/2 (la forza all’equilibrio che porta a una probabilità del 50% di dispiegarsi), la forcina si scioglie meccanicamente e viene generato un segnale fluorescente.
Basandoci sul sensore di tensione del DNA in tempo reale, descriviamo anche i protocolli per mappare le forze accumulate, che è particolarmente utile per studiare le interazioni tra i recettori sulle cellule immunitarie e il loro ligando naturale. Questo perché i recettori immunitari spesso mostrano legami di breve durata 3,14. Le forze accumulate vengono visualizzate utilizzando un filamento di “bloccaggio” che si lega preferenzialmente alle forcine di DNA aperte e consente la memorizzazione di segnali di fluorescenza associati a eventi di trazione meccanica (Figura 1C, tensione bloccata). Il filo di bloccaggio è progettato per legare un sito di legame criptico che viene esposto alla fusione indotta meccanicamente della forcina e bloccare la forcina nello stato aperto bloccando la ripiegatura della forcina, memorizzando così il segnale di tensione e generando una mappa di tensione accumulata. Inoltre, il filo di bloccaggio è progettato con un appiglio a otto nucleotidi, che consente una reazione di spostamento del filamento mediata dalla presa con il suo complemento completo, il filamento “sbloccante”. Con l’aggiunta del trefolo di sblocco, il filo di bloccaggio legato viene rimosso dal costrutto a forcina, cancellando il segnale di tensione memorizzato e ripristinando la forcina allo stato in tempo reale.
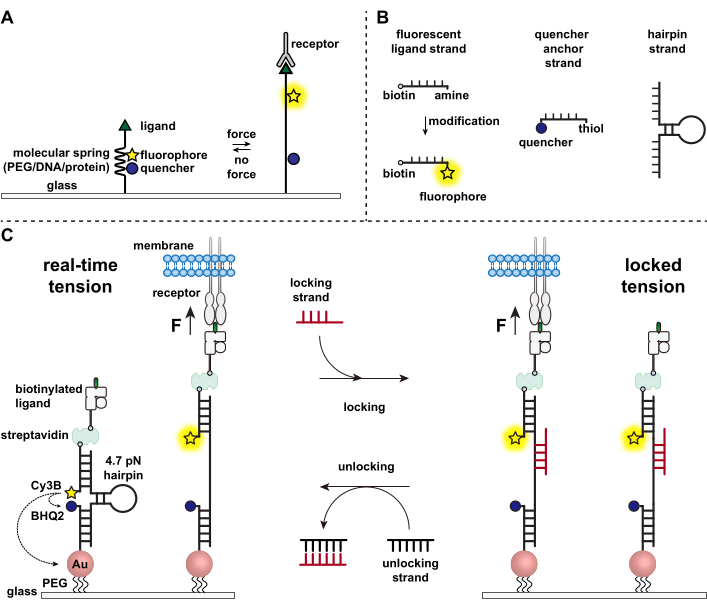
Figura 1: Schema delle sonde di tensione molecolari all’avanguardia . (A) Progettazione generale della sonda di tensione molecolare in tempo reale, (B) Filamenti per il costrutto della sonda di tensione basata sul DNA e (C) sonde di tensione basate sul DNA ingegnerizzate e loro commutazione tra stato in tempo reale e stato bloccato. Fare clic qui per visualizzare una versione ingrandita di questa figura.
Il protocollo principale consiste di quattro sezioni principali: preparazione degli oligonucleotidi, preparazione della superficie, imaging e analisi dei dati. Questo protocollo è stato dimostrato con successo dal nostro laboratorio e da altri in cellule T CD8+ OT-1 naïve e attivate, cellule CD4+ OT-II e ibridomi e può essere applicato per interrogare diversi recettori delle cellule immunitarie tra cui il recettore delle cellule T, il recettore della morte cellulare programmata (PD1) e le forze dell’antigene 1 associato alla funzione linfocitaria (LFA-1). Le cellule T CD8 + naïve OT-1 sono usate come linea cellulare di esempio in questo articolo.
Protocol
Representative Results
Discussion
Con le procedure dettagliate fornite qui, è possibile preparare substrati di sonda di tensione a forcina di DNA per mappare e quantificare la tensione del recettore prodotta dalle cellule immunitarie. Quando le cellule vengono placcate sul substrato della sonda di tensione della forcina del DNA, atterrano, si attaccano e si diffondono mentre i recettori percepiscono i ligandi sia chimicamente che meccanicamente, l’ultimo dei quali viene rilevato dalle nostre sonde. Tuttavia, in alcuni casi le cellule potrebbero non rius…
Declarações
The authors have nothing to disclose.
Acknowledgements
Questo lavoro è stato supportato da NIH Grants R01GM131099, NIH R01GM124472 e NSF CAREER 1350829. Ringraziamo il NIH Tetramer Facility per i ligandi pMHC. Questo studio è stato supportato, in parte, dall’Emory Comprehensive Glycomics Core.
Materials
| 3-hydroxypicolinic acid (3-HPA) | Sigma | 56197 | maldi-TOF-MS matrix |
| mPEG-SC | Biochempeg | MF001023-2K | surface prep |
| (3-Aminopropyl)triethoxysilane | Acros | AC430941000 | surface prep |
| 10x Red blood cell lysis buffer | Biolegend | 00-4333-57 | buffer |
| 8.8 nm gold nanoparticles, tannic acid | Nanocomposix | customized order | surface prep |
| Atto647N NHS ester | Sigma | 18373-1MG-F | fluorophore, oligo prep |
| Attofluor Cell Chamber, for microscopy | Thermo Fisher Scientific | A7816 | imaging |
| BD Syringes only with Luer-Lok | BD bioscience | 309657 | cells |
| biotinylated anti-mouse CD3e | ebioscience | 13-0031-82 | antibody/ligand |
| Biotinylated pMHC ovalbumin (SIINFEKL) | NIH Tetramer Core Facility at Emory University | NA | antibody/ligand |
| bovine serum albumin | Sigma | 735078001 | block non-specific interactions |
| Cell strainers | Biologix | 15-1100 | cells |
| Coverslip Mini-Rack, teflon | Thermo Fisher Scientific | C14784 | surface prep |
| Cy3B NHS ester | GE Healthcare | PA63101 | fluorophore, oligo prep |
| Dulbecco's phosphate-buffered saline (DPBS) | Corning | 21-031-CM | buffer |
| ethanol | Sigma | 459836 | surface prep |
| Hank’s balanced salts (HBSS) | Sigma | H8264 | buffer |
| hydrogen peroxide | Sigma | H1009 | surface prep |
| LA-PEG-SC | Biochempeg | HE039023-3.4K | surface prep |
| Midi MACS (LS) startup kit | Miltenyi Biotec | 130-042-301 | cells |
| mouse CD8+ T cell isolation kit | Miltenyi Biotec | 130-104-075 | cells |
| Nanosep MF centrifugal devices | Pall laboratory | ODM02C35 | oligo prep |
| No. 2 round glass coverslips | VWR | 48382-085 | surface prep |
| NTA-SAM | Dojindo Molecular Technologies | N475-10 | surface prep |
| P2 gel | Bio-rad | 1504118 | oligo prep |
| sufuric acid | EMD Millipore Corporation | SX1244-6 | surface prep |
| Sulfo-NHS acetate | Thermo Fisher Scientific | 26777 | surface prep |
| Equipment | |||
| Agilent AdvanceBio Oligonucleotide C18 column, 4.6 x 150 mm, 2.7 μm | 653950-702 | oligonucleotide preparation | |
| Barnstead Nanopure water purifying system | Thermo Fisher | water | |
| CFI Apo 100× NA 1.49 objective | Nikon | Microscopy | |
| Cy5 cube | CHROMA | Microscopy | |
| evolve electron multiplying charge coupled device (EMCCD) | Photometrics | Microscopy | |
| High-performance liquid chromatography | Agilent 1100 | oligonucleotide preparation | |
| Intensilight epifluorescence source | Nikon | Microscopy | |
| Matrix-assisted laser desorption/ionization time-of-flight mass spectrometer (MALDI-TOF-MS) | Voyager STR | oligonucleotide preparation | |
| Nanodrop 2000 UV-Vis Spectrophotometer | Thermo Fisher | oligonucleotide preparation | |
| Nikon Eclipse Ti inverted microscope | Nikon | Microscopy | |
| Nikon Perfect Focus System | Nikon | Microscopy | |
| NIS Elements software | Nikon | Microscopy | |
| quad band TIRF 405/488/561/647 cube | CHROMA | Microscopy | |
| RICM cube | CHROMA | Microscopy | |
| TIRF launcher with 488 nm (50 mW), 561 nm (50 mW), and 640 nm | Coherent | Microscopy | |
| TRITC cube | CHROMA | Microscopy | |
| oligo name | 5' modification / 3' modification | sequence (5' to 3') | Use |
| 15mer amine locking strand | 5' modification: no modification 3' modification: /3AmMO/ |
AAA AAA CAT TTA TAC CCT ACC TA | locking real-time tension signal |
| 15mer Atto647N locking strand | 5' modification: Atto647N 3' modification: /3AmMO/ |
AAA AAA CAT TTA TAC CCT ACC TA | locking real-time tension signal |
| 15mer non-fluoresccent locking strand | 5' modification: no modification 3' modification: no modification |
A AAA AAC ATT TAT AC | locking real-time tension signal for quantitative analysis |
| 4.7 pN hairpin strand | 5' modification: no modification 3' modification: no modification |
GTGAAATACCGCACAGATGCGT TTGTATAAATGTTTTTTTCATTTAT ACTTTAAGAGCGCCACGTAGCC CAGC |
hairpin probe |
| amine ligand strand | 5' modification: /5AmMC6/ 3' modification: /3Bio/ |
CGCATCTGTGCG GTA TTT CAC TTT | hairpin probe |
| BHQ2 anchor strand | 5' modification: /5ThiolMC6-D/ 3' modification: /3BHQ_2/ |
TTTGCTGGGCTACGTGGCGCTCTT | hairpin probe |
| Cy3B ligand strand | 5' modification: Cy3B 3' modification: /3Bio/ |
CGCATCTGTGCG GTA TTT CAC TTT | hairpin probe |
| unlocking strand | 5' modification: no modification 3' modification: no modification |
TAG GTA GGG TAT AAA TGT TTT TTT C | unlocking accumulated tension signal |
Referências
- Dustin, M. L. T-cell activation through immunological synapses and kinapses. Immunological Reviews. 221 (1), 77-89 (2008).
- Spillane, K. M., Tolar, P. B cell antigen extraction is regulated by physical properties of antigen-presenting cells. Journal of Cell Biology. 216 (1), 217-230 (2017).
- Feng, Y., et al. Mechanosensing drives acuity of αβ T-cell recognition. Proceedings of the National Academy of Sciences. 114 (39), 8204-8213 (2017).
- Hong, J., et al. A TCR mechanotransduction signaling loop induces negative selection in the thymus. Nature Immunology. 19 (12), 1379-1390 (2018).
- Basu, R., et al. Cytotoxic T cells use mechanical force to potentiate target cell killing. Cell. 165 (1), 100-110 (2016).
- Bashour, K. T., et al. CD28 and CD3 have complementary roles in T-cell traction forces. Proceedings of the National Academy of Sciences. 111 (6), 2241-2246 (2014).
- Ma, V. P. Y., Salaita, K. DNA nanotechnology as an emerging tool to study mechanotransduction in living systems. Small. 15 (26), 1900961 (2019).
- Liu, Y., Yehl, K., Narui, Y., Salaita, K. Tension sensing nanoparticles for mechano-imaging at the living/nonliving interface. Journal of the American Chemical Society. 135 (14), 5320-5323 (2013).
- Glazier, R., et al. DNA mechanotechnology reveals that integrin receptors apply pN forces in podosomes on fluid substrates. Nature Communications. 10 (1), 1-13 (2019).
- Galior, K., Liu, Y., Yehl, K., Vivek, S., Salaita, K. Titin-based nanoparticle tension sensors map high-magnitude integrin forces within focal adhesions. Nano Letters. 16 (1), 341-348 (2016).
- Zhang, Y., Ge, C., Zhu, C., Salaita, K. DNA-based digital tension probes reveal integrin forces during early cell adhesion. Nature Communications. 5, 5167 (2014).
- Liu, Y., et al. DNA-based nanoparticle tension sensors reveal that T-cell receptors transmit defined pN forces to their antigens for enhanced fidelity. Proceedings of the National Academy of Sciences. 113 (20), 5610-5615 (2016).
- Zhang, Y., et al. Platelet integrins exhibit anisotropic mechanosensing and harness piconewton forces to mediate platelet aggregation. Proceedings of the National Academy of Sciences. 115 (2), 325-330 (2018).
- Huang, J., et al. The kinetics of two-dimensional TCR and pMHC interactions determine T-cell responsiveness. Nature. 464 (7290), 932-936 (2010).
- Ma, R., et al. DNA probes that store mechanical information reveal transient piconewton forces applied by T cells. Proceedings of the National Academy of Sciences. 116 (34), 16949-16954 (2019).
- Hui, E., et al. T cell costimulatory receptor CD28 is a primary target for PD-1-mediated inhibition. Science. 355 (6332), 1428-1433 (2017).
- Whitley, K. D., Comstock, M. J., Chemla, Y. R. Elasticity of the transition state for oligonucleotide hybridization. Nucleic Acids Research. 45 (2), 547-555 (2016).
- Brockman, J. M., et al. Live-cell super-resolved PAINT imaging of piconewton cellular traction forces. Nature Methods. 17 (10), 1018-1024 (2020).

