Isolatie, karakterisering en totale DNA-extractie om endofytische schimmels in mycoheterotrofe planten te identificeren
Summary
Het huidige artikel heeft tot doel gedetailleerde en adequate protocollen te bieden voor de isolatie van plant-geassocieerde endofytische schimmels, langdurige bewaring van isolaten, morfologische karakterisering en totale DNA-extractie voor daaropvolgende moleculaire identificatie en metagenomische analyses.
Abstract
Mycoheterotrofe planten vertonen een van de meest extreme vormen van mycorrhiza-afhankelijkheid, omdat ze hun autotrofe capaciteit volledig hebben verloren. Net zo essentieel als elke andere vitale hulpbron, zijn de schimmels waarmee deze planten nauw associëren essentieel voor hen. Daarom zijn enkele van de meest relevante technieken bij het bestuderen van mycoheterotrofe soorten degene die het onderzoek van geassocieerde schimmels mogelijk maken, met name die welke wortels en ondergrondse organen bewonen. In deze context worden vaak technieken toegepast voor het identificeren van cultuurafhankelijke en cultuuronafhankelijke endofytische schimmels. Het isoleren van schimmelendofyten biedt een middel om ze morfologisch te identificeren, hun diversiteit te analyseren en inocula te behouden voor toepassingen in de symbiotische ontkieming van orchideeënzaden. Het is echter bekend dat er een grote verscheidenheid aan niet-kweekbare schimmels is die plantenweefsels bewonen. Cultuuronafhankelijke moleculaire identificatietechnieken bieden dus een bredere dekking van soortendiversiteit en -overvloed. Dit artikel beoogt de methodologische ondersteuning te bieden die nodig is voor het opstarten van twee onderzoeksprocedures: een cultuurafhankelijke en een onafhankelijke. Met betrekking tot het cultuurafhankelijke protocol worden de processen voor het verzamelen en bewaren van plantenmonsters van verzamelplaatsen naar laboratoriumfaciliteiten gedetailleerd, samen met het isoleren van draadvormige schimmels uit ondergrondse en luchtorganen van mycoheterotrofe planten, het bijhouden van een verzameling isolaten, het morfologisch karakteriseren van hyfen door middel van een objectglaasjescultuurmethodologie en moleculaire identificatie van schimmels door totale DNA-extractie. De gedetailleerde procedures omvatten cultuuronafhankelijke methodologieën en omvatten het verzamelen van plantenmonsters voor metagenomische analyses en totale DNA-extractie uit achlorofylachtige plantenorganen met behulp van een commerciële kit. Ten slotte worden ook continuïteitsprotocollen (bijv. polymerasekettingreactie [PCR], sequencing) voorgesteld voor analyses, en technieken worden hier gepresenteerd.
Introduction
Endofytische schimmels zijn per definitie schimmels die het inwendige van plantenorganen en -weefsels bewonen bij onopvallende infecties (d.w.z. zonder schade toe te brengen aan hun gastheer)1,2. Deze schimmels kunnen neutraal of gunstig interageren met waardplanten, kunnen resistentie verlenen tegen ziekteverwekkers en ongunstige omgevingsomstandigheden, en kunnen bijdragen aan de synthese van nuttige verbindingen voor de plant (bijv. groeifactoren en andere fytohormonen)1,3. Mycorrhiza-endofyten zijn schimmels die mycorrhiza-associaties met de plant tot stand brengen en deelnemen aan de overdracht van voedingsstoffen4. In Orchidaceae is de interactie met mycorrhiza-endofyten van fundamenteel belang voor het ontkiemen van zaden in de overgrote meerderheid van de soorten, en voor de vestiging van zaailingen in alle planten in defamilie5. In dergelijke contexten vertegenwoordigen mycoheterotrofe orchideeën een geval van totale afhankelijkheid van hun mycorrhiza-partners, aangezien ze gedurende hun hele levenscyclus afhankelijk zijn van de overdracht van minerale voedingsstoffen en koolstofverbindingen door deze schimmels6. Daarom is de isolatie en identificatie van associërende schimmels een fundamentele basis bij het onderzoeken van mycoheterotrofe levensstrategieën. Bovendien is er weinig bekend over de rol van schimmelendofyten in mycoheterotrofe planten of zelfs de werkelijke diversiteit van deze schimmels 7,8.
Het onderzoek naar endofytische schimmels kan worden uitgevoerd met behulp van verschillende technieken, die traditioneel als cultuuronafhankelijk of -afhankelijk worden omschreven, bijvoorbeeld: (a) directe observatie, (b) schimmelisolatie en morfologische en/of moleculaire identificatie, en (c) totale DNA-extractie van plantenweefsels en moleculaire identificatie9. In directe observatie (a) kunnen endofytische schimmels worden onderzocht terwijl ze zich nog in het inwendige van plantencellen en weefsels bevinden door middel van licht- of elektronenmicroscopie9, aangezien verschillende microscopieprotocollen worden beschreven door Pena-Passos et al.10. Door isolatiemethoden (b) kunnen endofyten van schimmels worden gekarakteriseerd op basis van hun kolonies, hyfen en morfologie van de voortplantings- of resistentiestructuur. Ook is het via isolatietechnieken mogelijk om de moleculaire identificatie van isolaten uit te voeren door middel van DNA-extractie, amplificatie van moleculaire identificatiesequenties (barcodes of vingerafdrukken) en sequencing11. Deze laatste techniek (c) maakt de moleculaire identificatie van endofytische schimmels mogelijk door middel van DNA-extractie in het inwendige van plantenweefsels (metabarcoding), gevolgd door bibliotheekvoorbereiding en sequencing12.
Bovendien kunnen schimmelisolaten worden toegepast in symbiotische kiemproeven, met behulp van zaden van autotrofe of mycoheterotrofe orchideeën. Een voorbeeld van een dergelijke toepassing is het onderzoek van Sisti et al.13, waarin de kieming en de beginstadia van de ontwikkeling van protocormen worden beschreven in Pogoniopsis schenckii, een mycoheterotrofe orchidee, in combinatie met enkele van zijn isolaten, bestaande uit niet-mycorrhiza-endofytische schimmels. Het toegepaste symbiotische kiemprotocol wordt gedetailleerd beschreven en gepresenteerd in een video van Pena-Passos et al.10. Het isoleren van schimmels in associatie met verschillende plantenorganen maakt divers onderzoek mogelijk met betrekking tot de aard van plant-schimmelinteracties (bijv. om ecologische of fysiologische aspecten van de associatie te begrijpen, evenals onderzoek naar de overdracht van voedingsstoffen van schimmels naar de plant)9.
De methodologieën die in sectie 1 worden gepresenteerd, zijn gebaseerd op een verzameling van ondergrondse orgaanmonsters, aangezien deze organen de meeste moeilijkheden opleveren bij het verzamelen, en ze zijn van groot belang omdat mycorrhiza-endofyten ze koloniseren. Beide opgenomen protocollen (stappen 1.1 en 1.2) kunnen echter worden toegepast op andere mycoheterotrofe plantenorganen (bijv. wortelstokken, bloemstengels en vruchten). De in stap 1.1 beschreven verzamelmethode is bedoeld voor het isoleren van endofytische schimmels (sectie 2), voor morfologische karakterisering (secties 4 en 5) en/of totale DNA-extractie voor isolaatidentificatie (sectie 6). Aan de andere kant wordt de in stap 1.2 beschreven verzamelmethode uitsluitend gebruikt voor de totale DNA-extractie van plantenweefsels voor metabarcodingtechnieken (punt 7). In hoofdstuk 3 worden vier methoden voor de opslag en conservering van draadvormige schimmels gepresenteerd, twee voor opslag op korte termijn (3-6 maanden) en de andere twee geschikt voor opslag op lange termijn (>1 jaar). De morfologische karakterisering (secties 4 en 5) kan in verband worden gebracht met moleculaire identificatie om deze te versterken en belangrijke informatie te verschaffen over de macro- en micromorfologie van schimmels. Figuur 1 geeft een overzicht van de collectieve methodologieën die daarna worden beschreven.
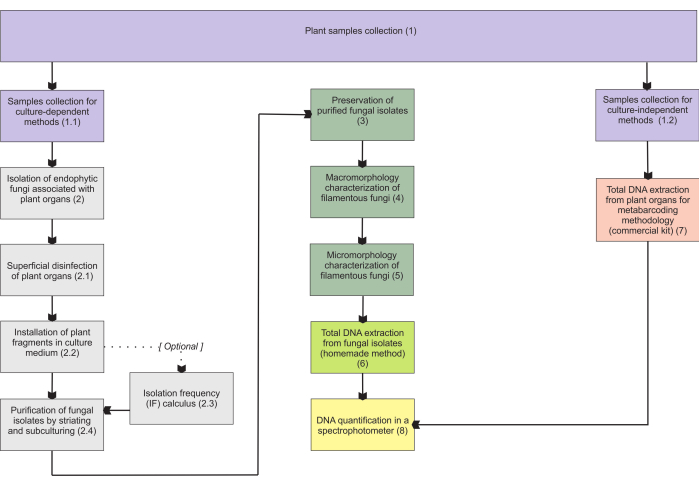
Figuur 1: Schematische samenvatting van de gepresenteerde methoden. Plantenverzameling en schimmelisolatie, conservering en moleculaire identificatie door middel van cultuurafhankelijke en -onafhankelijke methodologieën. Klik hier om een grotere versie van deze figuur te bekijken.
Protocol
Representative Results
Discussion
De oppervlakkige ontsmetting van plantenmonsters is een van de meest kritieke fasen in het gepresenteerde protocol. Geen contaminatie in de PDA-vaat met druppels van de laatste wasbeurt is zeer gewenst. Bacteriën worden vaak waargenomen als verontreinigingen in de isolatieschalen, meestal meer dan sporulerende schimmels in de lucht, aangezien endofytische bacteriën ook veel voorkomen in plantenweefsels 3,11. De toevoeging van antibiotica in het kweekmedium bij …
Declarações
The authors have nothing to disclose.
Acknowledgements
We danken de financiering van FAPESP (2015/26479-6) en CNPq (447453/2014-9). JLSM bedankt CNPq voor productiviteitssubsidies (303664/2020-7). MPP bedankt Capes (master’s degree scholarship, proces 88887.600591/2021-00) en CNPq.
Materials
| Adhesive tape | (from any company, for adhesive tape mount in micromorphological analyses) | ||
| Ampicillin | Sigma-Aldrich | A5354 | (for installation of plant fragments; other antibiotics may be used – check step 2.2.1) |
| Autoclave | (from any company, for materials sterilization in many steps) | ||
| Bacteriological agar | Sigma-Aldrich | A1296 | (for many steps) |
| C1, C2, C3, C4, C5, and C6 solutions | Qiagen | 12888-50 | (purchased with DNeasy PowerSoil kit) |
| Centrifuge | Merck/Eppendorf | 5810 G | (for total DNA extraction from fungal isolates) |
| Centrifuge tubes | Merck | CLS430828 | (for samples collection) |
| Chloroform | Sigma-Aldrich | C2432 | (for total DNA extraction from fungal isolates) |
| Congo red | Supelco | 75768 | (for hyphae staining) |
| Cryotubes | Merck | BR114831 | (for many steps) |
| Ethanol | Supelco | 100983 | It will be necessary to carry out the appropriate dilutions (for many steps) |
| Ethylenediaminetetraacetic acid (EDTA) | Sigma-Aldrich | 3609 | (for total DNA extraction from fungal isolates) |
| Filter paper | Merck | WHA10010155 | (for many steps) |
| Glass test tubes | Merck | CLS7082516 | (for cryopreservation in unhulled rice grains) |
| Glass wool | Supelco | 20411 | (for cryopreservation in unhulled rice grains) |
| Glucose | Sigma-Aldrich | G8270 | Or dextrose (for cryopreservation in vermiculite) |
| Glycerol | Sigma-Aldrich | G5516 | Or glycerin (for cryopreservation in vermiculite, for preparing LPCB) |
| Isopropanol | Sigma-Aldrich | 563935 | (for total DNA extraction from fungal isolates) |
| Lactic acid | Sigma-Aldrich | 252476 | (for preparing LPCB – hyphae staining) |
| Lactophenol blue solution (LPCB) | Sigma-Aldrich | 61335 | (for hyphae staining) |
| Laminar flow hood | (class I, from any company, for many steps) | ||
| Light microscope | (from any company, for hyphae observation) | ||
| MB Spin Columns | Qiagen | 12888-50 | (purchased with DNeasy PowerSoil kit) |
| Methyl blue (cotton blue) | Sigma-Aldrich | M5528 | (for preparing LPCB – hyphae staining) |
| Microcentrifuge tube (1.5 mL) | Merck | HS4323 | (for total DNA extraction from fungal isolates) |
| Microcentrifuge tube (2 mL) | Merck | BR780546 | (for many steps) |
| Mineral oil | (for preservation of fungal isolates) | ||
| Paper bags | Average size 150 mm x 200 mm (for samples collection) | ||
| Petri dish (Glass, 120 mm x 20 mm) | Merck/Pyrex | SLW1480/10D | (autoclavable, for fungi slide culture, prefer higher ones) |
| Petri dish (Glass, 50 mm x 17 mm) | Merck/Aldrich | Z740618 | (for purification of fungal isolates); alternatively: polystyrene petri dishes (sterile, γ-irradiated, non-autoclavable) |
| Petri dish (Glass, 80 mm x 15 mm) | Merck/Brand | BR455732 | (for installation of plant fragments); alternatively: polystyrene petri dishes (sterile, γ-irradiated, non-autoclavable) |
| Phenol | Sigma-Aldrich | P1037 | (for total DNA extraction from fungal isolates, for preparing LPCB) |
| Porcelain mortar | Sigma-Aldrich | Z247464 | (for total DNA extraction from fungal isolates) |
| Porcelain pestle | Sigma-Aldrich | Z247502 | (for total DNA extraction from fungal isolates) |
| Potato dextrose agar (PDA) | Millipore | P2182 | (for many steps) |
| PowerBead tubes | Qiagen | 12888-50 | (purchased with DNeasy PowerSoil kit) |
| Rapid mounting medium (Entellan) | Sigma-Aldrich | 1.0796 | (for fungi slide culture) |
| Silica gel | Supelco | 717185 | (for cryopreservation in unhulled rice grains) |
| Sodium chloride (NaCl) | Sigma-Aldrich | S9888 | (for total DNA extraction from fungal isolates) |
| Sodium dodecyl sulfate (SDS) | Sigma-Aldrich | L3771 | Lauryl sulfate sodium salt (for total DNA extraction from fungal isolates) |
| Sodium hypochlorite (w/ 2% active chlorine) | (commercial product, for superficial desinfestation) | ||
| Soil DNA extraction kit (DNeasy PowerSoil kit) | Qiagen | 12888-50 | (for total DNA extraction from plant organs) |
| Spectrophotometer – Nanodrop 2000/2000c | ThermoFisher Scientific | ND2000CLAPTOP | (for total DNA extraction from plant organs) |
| Stereomicroscope | (=dissecting microscope, from any company, for macromorphological analyses) | ||
| Tetracycline | Sigma-Aldrich | T7660 | (for installation of plant fragments) |
| Thermoblock | Merck/Eppendorf | EP5362000035 | (or from other companies) |
| Tissue homogenizer and cell lyzer | SPEX SamplePrep | 2010 Geno/Grinder – Automated Tissue Homogenizer and Cell Lyzer (for total DNA extraction from plant organs) | |
| Toluidine blue O | Sigma-Aldrich/Harleco | 364-M | (for hyphae staining) |
| Trehalose | Sigma-Aldrich | T9531 | (for cryopreservation in vermiculite) |
| Tris Base Solution (Tris) | Sigma-Aldrich | T1699 | (for total DNA extraction from fungal isolates) |
| Unhulled rice grains | (for cryopreservation) | ||
| U-shaped glass rod | (or an adaptation – check step 5.4.1, for fungi slide culture) | ||
| Vermiculite | Fine granulometry (for cryopreservation in vermiculite) | ||
| Vortexer | Sigma-Aldrich/BenchMixer | BMSBV1000 | (for total DNA extraction from fungal isolates) |
| Yeast extract | Sigma-Aldrich | Y1625 | (for cryopreservation in vermiculite) |
Referências
- de Azevedo, J. L. Endophytic microorganisms. Ecologia Microbiana. , 117-137 (1998).
- Stone, J. K., Bacon, C. W., White, J. F. An overview of endophytic microbes: endophytism defined. Microbial Endophytes. , 17-44 (2000).
- Schulz, B., Boyle, C. What are Endophytes. Microbial Root Endophytes. , 1-13 (2006).
- Smith, S. E., Read, D. J. . Mycorrhizal Symbiosis. , (2008).
- Rasmussen, H. N., Dixon, K. W., Jersáková, J., Těšitelová, T. Germination and seedling establishment in orchids: a complex of requirements. Annals of Botany. 116 (3), 391-402 (2015).
- Rasmussen, H. N., Rasmussen, F. N. Orchid mycorrhiza: implications of a mycophagous life style. Oikos. 118 (3), 334-345 (2009).
- Ma, X., Kang, J., Nontachaiyapoom, S., Wen, T., Hyde, K. D. Non-mycorrhizal endophytic fungi from orchids. Current Science. 109 (1), 72-87 (2015).
- Favre-Godal, Q., Gourguillon, L., Lordel-Madeleine, S., Gindro, K., Choisy, P. Orchids and their mycorrhizal fungi: an insufficiently explored relationship. Mycorrhiza. 30 (1), 5-22 (2020).
- Sun, X., Guo, L. -. D. Endophytic fungal diversity: review of traditional and molecular techniques. Mycology. 3 (1), 65-76 (2012).
- Pena-Passos, M., Sisti, L. S., Mayer, J. L. S. Microscopy techniques for interpreting fungal colonization in mycoheterotrophic plants tissues and symbiotic germination of seeds. Journal of Visualized Experiments. (183), e63777 (2022).
- Araújo, W. L., et al. . Endophytic microorganisms: Theoretical and Practical Aspects of Isolation and Characterization. 1st ed. 1, 257 (2014).
- de Souza, R. S. C., et al. Unlocking the bacterial and fungal communities assemblages of sugarcane microbiome. Scientific Reports. 6, 28774 (2016).
- Sisti, L. S., et al. The role of non-mycorrhizal fungi in germination of the mycoheterotrophic orchid Pogoniopsis schenckii Cogn. Frontiers in Plant Science. 10, 1589 (2019).
- Araújo, W. L., et al. Variability and interactions between endophytic bacteria and fungi isolated from leaf tissues of citrus rootstocks. Canadian Journal of Microbiology. 47 (3), 229-236 (2001).
- Castellani, A. Further researches on the long viability and growth of many pathogenic fungi and some bacteria in sterile distilled water. Mycopathologia. 20 (1-2), 1-6 (1963).
- Currah, R. S., Zelmer, C. D., Hambleton, S., Richardson, K. A. Fungi from orchid mycorrhizas. Orchid Biology: Reviews and Perspectives, VII. , 117-170 (1997).
- Freitas, E. F. S., et al. Diversity of mycorrhizal Tulasnella associated with epiphytic and rupicolous orchids from the Brazilian Atlantic Forest, including four new species. Scientific Reports. 10 (1), 7069 (2020).
- Sato, M., Inaba, S., Noguchi, M., Nakagiri, A. Vermiculite as a culture substrate greatly improves the viability of frozen cultures of ectomycorrhizal basidiomycetes. Fungal Biology. 124 (8), 742-751 (2020).
- Pereira, O. L., Kasuya, M. C. M., Borges, A. C., Araújo, E. F. D. Morphological and molecular characterization of mycorrhizal fungi isolated from neotropical orchids in Brazil. Canadian Journal of Botany. 83 (1), 54-65 (2005).
- Riddell, R. W. Permanent stained mycological preparations obtained by slide culture. Mycologia. 42 (2), 265-270 (1950).
- Walsh, T. J., Hayden, R. T., Larone, D. H. . Larone’s Medically Important Fungi: A Guide to Identification. , (2018).
- Microscopy: Chemical Reagents. British Mycological Society Available from: https://www.britmycolsoc.org.uk/field_mycology/microscopy/reagents (2022)
- Senanayake, I. C., et al. Morphological approaches in studying fungi: Collection, examination, isolation, sporulation and preservation. Mycosphere. 11 (1), 2678-2754 (2020).
- Slifkin, M., Cumbie, R. Congo red as a fluorochrome for the rapid detection of fungi. Journal of Clinical Microbiology. 26 (5), 827-830 (1988).
- Raeder, U., Broda, P. Rapid preparation of DNA from filamentous fungi. Letters in Applied Microbiology. 1 (1), 17-20 (1985).
- Martins, M. K., et al. Molecular characterization of endophytic microorganisms. Endophytic microorganisms: theoretical and practical aspects of isolation and characterization. 1st edition. , 189-211 (2014).
- Rayner, R. W. A Mycological Colour Chart. Commonwealth Mycological Institute. , (1970).
- Kornerup, A., Wanscher, J. H. . Methuen Handbook of Colour. Methuen handbook of colour. , (1967).
- Ridgway, R. . Color Standards and Color Nomenclature. , (1912).
- McGinnis, M. R. . Laboratory Handbook of Medical Mycology. , (2012).
- Webster, J., Weber, R. . Introduction to Fungi. , (2007).
- Sridharan, G., Shankar, A. A. Toluidine blue: A review of its chemistry and clinical utility. Journal of Oral and Maxillofacial Pathology. 16 (2), 251-255 (2012).
- Smith, D., Onions, A. H. S. A comparison of some preservation techniques for fungi. Transactions of the British Mycological Society. 81 (3), 535-540 (1983).
- Ryan, M. J., Smith, D., Jeffries, P. A decision-based key to determine the most appropriate protocol for the preservation of fungi. World Journal of Microbiology and Biotechnology. 16 (2), 183-186 (2000).
- Lalaymia, I., Cranenbrouck, S., Declerck, S. Maintenance and preservation of ectomycorrhizal and arbuscular mycorrhizal fungi. Mycorrhiza. 24 (5), 323-337 (2014).
- Zettler, L. W., Corey, L. L. Orchid mycorrhizal fungi: isolation and identification techniques. Orchid Propagation: From Laboratories to Greenhouses-Methods and Protocols. , 27-59 (2018).
- Yu, S., Wang, Y., Li, X., Yu, F., Li, W. The factors affecting the reproducibility of micro-volume DNA mass quantification in Nanodrop 2000 spectrophotometer. Optik. 145, 555-560 (2017).
- Martos, F., et al. Independent recruitment of saprotrophic fungi as mycorrhizal partners by tropical achlorophyllous orchids. New Phytologist. 184 (3), 668-681 (2009).
- Schoch, C. L., et al. Nuclear ribosomal internal transcribed spacer (ITS) region as a universal DNA barcode marker for Fungi. Proceedings of the National Academy of Sciences. 109 (16), 6241-6246 (2012).
- White, T. J., Bruns, T., Lee, S., Taylor, J. Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics. PCR Protocols: A Guide to Methods and Applications. 18 (1), 315-322 (1990).
- Sanger, F., Nicklen, S., Coulson, A. R. DNA sequencing with chain-terminating inhibitors. Proceedings of the National Academy of Sciences. 74 (12), 5463-5467 (1977).
- Ranjard, L., et al. Characterization of bacterial and fungal soil communities by automated ribosomal intergenic spacer analysis fingerprints: biological and methodological variability. Applied and Environmental Microbiology. 67 (10), 4479-4487 (2001).
- Metzker, M. L. Sequencing technologies-the next generation. Nature Reviews Genetics. 11 (1), 31-46 (2010).

