Aislamiento, caracterización y extracción de ADN total para identificar hongos endófitos en plantas micoheterótrofas
Summary
El presente artículo tiene como objetivo proporcionar protocolos detallados y adecuados para el aislamiento de hongos endófitos asociados a plantas, la preservación a largo plazo de aislados, la caracterización morfológica y la extracción de ADN total para su posterior identificación molecular y análisis metagenómicos.
Abstract
Las plantas micoheterótrofas presentan una de las formas más extremas de dependencia micorrícica, habiendo perdido totalmente su capacidad autótrofa. Tan esencial como cualquier otro recurso vital, los hongos con los que estas plantas se asocian íntimamente son esenciales para ellas. De ahí que algunas de las técnicas más relevantes en el estudio de especies micoheterótrofas sean las que permiten la investigación de hongos asociados, especialmente aquellos que habitan en raíces y órganos subterráneos. En este contexto, se aplican comúnmente técnicas para identificar hongos endófitos dependientes e independientes del cultivo. El aislamiento de endófitos fúngicos proporciona un medio para identificarlos morfológicamente, analizar su diversidad y mantener los inóculos para aplicaciones en la germinación simbiótica de semillas de orquídeas. Sin embargo, se sabe que existe una gran variedad de hongos no cultivables que habitan en los tejidos vegetales. Por lo tanto, las técnicas de identificación molecular independientes del cultivo ofrecen una cobertura más amplia de la diversidad y abundancia de especies. El objetivo de este artículo es proporcionar el apoyo metodológico necesario para iniciar dos procedimientos de investigación: uno dependiente de la cultura y otro independiente. En cuanto al protocolo dependiente del cultivo, se detallan los procesos de recolección y mantenimiento de muestras de plantas desde los sitios de recolección hasta las instalaciones de laboratorio, junto con el aislamiento de hongos filamentosos de órganos subterráneos y aéreos de plantas micoheterótrofas, el mantenimiento de una colección de aislados, la caracterización morfológica de las hifas mediante la metodología de cultivo de portaobjetos y la identificación molecular de hongos mediante la extracción total de ADN. Abarcando metodologías independientes del cultivo, los procedimientos detallados incluyen la recolección de muestras de plantas para análisis metagenómicos y la extracción total de ADN de órganos de plantas aclorofílicas utilizando un kit comercial. Por último, también se sugieren protocolos de continuidad (p. ej., reacción en cadena de la polimerasa [PCR], secuenciación) para los análisis, y aquí se presentan técnicas.
Introduction
Los hongos endófitos son, por definición, aquellos que habitan en el interior de los órganos y tejidos de las plantas en infecciones discretas (es decir, sin causar daño a su huésped)1,2. Estos hongos pueden interactuar de forma neutra o beneficiosa con las plantas hospederas, pueden conferir resistencia a patógenos y condiciones ambientales desfavorables, y pueden contribuir a la síntesis de compuestos beneficiosos para la planta (por ejemplo, factores de crecimiento y otras fitohormonas)1,3. Los endófitos micorrícicos son hongos que establecen asociaciones micorrícicas con la planta, participando en la transferencia de nutrientes4. En Orchidaceae, la interacción con endófitos micorrícicos es fundamental para la germinación de semillas en la gran mayoría de las especies, y el establecimiento de plántulas en todas las plantas de la familia5. En tales contextos, las orquídeas micoheterótrofas representan un caso de dependencia total con respecto a sus socios micorrícicos, ya que dependen de la transferencia de nutrientes minerales y compuestos de carbono por parte de estos hongos durante todo su ciclo de vida6. Por lo tanto, el aislamiento e identificación de hongos asociados es una base fundamental a la hora de investigar estrategias de vida micoheterótrofas. Además, poco se sabe sobre el papel de los endófitos fúngicos en las plantas micoheterótrofas o incluso la diversidad real de estos hongos 7,8.
La investigación de hongos endófitos puede llevarse a cabo mediante diferentes técnicas, tradicionalmente descritas como independientes o dependientes del cultivo, por ejemplo: (a) observación directa, (b) aislamiento de hongos e identificación morfológica y/o molecular, y (c) extracción total de ADN de tejidos vegetales e identificación molecular9. En observación directa (a), los hongos endófitos pueden ser investigados mientras aún se encuentran en el interior de células y tejidos vegetales mediante microscopía óptica o electrónica9, ya que los diferentes protocolos de microscopía son detallados por Peña-Passos et al.10. Mediante métodos de aislamiento (b), los endófitos fúngicos pueden caracterizarse de acuerdo con sus colonias, hifas y morfología de la estructura reproductiva o de resistencia. Además, a través de técnicas de aislamiento, es posible llevar a cabo la identificación molecular de aislados a través de la extracción de ADN, la amplificación de secuencias de identificación molecular (códigos de barras o huellas dactilares) y la secuenciación11. Esta última técnica (c) permite la identificación molecular de hongos endófitos mediante la extracción de ADN en el interior de los tejidos vegetales (metacódigo de barras), seguida de la preparación y secuenciación de la biblioteca12.
Además, los aislados de hongos pueden aplicarse en ensayos de germinación simbiótica, utilizando semillas de orquídeas autótrofas o micoheterótrofas. Un ejemplo de esta aplicación es la investigación realizada por Sisti et al.13, describiendo la germinación y las etapas iniciales del desarrollo de protocormos en Pogoniopsis schenckii, una orquídea micoheterótrofa, en asociación con algunos de sus aislados, que comprenden hongos endófitos no micorrícicos. El protocolo de germinación simbiótica aplicado se detalla y presenta en un video de Pena-Passos et al.10. El aislamiento de hongos en asociación con diferentes órganos de la planta permite diversos enfoques de investigación con respecto a la naturaleza de las interacciones planta-hongo (por ejemplo, para comprender los aspectos ecológicos o fisiológicos de la asociación, así como indagaciones sobre la transferencia de nutrientes de los hongos a la planta)9.
Las metodologías presentadas en la sección 1 se basan en la recolección de muestras de órganos subterráneos, ya que estos órganos presentan más dificultades en la recolección, y son de gran interés ya que los endófitos micorrícicos los colonizan. Sin embargo, ambos protocolos incluidos (pasos 1.1 y 1.2) pueden aplicarse a otros órganos de plantas micoheterótrofas (p. ej., rizomas, tallos florales y frutos). La metodología de recolección descrita en el paso 1.1 está diseñada para aislar hongos endófitos (sección 2), para la caracterización morfológica (secciones 4 y 5) y/o la extracción total de ADN para la identificación aislada (sección 6). Por otro lado, la metodología de recolección descrita en el paso 1.2 se asigna exclusivamente a la extracción de ADN total de tejidos vegetales para técnicas de metabarcoding (sección 7). En la sección 3 se presentan cuatro métodos de almacenamiento y conservación de hongos filamentosos, dos para el almacenamiento a corto plazo (3-6 meses) y los otros dos adecuados para el almacenamiento a largo plazo (>1 año). La caracterización morfológica (secciones 4 y 5) puede asociarse con la identificación molecular para reforzarla y proporcionar información importante sobre la macro y micromorfología fúngica. En la Figura 1 se resumen las metodologías colectivas que se describen a continuación.
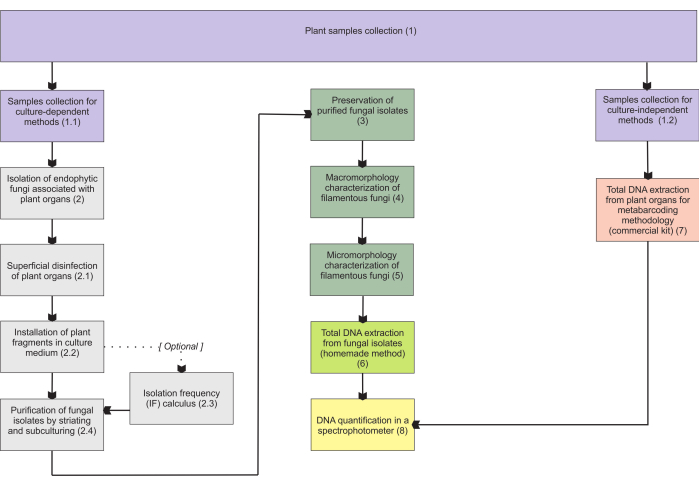
Figura 1: Resumen esquemático de los métodos presentados. Recolección de plantas y aislamiento, preservación e identificación molecular de hongos mediante metodologías dependientes e independientes del cultivo. Haga clic aquí para ver una versión más grande de esta figura.
Protocol
Representative Results
Discussion
La desinfestación superficial de muestras de plantas es una de las etapas más críticas en el protocolo presentado. Es muy deseable que no se contaminen las antenas PDA con gotas del último lavado. Las bacterias se observan con frecuencia como contaminantes en las placas de aislamiento, generalmente más que los hongos esporulantes en el aire, considerando que las bacterias endófitas también son comunes dentro de los tejidos vegetales 3,11. Por lo tanto, la …
Declarações
The authors have nothing to disclose.
Acknowledgements
Agradecemos el financiamiento de la FAPESP (2015/26479-6) y del CNPq (447453/2014-9). JLSM agradece al CNPq por las becas de productividad (303664/2020-7). El MPP agradece a la Capes (beca de maestría, proceso 88887.600591/2021-00) y al CNPq.
Materials
| Adhesive tape | (from any company, for adhesive tape mount in micromorphological analyses) | ||
| Ampicillin | Sigma-Aldrich | A5354 | (for installation of plant fragments; other antibiotics may be used – check step 2.2.1) |
| Autoclave | (from any company, for materials sterilization in many steps) | ||
| Bacteriological agar | Sigma-Aldrich | A1296 | (for many steps) |
| C1, C2, C3, C4, C5, and C6 solutions | Qiagen | 12888-50 | (purchased with DNeasy PowerSoil kit) |
| Centrifuge | Merck/Eppendorf | 5810 G | (for total DNA extraction from fungal isolates) |
| Centrifuge tubes | Merck | CLS430828 | (for samples collection) |
| Chloroform | Sigma-Aldrich | C2432 | (for total DNA extraction from fungal isolates) |
| Congo red | Supelco | 75768 | (for hyphae staining) |
| Cryotubes | Merck | BR114831 | (for many steps) |
| Ethanol | Supelco | 100983 | It will be necessary to carry out the appropriate dilutions (for many steps) |
| Ethylenediaminetetraacetic acid (EDTA) | Sigma-Aldrich | 3609 | (for total DNA extraction from fungal isolates) |
| Filter paper | Merck | WHA10010155 | (for many steps) |
| Glass test tubes | Merck | CLS7082516 | (for cryopreservation in unhulled rice grains) |
| Glass wool | Supelco | 20411 | (for cryopreservation in unhulled rice grains) |
| Glucose | Sigma-Aldrich | G8270 | Or dextrose (for cryopreservation in vermiculite) |
| Glycerol | Sigma-Aldrich | G5516 | Or glycerin (for cryopreservation in vermiculite, for preparing LPCB) |
| Isopropanol | Sigma-Aldrich | 563935 | (for total DNA extraction from fungal isolates) |
| Lactic acid | Sigma-Aldrich | 252476 | (for preparing LPCB – hyphae staining) |
| Lactophenol blue solution (LPCB) | Sigma-Aldrich | 61335 | (for hyphae staining) |
| Laminar flow hood | (class I, from any company, for many steps) | ||
| Light microscope | (from any company, for hyphae observation) | ||
| MB Spin Columns | Qiagen | 12888-50 | (purchased with DNeasy PowerSoil kit) |
| Methyl blue (cotton blue) | Sigma-Aldrich | M5528 | (for preparing LPCB – hyphae staining) |
| Microcentrifuge tube (1.5 mL) | Merck | HS4323 | (for total DNA extraction from fungal isolates) |
| Microcentrifuge tube (2 mL) | Merck | BR780546 | (for many steps) |
| Mineral oil | (for preservation of fungal isolates) | ||
| Paper bags | Average size 150 mm x 200 mm (for samples collection) | ||
| Petri dish (Glass, 120 mm x 20 mm) | Merck/Pyrex | SLW1480/10D | (autoclavable, for fungi slide culture, prefer higher ones) |
| Petri dish (Glass, 50 mm x 17 mm) | Merck/Aldrich | Z740618 | (for purification of fungal isolates); alternatively: polystyrene petri dishes (sterile, γ-irradiated, non-autoclavable) |
| Petri dish (Glass, 80 mm x 15 mm) | Merck/Brand | BR455732 | (for installation of plant fragments); alternatively: polystyrene petri dishes (sterile, γ-irradiated, non-autoclavable) |
| Phenol | Sigma-Aldrich | P1037 | (for total DNA extraction from fungal isolates, for preparing LPCB) |
| Porcelain mortar | Sigma-Aldrich | Z247464 | (for total DNA extraction from fungal isolates) |
| Porcelain pestle | Sigma-Aldrich | Z247502 | (for total DNA extraction from fungal isolates) |
| Potato dextrose agar (PDA) | Millipore | P2182 | (for many steps) |
| PowerBead tubes | Qiagen | 12888-50 | (purchased with DNeasy PowerSoil kit) |
| Rapid mounting medium (Entellan) | Sigma-Aldrich | 1.0796 | (for fungi slide culture) |
| Silica gel | Supelco | 717185 | (for cryopreservation in unhulled rice grains) |
| Sodium chloride (NaCl) | Sigma-Aldrich | S9888 | (for total DNA extraction from fungal isolates) |
| Sodium dodecyl sulfate (SDS) | Sigma-Aldrich | L3771 | Lauryl sulfate sodium salt (for total DNA extraction from fungal isolates) |
| Sodium hypochlorite (w/ 2% active chlorine) | (commercial product, for superficial desinfestation) | ||
| Soil DNA extraction kit (DNeasy PowerSoil kit) | Qiagen | 12888-50 | (for total DNA extraction from plant organs) |
| Spectrophotometer – Nanodrop 2000/2000c | ThermoFisher Scientific | ND2000CLAPTOP | (for total DNA extraction from plant organs) |
| Stereomicroscope | (=dissecting microscope, from any company, for macromorphological analyses) | ||
| Tetracycline | Sigma-Aldrich | T7660 | (for installation of plant fragments) |
| Thermoblock | Merck/Eppendorf | EP5362000035 | (or from other companies) |
| Tissue homogenizer and cell lyzer | SPEX SamplePrep | 2010 Geno/Grinder – Automated Tissue Homogenizer and Cell Lyzer (for total DNA extraction from plant organs) | |
| Toluidine blue O | Sigma-Aldrich/Harleco | 364-M | (for hyphae staining) |
| Trehalose | Sigma-Aldrich | T9531 | (for cryopreservation in vermiculite) |
| Tris Base Solution (Tris) | Sigma-Aldrich | T1699 | (for total DNA extraction from fungal isolates) |
| Unhulled rice grains | (for cryopreservation) | ||
| U-shaped glass rod | (or an adaptation – check step 5.4.1, for fungi slide culture) | ||
| Vermiculite | Fine granulometry (for cryopreservation in vermiculite) | ||
| Vortexer | Sigma-Aldrich/BenchMixer | BMSBV1000 | (for total DNA extraction from fungal isolates) |
| Yeast extract | Sigma-Aldrich | Y1625 | (for cryopreservation in vermiculite) |
Referências
- de Azevedo, J. L. Endophytic microorganisms. Ecologia Microbiana. , 117-137 (1998).
- Stone, J. K., Bacon, C. W., White, J. F. An overview of endophytic microbes: endophytism defined. Microbial Endophytes. , 17-44 (2000).
- Schulz, B., Boyle, C. What are Endophytes. Microbial Root Endophytes. , 1-13 (2006).
- Smith, S. E., Read, D. J. . Mycorrhizal Symbiosis. , (2008).
- Rasmussen, H. N., Dixon, K. W., Jersáková, J., Těšitelová, T. Germination and seedling establishment in orchids: a complex of requirements. Annals of Botany. 116 (3), 391-402 (2015).
- Rasmussen, H. N., Rasmussen, F. N. Orchid mycorrhiza: implications of a mycophagous life style. Oikos. 118 (3), 334-345 (2009).
- Ma, X., Kang, J., Nontachaiyapoom, S., Wen, T., Hyde, K. D. Non-mycorrhizal endophytic fungi from orchids. Current Science. 109 (1), 72-87 (2015).
- Favre-Godal, Q., Gourguillon, L., Lordel-Madeleine, S., Gindro, K., Choisy, P. Orchids and their mycorrhizal fungi: an insufficiently explored relationship. Mycorrhiza. 30 (1), 5-22 (2020).
- Sun, X., Guo, L. -. D. Endophytic fungal diversity: review of traditional and molecular techniques. Mycology. 3 (1), 65-76 (2012).
- Pena-Passos, M., Sisti, L. S., Mayer, J. L. S. Microscopy techniques for interpreting fungal colonization in mycoheterotrophic plants tissues and symbiotic germination of seeds. Journal of Visualized Experiments. (183), e63777 (2022).
- Araújo, W. L., et al. . Endophytic microorganisms: Theoretical and Practical Aspects of Isolation and Characterization. 1st ed. 1, 257 (2014).
- de Souza, R. S. C., et al. Unlocking the bacterial and fungal communities assemblages of sugarcane microbiome. Scientific Reports. 6, 28774 (2016).
- Sisti, L. S., et al. The role of non-mycorrhizal fungi in germination of the mycoheterotrophic orchid Pogoniopsis schenckii Cogn. Frontiers in Plant Science. 10, 1589 (2019).
- Araújo, W. L., et al. Variability and interactions between endophytic bacteria and fungi isolated from leaf tissues of citrus rootstocks. Canadian Journal of Microbiology. 47 (3), 229-236 (2001).
- Castellani, A. Further researches on the long viability and growth of many pathogenic fungi and some bacteria in sterile distilled water. Mycopathologia. 20 (1-2), 1-6 (1963).
- Currah, R. S., Zelmer, C. D., Hambleton, S., Richardson, K. A. Fungi from orchid mycorrhizas. Orchid Biology: Reviews and Perspectives, VII. , 117-170 (1997).
- Freitas, E. F. S., et al. Diversity of mycorrhizal Tulasnella associated with epiphytic and rupicolous orchids from the Brazilian Atlantic Forest, including four new species. Scientific Reports. 10 (1), 7069 (2020).
- Sato, M., Inaba, S., Noguchi, M., Nakagiri, A. Vermiculite as a culture substrate greatly improves the viability of frozen cultures of ectomycorrhizal basidiomycetes. Fungal Biology. 124 (8), 742-751 (2020).
- Pereira, O. L., Kasuya, M. C. M., Borges, A. C., Araújo, E. F. D. Morphological and molecular characterization of mycorrhizal fungi isolated from neotropical orchids in Brazil. Canadian Journal of Botany. 83 (1), 54-65 (2005).
- Riddell, R. W. Permanent stained mycological preparations obtained by slide culture. Mycologia. 42 (2), 265-270 (1950).
- Walsh, T. J., Hayden, R. T., Larone, D. H. . Larone’s Medically Important Fungi: A Guide to Identification. , (2018).
- Microscopy: Chemical Reagents. British Mycological Society Available from: https://www.britmycolsoc.org.uk/field_mycology/microscopy/reagents (2022)
- Senanayake, I. C., et al. Morphological approaches in studying fungi: Collection, examination, isolation, sporulation and preservation. Mycosphere. 11 (1), 2678-2754 (2020).
- Slifkin, M., Cumbie, R. Congo red as a fluorochrome for the rapid detection of fungi. Journal of Clinical Microbiology. 26 (5), 827-830 (1988).
- Raeder, U., Broda, P. Rapid preparation of DNA from filamentous fungi. Letters in Applied Microbiology. 1 (1), 17-20 (1985).
- Martins, M. K., et al. Molecular characterization of endophytic microorganisms. Endophytic microorganisms: theoretical and practical aspects of isolation and characterization. 1st edition. , 189-211 (2014).
- Rayner, R. W. A Mycological Colour Chart. Commonwealth Mycological Institute. , (1970).
- Kornerup, A., Wanscher, J. H. . Methuen Handbook of Colour. Methuen handbook of colour. , (1967).
- Ridgway, R. . Color Standards and Color Nomenclature. , (1912).
- McGinnis, M. R. . Laboratory Handbook of Medical Mycology. , (2012).
- Webster, J., Weber, R. . Introduction to Fungi. , (2007).
- Sridharan, G., Shankar, A. A. Toluidine blue: A review of its chemistry and clinical utility. Journal of Oral and Maxillofacial Pathology. 16 (2), 251-255 (2012).
- Smith, D., Onions, A. H. S. A comparison of some preservation techniques for fungi. Transactions of the British Mycological Society. 81 (3), 535-540 (1983).
- Ryan, M. J., Smith, D., Jeffries, P. A decision-based key to determine the most appropriate protocol for the preservation of fungi. World Journal of Microbiology and Biotechnology. 16 (2), 183-186 (2000).
- Lalaymia, I., Cranenbrouck, S., Declerck, S. Maintenance and preservation of ectomycorrhizal and arbuscular mycorrhizal fungi. Mycorrhiza. 24 (5), 323-337 (2014).
- Zettler, L. W., Corey, L. L. Orchid mycorrhizal fungi: isolation and identification techniques. Orchid Propagation: From Laboratories to Greenhouses-Methods and Protocols. , 27-59 (2018).
- Yu, S., Wang, Y., Li, X., Yu, F., Li, W. The factors affecting the reproducibility of micro-volume DNA mass quantification in Nanodrop 2000 spectrophotometer. Optik. 145, 555-560 (2017).
- Martos, F., et al. Independent recruitment of saprotrophic fungi as mycorrhizal partners by tropical achlorophyllous orchids. New Phytologist. 184 (3), 668-681 (2009).
- Schoch, C. L., et al. Nuclear ribosomal internal transcribed spacer (ITS) region as a universal DNA barcode marker for Fungi. Proceedings of the National Academy of Sciences. 109 (16), 6241-6246 (2012).
- White, T. J., Bruns, T., Lee, S., Taylor, J. Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics. PCR Protocols: A Guide to Methods and Applications. 18 (1), 315-322 (1990).
- Sanger, F., Nicklen, S., Coulson, A. R. DNA sequencing with chain-terminating inhibitors. Proceedings of the National Academy of Sciences. 74 (12), 5463-5467 (1977).
- Ranjard, L., et al. Characterization of bacterial and fungal soil communities by automated ribosomal intergenic spacer analysis fingerprints: biological and methodological variability. Applied and Environmental Microbiology. 67 (10), 4479-4487 (2001).
- Metzker, M. L. Sequencing technologies-the next generation. Nature Reviews Genetics. 11 (1), 31-46 (2010).

