Membrane Transport processos analisados por um Sistema Nanopore Chip altamente paralelos na resolução Protein Individual
Summary
The presented protocol describes the analysis of membrane protein mediated transport on the single transporter level using pore-spanning solvent-free lipid bilayers. This is achieved by the creation of bulk produced nanopore array chips, combined with highly parallel data acquisition and analysis, enabling the future establishment of membrane protein effector screenings.
Abstract
transporte de proteínas de membrana no nível de proteína único ainda escapa análise detalhada, se o substrato translocado é não-electrogenic. Esforços consideráveis têm sido feitas neste campo, mas as técnicas que permitem a análise automatizada de transporte de alto rendimento, em combinação com técnicas de bicamada lipídica livre de solvente necessária para a análise dos transportadores de membrana são raras. Esta classe de transportadores, porém, é crucial na homeostase celular e, portanto, um alvo chave no desenvolvimento de medicamentos e metodologias para ganhar novos conhecimentos precisava desesperadamente.
O manuscrito apresentado aqui descreve a criação e gestão de um romance biochip para a análise dos processos de transporte de proteínas mediada membrana em resolução transporter única. O biochip é composto de microcavidades fechados por nanoporos, que é altamente paralelo na sua concepção e pode ser produzido em quantidade e de qualidade industrial. lipossomas proteína-abrigando pode ser directamente aplicada aa superfície do chip formar bicamadas lipídicas-medindo poros auto-montadas usando SSM-técnicas (sólido suportado membranas lipídicas). Pore-abrangendo partes da membrana são independentes, proporcionando a interface para a translocação de substrato para dentro ou para fora do espaço da cavidade, o que pode ser seguido por leitura fluorescente multi-espectral em tempo real. O estabelecimento de procedimentos operacionais padronizados (POPs) permite a criação simples de bicamadas lipídicas-abrigando proteína na superfície do chip de proteína de membrana praticamente todos os que podem ser reconstituídos funcionalmente. O único pré-requisito é o estabelecimento de um sistema de leitura de fluorescência para substratos de transporte não eletrogênica.
aplicações de alto teor de triagem são realizáveis pelo uso de automatizados microscópios fluorescentes invertidos gravação múltiplos chips em paralelo. Grandes conjuntos de dados podem ser analisados usando o software de análise de design personalizado disponível gratuitamente. Três cores de multi espectral fluorescenteleia-out, além disso, permite a discriminação de dados imparcial em diferentes classes de eventos, eliminando falsos resultados positivos.
A tecnologia de chip é atualmente baseado em SiO2 superfícies, mas ainda funcionalização usando superfícies de fichas revestidas de ouro também é possível.
Introduction
A análise das proteínas da membrana tornou-se de crescente interesse para a pesquisa básica e farmacêutica nos últimos 20 anos. O desenvolvimento de novos medicamentos depende da identificação e caracterização de novos alvos detalhada, actualmente ser um dos factores limitantes. O facto de que cerca de 60% de todos os alvos de drogas são proteínas de membrana 1, faz com que o desenvolvimento de técnicas para elucidar a sua função mais importante.
No passado, as técnicas para o estudo de canais electrogénico e transportadores têm sido desenvolvidos na multiplicidade 2-4. substratos não eletrogênica em contrário apresentar uma tarefa mais desafiadora. Eles são no entanto, de especial interesse como alvos de drogas de primeira linha, uma vez que controlam o fluxo de solutos e nutrientes através da membrana celular e como função de receptores principais em cascatas de sinalização 5.
Um esforço considerável foi colocado no desenvolvimento de techniques para estudar a função das proteínas transportadoras de membrana 6, 7. Os sistemas que utilizam membranas sólidas apoiado surgiram como ferramentas mais promissoras neste campo 8-10, incluindo bicamadas lipídicas suportados sólidos, bicamadas tethered 11, 12, membranas lipídicas microblack 13, 14 e nativas vesícula matrizes 15, 16 para citar alguns. Alguns deles estão ainda disponíveis como instalações comerciais 17, 18. Alguns exemplos foram publicados combinando a capacidade de estudar as proteínas de membrana individuais de uma maneira altamente paralela 14, 19, um pré-requisito para as aplicações de rastreio. No entanto, estes métodos raramente ponte de pesquisa básica ao ambiente industrial. As dificuldades muitas vezes encontram-se na capacidade do sistema para ser automatizável, a produção de custo elevado e / ou preparação laboriosa. Uma abordagem overcoming todos os obstáculos acima mencionados é o objetivo final.
A técnica aqui apresentada foi desenvolvido para o estudo de canais de membrana e transportadores in vitro num ambiente controlado no nível de proteína única 20-22. Reconstituição de proteínas de membrana purificadas em LUV é muito mais estabelecida do que as abordagens comparáveis para GUVs 23 – 26 ou lipídios preto membranas 27. Eles podem ser directamente aplicadas à superfície do chip, em que a formação de duas camadas tem lugar através de um processo de auto-montagem. O desenho com fundo de vidro nanoporoso do chip (Fig. 1) permite a microscopia de ar, que permite a automatização simples do sistema. Em combinação com uma platina motorizada múltiplos chips podem ser medidos ao mesmo tempo, com cada campo de visão contendo milhares de cavidades seladas para análise.
<p class="jove_content" fo:keep-together.within-page= "1">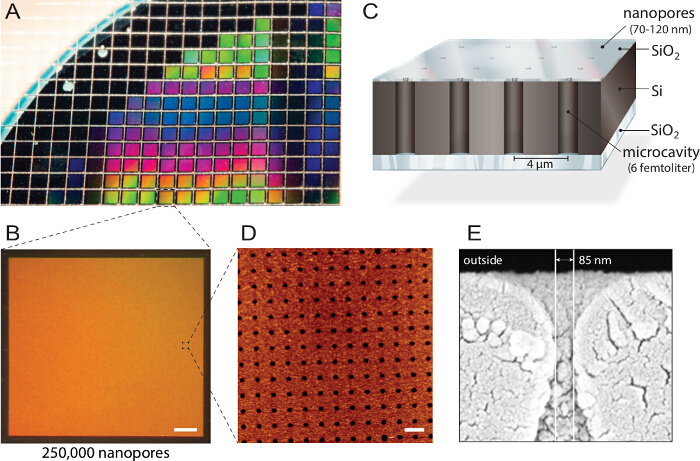
Figura 1. Projeto de biochips nanopore multiplexados. A) A silício sobre isolante (SOI) wafer é estruturado por ataque reativa-ion. Cerca de 1.150 fichas individuais são fabricados a partir de cada wafer com propriedades idênticas e qualidade. B) Cada chip inclui 250.000 microcavidades individuais com aberturas nano. Barra de escala:. 200 mm C) Cada cavidade é endereçável através de fluorescência multi-espectral de leitura. Um não transparentes superiores blocos da camada os sinais fluorescentes a partir do reservatório tampão, tornando o biochip compatível com microscópios de fluorescência invertido. D) microscopia de força atómica (AFM), imagiologia revela uniformemente dispostas aberturas dos poros e a rugosidade da superfície da camada de dióxido de silício de 3,6 nm (n = 40) ideal para a fusão das vesículas. Barra de escala:. 5 m E) mic eletrônica de varreduraroscopy imagem (SEM) mostra uma secção transversal através da nano-poros que permite o acesso às cavidades femtoliter dentro do chip de silício. Este valor foi reutilizado com permissão de 21. Por favor clique aqui para ver uma versão maior desta figura.
Toda a análise dos dados é feita usando freeware para garantir o acesso irrestrito para os usuários finais. séries temporais são analisados utilizando software de processamento de imagem gratuito e um processamento em lote que permite o software de análise personalizada curva de construção e correlação direta de grandes conjuntos de dados com múltiplos canais fluorescentes e milhares de curvas.
A proteína modelo utilizado neste protocolo é o canal mechanosensitive de proteína grande condutância (MsCl) canal derivado de E. coli. Ele funciona como uma válvula para libertar choque osmótico na natureza, mas foi modificado de tal maneira que racionalmente concebidos Synthetic funcionalidades pode ser covalentemente ligado ao lado de canais de constrição. Via-carga de repulsão do activador ligado covalentemente (MTSET) do canal é accionado para abrir, a criação de um nano-válvula. Pequenas moléculas como os íons, água, pequenas proteínas, mas também pequenas fluoróforos pode permear através do canal. Aqui, a proteína é utilizada como um modelo para demonstrar a capacidade do sistema para detectar a translocação mediada pela proteína.
Protocol
Representative Results
Discussion
A técnica apresentada aqui permite uma análise altamente paralela de transporte de proteínas de membrana. sistemas de proteína da membrana reconstituídas pode ser directamente aplicado ao biochip, fazer a adaptação de cada teoricamente transportador de membrana ou canalizar possível. análise de Transporte só é limitado pelo estabelecimento de um sistema de leitura de fluorescente, quer através de mudança direta de fluorescência (translocação de fluoróforos ou substratos marcados com fluorescência) ou a…
Disclosures
The authors have nothing to disclose.
Acknowledgements
We thank Barbara Windschiegl for her help in establishing SOPs; Dennis Remme for his work on the NanoCalcFX software and Alina Kollmannsperger, Markus Braner and Milan Gerovac for helpful suggestions on the manuscript. The German-Israeli Project Cooperation (DIP) provided by the DFG and the Federal Ministry of Education and Research to R.T., as well as the Federal Ministry of Economics and Technology (ZIM R&D Project) to R.T. and Nanospot GmbH supported this work.
Materials
| Reagent | |||
| [2-(Trimethylammonium)ethyl] methanethiosulfonate |
Toronto Research Chemicals Inc. | T792900 | MTSET; hydrolized by water. Keep as dry pouder aliquot at -80 °C. Use immediately (30 minutes) after solubilization in buffer. |
| 1 ml gas-tight syringe | Hamilton | #1001 | |
| 10 ml round flask | Schott Duran | ||
| 2.7 mm glas beads | Roth | N032.1 | |
| 2-Propanole | Roth | 9866.5 | |
| 30 cm Luer-Lock Extension Tube | Sarstedt | 744304 | |
| Acetone | Roth | 5025.5 | |
| Bio-Beads SM-2 Adsorbent | Bio-Rad | 152-3920 | need to be activated before first use |
| CaCl2 Dihydrat | Roth | HN04.3 | |
| Calcein | Sigma | C0875 | store dark at -20 °C |
| Chloroform reagent grade | VWR Chemicals | 22711324 | |
| DOPEATTO390 | ATTO-TEC | AD 390-165 | store dark at -20 °C |
| Ethanol absolute | Sigma-Aldrich | 32205 | |
| Injekt Single-use syringe | Braun | 460 60 51V | |
| Injekt-F single-use syringe | Braun | 91 66 017V | |
| Keck clips | Schott | KC29 | |
| L-α-Phosphatidylcholine, 20% (Soy) | Avanti Polar Lipids | 5416016 | store under inert gas at -20 °C |
| NaCl 99.5% p.a. | Roth | 3957.2 | |
| Nanopore E100 wafer/chips | Micromotive (Mainz/Germany) | available on request | |
| Nucleopore Track-Etch Membrane 0.4 µm | Whatman | 800282 | |
| Oregon Green Dextran 488 (70 kDa) | life Technologies | D-7173 | store dark at -20 °C |
| Oy647 | Luminartis (Münster/Germany) | OY-647-T-1mg | store dark at -20 °C |
| Rotilabo-syringe filtration, unsterile, pore-size 0.22 µm | Roth | P 818.1 | |
| Sephadex G-50 | Sigma-Aldrich | G5080 | column material for size exclusion chromatography |
| Silastic MDX4-4210 | Dow Corning | curing agent for chip fixation onto cover glass support | |
| sticky-Slide 8-well | ibidi | 80828 | multi-well chamber for the mounting onto glass slides (chip holder) |
| Three-way stopcock blue | Sarstedt | 744410001 | |
| Tris Pufferan 99.9% Ultra Quality | Roth | 5429.2 | |
| Triton-X 100 | Roth | 6683.1 | |
| Whatman 0.2 µm cellulose nitrate membrane filter | Roth | NH69.1 | |
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| Büchi 461 water bath | Büchi | ||
| Büchi Rotavapor RE 111 | Büchi | ||
| Cary Eclipse Fluorescence Spectrophtometer | Varian | ||
| LiposoFast Mini Extruder | Avestin | ||
| Membrane pump | Vaccubrand | 15430 | |
| Nanosight Nanoparticle Tracking Microscope | Malvern / Nanosight | LM 14C | |
| NyONE microscope | Synentec | available on request | |
| Pump control | Vaccubrand | CVC 2II | |
| Sonicator bath Sonorex RK100H | Brandelin electronic | 31200001107477 | |
| Vaccum pump RC5 | Vaccubrand | 1805400204 | |
| Water bath W13 | Haake | 002-9910 | |
| Plasma Cleaner PDC-37G | Harrick Plasma | PDC-37G | |
| Name | Company | Catalog Number | Comments |
| Software | |||
| ImageJ | Open Source | http://imagej.nih.gov/ij/ | scientific image processing software |
| NanoCalcFX | Freeware | http://sourceforge.net/projects/nanocalc/ | data analysis/evaluation software for massive transport kinetic datasets |
| NTA 2.3 Analytical Software | Nanosight | data acquisition and analysis software for nanoparticle tracking microscope | |
| NTA 2.3 Temperature Comms | Nanosight | temperature controle software for nanoparticle tracking microscope |
References
- Yildirim, M. A., Goh, K. I., Cusick, M. E., Barabasi, A. L., Vidal, M. Drug-target network. Nat Biotechnol. 25, 1119 (2007).
- Hamill, O. P., Marty, A., Neher, E., Sakmann, B., Sigworth, F. Improved patch-clamp techniques for high-resolution current recording from cells and cell-free membrane patches. Pflugers Arch. 391, 85-100 (1981).
- Osaki, T., Suzuki, H., Le Pioufle, B., Takeuchi, S. Multichannel simultaneous measurements of single-molecule translocation in α-hemolysin nanopore array. Anal. Chem. 81, 9866-9870 (2009).
- Carrillo, L., et al. High-resolution membrane capacitance measurements for studying endocytosis and exocytosis in yeast. Traffic. , (2015).
- Giacomini, K. M., et al. Membrane transporters in drug development. Nat. Rev. Drug Discov. 9, 215-236 (2010).
- Castell, O. K., Berridge, J., Wallace, M. I. Quantification of membrane protein inhibition by optical ion flux in a droplet interface bilayer array. Angew. Chem. Int. Ed. 51, 3134-3138 (2012).
- Zollmann, T., et al. Single liposome analysis of peptide translocation by the ABC transporter TAPL. Proc. Natl. Acad. Sci. U.S.A. 112, 2046-2051 (2015).
- Tamm, L. K., McConnell, H. M. Supported phospholipid bilayers. Biophys. J. 47, 105-113 (1985).
- Sackmann, E. Supported membranes: scientific and practical applications. Science. 271, 43-48 (1996).
- Castellana, E. T., Cremer, P. S. Solid supported lipid bilayers: From biophysical studies to sensor design. Surf. Sci. Rep. 61, 429-444 (2006).
- Wagner, M. L., Tamm, L. K. Tethered polymer-supported planar lipid bilayers for reconstitution of integral membrane proteins: silane-polyethyleneglycol-lipid as a cushion and covalent linker. Biophys. J. 79, 1400-1414 (2000).
- Naumann, C. A., et al. The polymer-supported phospholipid bilayer: tethering as a new approach to substrate-membrane stabilization. Biomacromolecules. 3, 27-35 (2002).
- Weiskopf, D., Schmitt, E. K., Klühr, M. H., Dertinger, S. K., Steinem, C. Micro-BLMs on highly ordered porous silicon substrates: Rupture process and lateral mobility. Langmuir. 23, 9134-9139 (2007).
- Watanabe, R., et al. Arrayed lipid bilayer chambers allow single-molecule analysis of membrane transporter activity. Nat. Commun. 5, (2014).
- Stamou, D., Duschl, C., Delamarche, E., Vogel, H. Self-Assembled Microarrays of Attoliter Molecular Vessels. Angew. Chem. Int. Ed. 115, 5738-5741 (2003).
- Lohr, C., et al. Single Liposomes Used to Study the Activity of Individual Transporters. Biophysical Journal. 106, 229a (2014).
- Dunlop, J., Bowlby, M., Peri, R., Vasilyev, D., Arias, R. High-throughput electrophysiology: an emerging paradigm for ion-channel screening and physiology. Nat. Rev. Drug Discov. 7, 358-368 (2008).
- Milligan, C. J., et al. Robotic multiwell planar patch-clamp for native and primary mammalian cells. Nat. Protoc. 4, 244-255 (2009).
- Soga, N., Watanabe, R., Noji, H. Attolitre-sized lipid bilayer chamber array for rapid detection of single transporters. Sci. Rep. 5, (2015).
- Kleefen, A., et al. Multiplexed parallel single transport recordings on nanopore arrays. Nano Lett. 10, 5080-5087 (2010).
- Wei, R., Gatterdam, V., Wieneke, R., Tampé, R., Rant, U. Stochastic sensing of proteins with receptor-modified solid-state nanopores. Nat. Nanotechnol. 7, 257-263 (2012).
- Urban, M., et al. Highly parallel transport recordings on a membrane-on-nanopore chip at single molecule resolution. Nano Lett. 14, 1674-1680 (2014).
- Kusters, I., Van Oijen, A. M., Driessen, A. J. Membrane-on-a-Chip: Microstructured Silicon/Silicon-Dioxide Chips for High-Throughput Screening of Membrane Transport and Viral Membrane Fusion. ACS Nano. 8, 3380-3392 (2014).
- Hansen, J. S., Thompson, J. R., Hélix-Nielsen, C., Malmstadt, N. Lipid directed intrinsic membrane protein segregation. J. Am. Chem. Soc. 135, 17294-17297 (2013).
- Heinemann, F., Schwille, P. Preparation of Micrometer-Sized Free-Standing Membranes. ChemPhysChem. 12, 2568-2571 (2011).
- Lazzara, T. D., Carnarius, C., Kocun, M., Janshoff, A., Steinem, C. Separating attoliter-sized compartments using fluid pore-spanning lipid bilayers. ACS nano. 5, 6935-6944 (2011).
- Winterhalter, M. Black lipid membranes. Curr. Opin. Colloid Interface Sci. 5, 250-255 (2000).
- Koçer, A., Walko, M., Feringa, B. L. Synthesis and utilization of reversible and irreversible light-activated nanovalves derived from the channel protein MscL. Nat. Protoc. 2, 1426-1437 (2007).

