Triagem para Funcional não-codificante variantes genéticas Usando eletroforética Mobilidade Mudança Assay (EMSA) e DNA afinidade Precipitation Assay (DAPA)
Summary
We present a strategic plan and protocol for identifying non-coding genetic variants affecting transcription factor (TF) DNA binding. A detailed experimental protocol is provided for electrophoretic mobility shift assay (EMSA) and DNA affinity precipitation assay (DAPA) analysis of genotype-dependent TF DNA binding.
Abstract
Population and family-based genetic studies typically result in the identification of genetic variants that are statistically associated with a clinical disease or phenotype. For many diseases and traits, most variants are non-coding, and are thus likely to act by impacting subtle, comparatively hard to predict mechanisms controlling gene expression. Here, we describe a general strategic approach to prioritize non-coding variants, and screen them for their function. This approach involves computational prioritization using functional genomic databases followed by experimental analysis of differential binding of transcription factors (TFs) to risk and non-risk alleles. For both electrophoretic mobility shift assay (EMSA) and DNA affinity precipitation assay (DAPA) analysis of genetic variants, a synthetic DNA oligonucleotide (oligo) is used to identify factors in the nuclear lysate of disease or phenotype-relevant cells. For EMSA, the oligonucleotides with or without bound nuclear factors (often TFs) are analyzed by non-denaturing electrophoresis on a tris-borate-EDTA (TBE) polyacrylamide gel. For DAPA, the oligonucleotides are bound to a magnetic column and the nuclear factors that specifically bind the DNA sequence are eluted and analyzed through mass spectrometry or with a reducing sodium dodecyl sulfate polyacrylamide gel electrophoresis (SDS-PAGE) followed by Western blot analysis. This general approach can be widely used to study the function of non-coding genetic variants associated with any disease, trait, or phenotype.
Introduction
Sequenciação e estudos de genotipagem baseados, incluindo Estudos do Genoma-Wide Association (GWAS), estudos locus candidato, e deep-sequenciação estudos, identificamos muitas variantes genéticas que estão estatisticamente associados com uma doença, característica ou fenótipo. Ao contrário do que as previsões iniciais, a maior parte destas variantes (85-93%) estão localizados em regiões não codificantes e não alteram a sequência de aminoácidos de proteínas de 1,2. Interpretando a função destas variantes não-codificantes e determinar os mecanismos biológicos que ligam-los para a doença associada, traço, ou fenótipo provou desafiador 3-6. Nós desenvolvemos uma estratégia geral para identificar os mecanismos moleculares que ligam variantes para um importante intermediário fenótipo – a expressão do gene. Esse gasoduto é projetado especificamente para identificar a modulação da ligação de variantes genéticas TF. Esta estratégia combina abordagens computacionais e técnicas de biologia molecular voltadas para preverefeitos biológicos das variantes candidatos in silico e verifique estas previsões empiricamente (Figura 1).
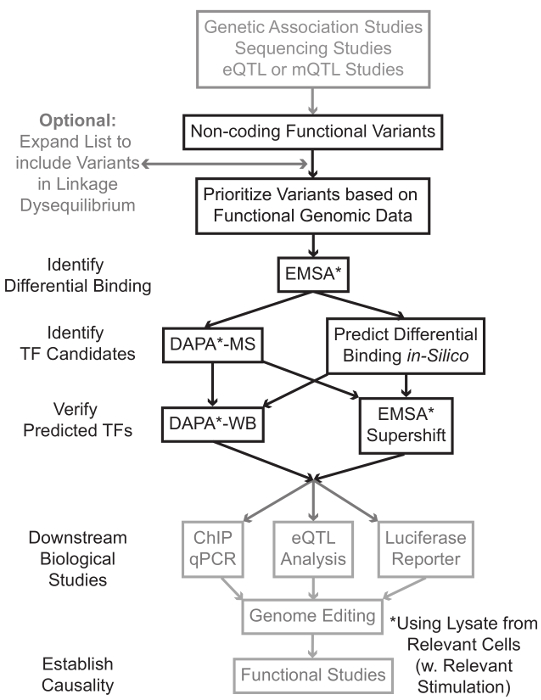
Figura 1:.. Uma abordagem estratégica para a análise de Passos não-codificantes variantes genéticas que não estão incluídos no protocolo detalhado associado a este manuscrito estão com fundo cinza Por favor clique aqui para ver uma versão maior desta figura.
Em muitos casos, é importante começar pela expansão da lista de variantes para incluir todos aqueles em alta linkage-desequilíbrio (LD), com cada variante estatisticamente associada. LD é uma medida da associação não aleatória de alelos em duas posições cromossómica diferente, o que pode ser medido pela estatística de R 2 7. R2 é uma medida da linkage desequilíbrio entre duas variantes, com um r 2 = 1 denotando ligação perfeita entre duas variantes. Os alelos em alta LD são encontrados a co-segregar no cromossoma através de populações ancestrais. matrizes de genotipagem circulante não inclui todas as variantes conhecidas do genoma humano. Em vez disso, eles explorar o LD dentro do genoma humano e incluem um subconjunto das variantes conhecidas que funcionam como proxies para outras variantes dentro de uma região particular de LD 8. Assim, uma variante sem qualquer consequência biológica pode estar associada com uma doença em particular, porque é em LD com a variante a-causal variante com um efeito biológico significativo. Processualmente, recomenda-se para converter a versão mais recente dos 1.000 genomas projetar 9 arquivos de chamada variante (VCF) em arquivos binários compatíveis com Plink 10,11, uma ferramenta de código aberto para análise de associação do genoma inteiro. Posteriormente, todas as outras variantes genéticas com LD r 2> 0,8 com cada va genética de entradaRiant podem ser identificados como candidatos. É importante usar a população de referência apropriado para este passo-por exemplo, se uma variante foi identificada em indivíduos de ascendência europeia, dados de indivíduos de ascendência similar deve ser usado para a expansão LD.
LD expansão muitas vezes resulta em dezenas de variantes de candidatos, e é provável que apenas uma pequena fracção delas contribuir para mecanismo da doença. Muitas vezes, isto não é exequível para examinar experimentalmente cada uma destas variantes individualmente. Portanto, é útil para alavancar os milhares de conjuntos de dados genômicos funcionais publicamente disponíveis como um filtro para priorizar as variantes. Por exemplo, o consórcio CODIFICAR 12 realizou milhares de experiências Chip-seguintes que descrevem a ligação de TF e co-factores, e marcas de histona numa vasta variedade de contextos, juntamente com os dados de acessibilidade cromatina de tecnologias tais como a DNase-SEQ 13, ATAC -seq 14 e FAIRE-seq 15. databases e servidores web, tais como o navegador UCSC Genome 16, Roteiro Epigenomics 17, Blueprint Epigenome 18, Cistrome 19, e remapear 20 permitem o acesso gratuito aos dados produzidos por essas e outras técnicas experimentais através de uma ampla gama de tipos e condições celulares. Quando existem muitas variantes para examinar experimentalmente, estes dados podem ser utilizadas para hierarquizar os localizados dentro das regiões reguladoras prováveis em tipos de células e de tecidos relevantes. Além disso, nos casos em que uma variante está dentro de um pico ChIP-seq para uma proteína específica, esses dados podem fornecer potenciais clientes em potencial quanto à TF específico (s) ou co-fatores cuja ligação pode estar afetando.
Em seguida, as variantes resultantes priorizadas são testadas experimentalmente para validar proteína dependente do genótipo previu ligação usando EMSA 21,22. EMSA mede a mudança na migração do oligo num gel de TBE não redutoras. oligo marcado com fluorescência é incubada com olisado nuclear, e ligação de factores nucleares irá retardar o movimento do oligo no gel. Desta maneira, oligo que se tenha ligado mais factores nucleares vai apresentar-se como um sinal de fluorescência mais forte sobre a digitalização. Notavelmente, a EMSA não requer previsões sobre as proteínas específicas cuja ligação será afetado.
Uma vez que as variantes são identificados que estão localizados dentro das regiões reguladoras previstos e são capazes de factores nucleares de ligação diferencialmente, métodos computacionais são utilizados para prever o TF específico (s) cuja ligação que possa afectar. Nós preferimos usar CIS-BP 23,24, RegulomeDB 25, UNIProbe 26, e Jaspar 27. Uma vez candidato TFs são identificados, essas previsões podem ser testados especificamente utilizando anticorpos contra esses TFs (EMSA-supershifts e DAPA-Westerns). Um EMSA-supershift envolve a adição de um anticorpo específico para o TF ao lisado nuclear e oligo. Um resultado positivo em um EMSA-supershift é represented como mais um desvio na banda de EMSA, ou uma perda da banda (revisto em referência 28). No DAPA complementar, uma cadeia dupla oligo 5'-biotinilado contendo a variante e a 20 pares de bases que flanqueia nucleótidos são incubadas com lisado nuclear do tipo de célula relevante (s) para capturar quaisquer factores nucleares que se ligam especificamente os oligos. O complexo do factor nuclear-duplex oligonucleótido é imobilizado por estreptavidina micropérolas em uma coluna magnética. Os fatores nucleares ligados são recolhidos directamente através de eluição 29,48. previsões de ligação pode então ser avaliada por uma transferência de Western utilizando anticorpos específicos para a proteína. Nos casos em que não existem previsões óbvias, ou muitas previsões, as eluições de variantes menus pendentes dos experimentos DAPA podem ser enviadas para um núcleo de proteômica para identificar TFs candidatos usando espectrometria de massa, que podem posteriormente ser validados usando estes previamente descrito métodos.
No restante do articlE, o protocolo detalhado para análise EMSA e DAPA de variantes genéticas é fornecido.
Protocol
Representative Results
Discussion
Apesar de avanços em tecnologias de sequenciamento e genotipagem tem bastante reforçada a nossa capacidade para identificar variantes genéticas associadas à doença, a nossa capacidade de compreender os mecanismos funcionais impactados por estas variantes está atrasado. Uma das principais fontes do problema é que muitas variantes associados à doença estão localizados em n-codificante em regiões do genoma, que provavelmente afectam mais difícil de prever mecanismos que controlam a expressão do gene. Aqu…
Disclosures
The authors have nothing to disclose.
Acknowledgements
We thank Erin Zoller, Jessica Bene, and Lindsey Hays for input and direction in protocol development. MTW was supported in part by NIH R21 HG008186 and a Trustee Award grant from the Cincinnati Children’s Hospital Research Foundation. ZHP was supported in part by T32 GM063483-13.
Materials
| Custom DNA Oligonucleotides | Integrated DNA Technologies | http://www.idtdna.com/site/order/oligoentry | |
| Potassium Chloride | Fisher Scientific | BP366-500 | KCl, for CE buffer |
| HEPES (1M) | Fisher Scientific | 15630-080 | For CE and NE buffer |
| EDTA (0.5M), pH 8.0 | Life Technologies | R1021 | For CE, NE, and annealing buffer |
| Sodium Chloride | Fisher Scientific | BP358-1 | NaCl, for NE buffer |
| Tris-HCl (1M), pH 8.0 | Invitrogen | BP1756-100 | For annealing buffer |
| Phosphate Buffered Saline (1X) | Fisher Scientific | MT21040CM | PBS, for cell wash |
| DL-Dithiothreitol solution (1M) | Sigma | 646563 | Reducing agent |
| PMSF | Thermo Scientific | 36978 | Protease Inhibitor |
| Phosphatase Inhibitor Cocktail | Thermo Scientific | 78420 | Prevents dephosphorylation of TFs |
| Nonidet P-40 Substitute | IBI Scientific | IB01140 | NP-40, for nuclear extraction |
| BCA Protein Assay Kit | Thermo Scientific | 23225 | For measuring protein concentration |
| Odyssey EMSA Buffer Kit | Licor | 829-07910 | Contains all necessary EMSA buffers |
| TBE Gels, 6%, 12 Wells | Invitrogen | EC6265BOX | For EMSA |
| TBE Buffer (10X) | Thermo Scientific | B52 | For EMSA |
| FactorFinder Starting Kit | Miltenyi Biotec | 130-092-318 | Contains all necessary DAPA buffers |
| Licor Odyssey CLx | Licor | Recommended scanner for DAPA/EMSA | |
| Antibiotic-Antimycotic | Gibco | 15240-062 | Contains 10,000 units/mL of penicillin, 10,000 µg/mL of streptomycin, and 25 µg/mL of Fungizone® Antimycotic |
| Fetal Bovine Serum | Gibco | 26140-079 | FBS, for culture media |
| RPMI 1640 Medium | Gibco | 22400-071 | Contains L-glutamine and 25mM HEPES |
References
- Hindorff, L. A., et al. Potential etiologic and functional implications of genome-wide association loci for human diseases and traits. Proc Natl Acad Sci U S A. 106 (23), 9362-9367 (2009).
- Maurano, M. T., et al. Systematic localization of common disease-associated variation in regulatory DNA. Science. 337 (6099), 1190-1195 (2012).
- Ward, L. D., Kellis, M. Interpreting noncoding genetic variation in complex traits and human disease. Nat Biotechnol. 30 (11), 1095-1106 (2012).
- Paul, D. S., Soranzo, N., Beck, S. Functional interpretation of non-coding sequence variation: concepts and challenges. Bioessays. 36 (2), 191-199 (2014).
- Zhang, F., Lupski, J. R. Non-coding genetic variants in human disease. Hum Mol Genet. , (2015).
- Lee, T. I., Young, R. A. Transcriptional regulation and its misregulation in disease. Cell. 152 (6), 1237-1251 (2013).
- Slatkin, M. Linkage disequilibrium–understanding the evolutionary past and mapping the medical future. Nat Rev Genet. 9 (6), 477-485 (2008).
- Bush, W. S., Moore, J. H. Chapter 11: Genome-wide association studies. PLoS Comput Biol. 8 (12), e1002822 (2012).
- 1000 Genomes Project Consortium. An integrated map of genetic variation from 1,092 human genomes. Nature. 491 (7422), 56-65 (2012).
- Chang, C. C., et al. Second-generation PLINK: rising to the challenge of larger and richer datasets. Gigascience. 4, 7 (2015).
- Purcell, S., et al. PLINK: a tool set for whole-genome association and population-based linkage analyses. Am J Hum Genet. 81 (3), 559-575 (2007).
- ENCODE Project Consortium. An integrated encyclopedia of DNA elements in the human genome. Nature. 489 (7414), 57-74 (2012).
- Crawford, G. E., et al. Genome-wide mapping of DNase hypersensitive sites using massively parallel signature sequencing (MPSS). Genome Res. 16 (1), 123-131 (2006).
- Buenrostro, J. D., Giresi, P. G., Zaba, L. C., Chang, H. Y., Greenleaf, W. J. Transposition of native chromatin for fast and sensitive epigenomic profiling of open chromatin, DNA-binding proteins and nucleosome position. Nat Methods. 10 (12), 1213-1218 (2013).
- Giresi, P. G., Kim, J., McDaniell, R. M., Iyer, V. R., Lieb, J. D. FAIRE Formaldehyde-Assisted Isolation of Regulatory Elements) isolates active regulatory elements from human chromatin. Genome Res. 17 (6), 877-885 (2007).
- Kent, W. J., et al. The human genome browser at UCSC. Genome Res. 12 (6), 996-1006 (2002).
- Roadmap Epigenomics Consortium. Integrative analysis of 111 reference human epigenomes. Nature. 518 (7539), 317-330 (2015).
- Martens, J. H., Stunnenberg, H. G. BLUEPRINT: mapping human blood cell epigenomes. Haematologica. 98 (10), 1487-1489 (2013).
- Liu, T., et al. Cistrome: an integrative platform for transcriptional regulation studies. Genome Biol. 12 (8), R83 (2011).
- Griffon, A., et al. Integrative analysis of public ChIP-seq experiments reveals a complex multi-cell regulatory landscape. Nucleic Acids Res. 43 (4), e27 (2015).
- Staudt, L. M., et al. A lymphoid-specific protein binding to the octamer motif of immunoglobulin genes. Nature. 323 (6089), 640-643 (1986).
- Singh, H., Sen, R., Baltimore, D., Sharp, P. A. A nuclear factor that binds to a conserved sequence motif in transcriptional control elements of immunoglobulin genes. Nature. 319 (6049), 154-158 (1986).
- Weirauch, M. T., et al. Determination and inference of eukaryotic transcription factor sequence specificity. Cell. 158 (6), 1431-1443 (2014).
- Ward, L. D., Kellis, M. HaploReg: a resource for exploring chromatin states, conservation, and regulatory motif alterations within sets of genetically linked variants. Nucleic Acids Res. 40 (Database issue), D930-D934 (2012).
- Boyle, A. P., et al. Annotation of functional variation in personal genomes using RegulomeDB. Genome Res. 22 (9), 1790-1797 (2012).
- Hume, M. A., Barrera, L. A., Gisselbrecht, S. S., Bulyk, M. L. UniPROBE, update 2015: new tools and content for the online database of protein-binding microarray data on protein-DNA interactions. Nucleic Acids Res. 43 (Database issue), D117-D122 (2015).
- Mathelier, A., et al. JASPAR 2014: an extensively expanded and updated open-access database of transcription factor binding profiles. Nucleic Acids Res. 42 (Database issue), 142-147 (2014).
- Smith, M. F., Delbary-Gossart, S. Electrophoretic Mobility Shift Assay (EMSA). Methods Mol Med. 50, 249-257 (2001).
- Franza, B. R., Josephs, S. F., Gilman, M. Z., Ryan, W., Clarkson, B. Characterization of cellular proteins recognizing the HIV enhancer using a microscale DNA-affinity precipitation assay. Nature. 330 (6146), 391-395 (1987).
- . BCA Protein Assay Kit: User Guide Available from: https://tools.thermofisher.com/content/sfs/manuals/MAN0011430_Pierce_BCA_Protein_Asy_UG.pdf (2014)
- Wijeratne, A. B., et al. Phosphopeptide separation using radially aligned titania nanotubes on titanium wire. ACS Appl Mater Interfaces. 7 (21), 11155-11164 (2015).
- Silva, J. M., McMahon, M. The Fastest Western in Town: A Contemporary Twist on the Classic Western Blot Analysis. J. Vis. Exp. (84), (2014).
- Lu, X., et al. Lupus Risk Variant Increases pSTAT1 Binding and Decreases ETS1 Expression. Am J Hum Genet. 96 (5), 731-739 (2015).
- Ramana, C. V., Chatterjee-Kishore, M., Nguyen, H., Stark, G. R. Complex roles of Stat1 in regulating gene expression. Oncogene. 19 (21), 2619-2627 (2000).
- Fillebeen, C., Wilkinson, N., Pantopoulos, K. Electrophoretic Mobility Shift Assay (EMSA) for the Study of RNA-Protein Interactions: The IRE/IRP Example. J. Vis. Exp. (94), e52230 (2014).
- Heng, T. S., Painter, M. W. Immunological Genome Project, C. The Immunological Genome Project: networks of gene expression in immune cells. Nat Immunol. 9 (10), 1091-1094 (2008).
- Wu, C., et al. BioGPS: an extensible and customizable portal for querying and organizing gene annotation resources. Genome Biol. 10 (11), R130 (2009).
- Wu, C., Macleod, I., Su, A. I. BioGPS and MyGene.info: organizing online, gene-centric information. Nucleic Acids Res. 41 (Database issue), D561-D565 (2013).
- Wang, J., et al. Sequence features and chromatin structure around the genomic regions bound by 119 human transcription factors. Genome Res. 22 (9), 1798-1812 (2012).
- Holden, N. S., Tacon, C. E. Principles and problems of the electrophoretic mobility shift assay. J Pharmacol Toxicol Methods. 63 (1), 7-14 (2011).
- Xu, J., Liu, H., Park, J. S., Lan, Y., Jiang, R. Osr1 acts downstream of and interacts synergistically with Six2 to maintain nephron progenitor cells during kidney organogenesis. Development. 141 (7), 1442-1452 (2014).
- Yang, T. -. P., et al. Genevar: a database and Java application for the analysis and visualization of SNP-gene associations in eQTL studies. Bioinformatics. 26 (19), 2474-2476 (2010).
- Fort, A., et al. A liver enhancer in the fibrinogen gene cluster. Blood. 117 (1), 276-282 (2011).
- Solberg, N., Krauss, S. Luciferase assay to study the activity of a cloned promoter DNA fragment. Methods Mol Biol. 977, 65-78 (2013).
- Rahman, M., et al. A repressor element in the 5′-untranslated region of human Pax5 exon 1A. Gene. 263 (1-2), 59-66 (2001).
- Mali, P., et al. RNA-Guided Human Genome Engineering via Cas9. Science. 339 (6121), 823-826 (2013).

