Eksperimentel Design for Laser mikrodissektions RNA-Seq: Erfaringer fra en analyse af Maize Leaf Development
Summary
Many developmentally important genes have cell- or tissue-specific expression patterns. This paper describes LM RNA-seq experiments to identify genes that are differentially expressed at the maize leaf blade-sheath boundary and in lg1-R mutants compared to wild-type. The experimental considerations discussed here apply to transcriptomic analyses of other developmental phenomena.
Abstract
Gener med vigtige roller i udvikling har ofte rumligt og / eller tidsmæssigt begrænsede ekspressionsmønstre. Ofte er disse gentranskripter bliver ikke fundet eller er ikke identificeret som differentielt udtrykt (DE) i transkriptomisk analyser af hele planten organer. Laser mikrodissektions RNA-Seq (LM-RNA-Seq) er et kraftfuldt værktøj til at identificere gener, der er DE i bestemte udviklingsmæssige domæner. Men valget af cellulære domæner microdissect og sammenligne, og nøjagtigheden af de microdissections er afgørende for succes i forsøgene. Her to eksempler illustrerer design overvejelser for transcriptomics eksperimenter; en LM RNA-seq analyse for at identificere gener, der er DE langs majs blad proximal-distal akse, og et andet eksperiment for at identificere gener, der er DE i liguleless1-R (LG1-R) mutanter sammenlignet med vildtype. Centrale elementer, som bidrog til succes for disse eksperimenter blev detaljeret histologiske og i situ-hybridisering analyser af regionen, der skal analyseres, udvælgelse af blad primordier ved ækvivalente udviklingsstadier, anvendelse af morfologiske landmærker at vælge regioner for mikrodissektion, og mikrodissektion af netop målte domæner. Dette dokument indeholder en detaljeret protokol til analyse af udviklingsmæssige domæner ved LM RNA-Seq. De data, der præsenteres her illustrerer, hvordan den er valgt til mikrodissektion region vil påvirke de opnåede resultater.
Introduction
Majs blad er en ideel model til at studere dannelsen af udviklingsmæssige felter under morfogenese, da det har en tydelig grænse mellem bladet og hylsteret, der er modtagelig for genetisk dissektion (figur 1A). I de tidlige stadier af blad udvikling, en lineær bånd af mindre celler, den preligule bånd (PLB), opdeler bladet primordium i præ-bladet og præ-kappe domæner. En frynse-lignende Skedehinden og trekantede bladflige udvikler fra PLB (figur 1A, C, D). Genetiske skærme har identificeret mutationer, der forstyrrer blad-kappe grænsen. For eksempel recessiv liguleless1 (LG1) mutationer slette Skedehinden og bladflige 1, 2, 3, 4 (figur 1 B). In situ hybridisering afslørede, at LG1 transkript akkumuleres på PLB og nye Skedehinden, hvilket gør den til en fremragende markør for Skedehinden udvikling 5, 6 (figur 1E).
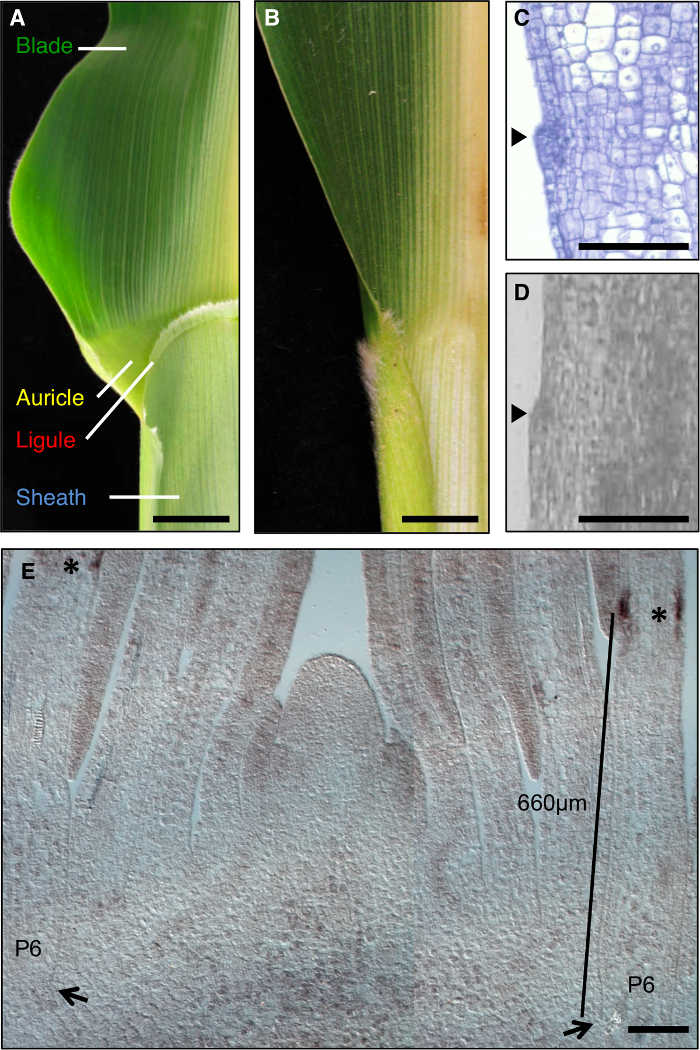
Figur 1: Vildtype og liguleless1-R majsblade. (A) Blade-kappe grænseområde af modent blad vildtype viser Skedehinden og øremuslingen strukturer. (B) Blade-kappe grænseområde af modent liguleless1-R blad viser fravær af skedehinde og øremuslingen strukturer. Blade i A og B er blevet skåret i halve langs midtribbe. (C) Længdesnit gennem vildtype blad primordium. Prøve er blevet behandlet og farvet for histologisk analyse. Den initierende Skedehinden fremgår som en bule rager frem fra planet af bladet (pilespids). (D) Langsgående sektion gennem vildtype blad primordium. Prøve er blevet forarbejdet til LM som beskrevet i teksten. Arrowhead indikerer indlede Skedehinden. (E) LG1 in situ hybridisering af skudspidsen laterale langsgående sektion. Stjerner angiver LG1 transkript akkumuleres mindst PLB af P6 blad primordium. Pile angiver base af P6 primordium. Bar indikerer måling fra bunden af primordium til PLB. Scale barer i A og B = 20 mm. Scale barer i CE = 100 um. Dette tal har været ændret siden henvisning 6 (Copyright American Society of Plant Biologer). Klik her for at se en større version af dette tal.
I denne undersøgelse blev LM RNA-Seq anvendes til at identificere en suite af gener, som udtrykkes forskelligt (DE) på blad-skede grænse i forhold til andre dele af bladet primordium og til IDE ntify gener, som er DE i LG1-R-mutanter i forhold til vildtype-søskende. LM RNA-Seq er en fremgangsmåde til kvantificering transkript akkumulering i specifikke celler eller cellulære domæner 7. LM systemer kombinerer en laser og et mikroskop med et digitalt kamera. Sektioneret væv er monteret på objektglas og set gennem mikroskopet. LM software omfatter typisk tegning værktøjer, der giver brugeren mulighed for at skitsere nogen valgte område for mikrodissektion. De laser skærer langs linjen, og den valgte væv slynget væk fra dias og ind i et rør ophængt over dias. LM tillader brugeren at microdissect præcise domæner, herunder specifikke cellelag og endda enkeltceller 8, 9. RNA kan derefter ekstraheres fra mikrodissekeret væv. Efterfølgende RNA-Seq komponent anvender næste generations sekventering til sekventering cDNA-biblioteker genereret fra det ekstraherede RNA 10,= "xref"> 11.
Vigtige fordele ved LM RNA-seq er evnen til at kvantificere transkript akkumulering i præcist definerede domæner og kapaciteten til at profilere hele transkriptom samtidigt 7. Teknikken er særligt velegnet til sondering tidlige udviklingsmæssige begivenheder, hvor området af interesse er ofte mikroskopisk. Tidligere undersøgelser har anvendt LM kombineret med microarray teknologi til at studere udviklingsprocesser i planter 9, 12, 13. RNA-Seq har den fordel, at kvantificere transkripter over et bredt dynamisk område, herunder lav-udtrykte gener, og forud sekvensinformation er ikke påkrævet 10, 11. Desuden LM RNA-Seq har potentiale til at fremhæve udviklingsmæssigt vigtige gener, der kan være savnet i mutagenese-skærme skyldes genetiske redundans eller for dødelighed af tabet-of-funktion mutant.
Udviklingsmæssigt vigtige gener, såsom snævre sheath1 (NS1) og skålformet cotyledon2 (CUC2), har ofte specifikke ekspressionsmønstre af blot én eller nogle få celler 17, 18, 19, 20. Mange udtrykkes kun i de tidlige udviklingsstadier og ikke i det modne organ. Når hele organer eller store domæner analyseres, er disse celle-specifikke udskrifter fortyndet og kan ikke påvises i mere traditionelle analyser. Ved at tillade analyser af præcist definerede domæner, LM-RNA-Seq muliggør disse vævsspecifikke gener kan identificeres og kvantificeres.
Afgørende faktorer i succes eksperimenterne beskrevet her var en grundig histologisk analyse, der guidede Valget af den passende udviklingsstadiet og domæne til analyse, og præcis overvånt af celle-væv domæner for LM. For at sikre, at tilsvarende domæner blev udtaget for alle replikater, blev væv opsamlet fra blad primordier på samme udviklingstrin og Mikrodissekterede domæner blev målt i forhold til morfologiske vartegn såsom den nye Skedehinden (figur 2). Det er kendt, at nogle gener udtrykkes i en gradient fra spidsen til bunden af bladet. Ved at måle præcise domæner, variation som følge af prøvetagning fra forskellige steder langs bladet proksimale-distal akse blev holdt på et minimum (figur 3A). Ved microdissecting domæner af samme størrelse, at variation som følge differentiel fortynding af cellespecifikke transkripter blev også reduceret (figur 3B). Laterale langsgående sektioner af skudspidsen blev anvendt til alle microdissections. Disse er sektioner, der er vinkelret på midtribbe-margin akse (figur 4). Brug kun dele, der omfatter SAM sikrer, at tilsvarende laterale regionerleaf primordier analyseres.
I prøver behandles og sektioneret for LM, den første morfologiske tegn på Skedehinden udvækst er et bump på adaxial side grundet periclinal celledelinger i adaxial epidermis (Figur 1D, figur 2). Det blev fastslået, at den spirende Skedehinden kunne identificeres pålideligt ved plastochron 7 trin blad primordier. Vi var interesserede i gener udtrykt i hele Skedehinden regionen, herunder den nye Skedehinden og cellerne umiddelbart distale, der vil danne ydre øre. For at sikre, at ækvivalente væv markeringer blev foretaget, blev Skedehinden bump anvendes som en morfologisk milepæl og et 100 um rektangel centreret på Skedehinden bump blev udvalgt til LM (figur 2A, 2B). Ækvivalente mellemstore rektangler af pre-bladet og præ-skede blev udvalgt fra de samme blade primordier.
Analyser af liguleless mutantplanter præsenteret en anden ChalleNSÆ; LG1-R-mutanter ikke danner en Skedehinden, derfor denne morfologiske træk kunne ikke anvendes til at vælge det område for LM. I stedet blev domænet for LG1 transkript akkumulering i vildtype blad primordier bestemt, og en region, der skulle omfatte dette domæne blev defineret. Disse foreløbige analyser blev udført på frøplanter fra samme plantning som blev anvendt ved den endelige analyse, da tidligere arbejde har vist, at placeringen af PLB varierer afhængigt af vækstbetingelserne. In situ hybridisering er angivet, at LG1 transkripter ophobes i PLB af P6 blad primordier (figur 1E). Vi valgte et domæne 400-900 um fra bunden af bladet primordier der omfattede domænet af LG1 ekspression (lilla rektangler, figur 2A) og fanget disse ækvivalente regioner fra vildtype og LG1-R planter. For at minimere variation i genetiske baggrund og vækstbetingelser, når man sammenligner transcript akkumulering i LG1-R og vildtype-planter, segregerende familier af mutanter og vildtype-søskende blev anvendt.
Protocol
Representative Results
Discussion
Eksperimentel design er en kritisk faktor i RNA-seq eksperimenter. Vigtige hensyn er den præcise domæne (r) og udviklingsstadiet (er), der skal analyseres, og hvilke sammenligninger vil blive foretaget. Det er afgørende at tænke i sammenligninger, da produktionen er typisk en liste af gener, der er DE mellem to eller flere betingelser. Som med alle eksperimenter, er det vigtigt at ændre kun én variabel ad gangen. For eksempel, når man sammenligner forskellige blade domæner, blade af samme alder og udviklingsstad…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Forfatterne takker S. Hake for løbende samarbejde og stimulerende diskussioner om Skedehinden udvikling. Dette arbejde er støttet af National Science Foundation Grants MCB 1052051 og IOS-1.848.478.
Materials
| Diethyl pyrocarbonate | Sigma-Aldrich | 159220 | Used for RNase treatment of solutions |
| Razor blades | Electron Microscopy Sciences | 72000 | |
| RNase Zap | Sigma-Aldrich | R2020-250ML | RNase decontamination solution |
| Ethanol absolute 200 proof | Fisher Scientific | BP28184 | |
| Acetic acid, glacial | Sigma-Aldrich | A6283 | |
| Glass vials – 22mL | VWR | 470206-384 | |
| Xylenes, histological grade | Sigma-Aldrich | 534056-4L | |
| Paraplast plus | Sigma-Aldrich | P3683-1KG | |
| Disposable base molds, 15 x 15 x 5mm | VWR | 15154-072 | |
| Embedding rings | VWR | 15154-303 | |
| Silica gel packets | Electron Microscopy Sciences | 71206-01 | Desiccant for storage of paraffin blocks |
| Oven | Fisher Scientific | 15-103-0503 | Oven must maintain temperature of 60° C |
| Paraffin embedding station | Leica | EG1160 | |
| Microtome | Leica | RM2235 | |
| Slide warmer | Electron Microscopy Sciences | 71317-10 | |
| Coplin jars | Electron Microscopy Sciences | 70316-02 | |
| Laser microdissector | Zeiss | ||
| KIMWIPES™ Delicate Task Wipers | Kimberly-Clark Professional | 34120 | Lint-free wipes for wicking excess solutions from microscope slides |
| Membrane Slide 1.0 PEN | Zeiss | 415190-9041-000 | Slides for laser microdissection |
| Adhesive Cap 200 opaque | Zeiss | 415190-9181-000 | Tubes for laser microdissection |
| PicoPure RNA Isolation Kit | ThermoFisher Scientific | KIT0204 |
References
- Becraft, P. W., Bongard-Pierce, D. K., Sylvester, A. W., Poethig, R. S., Freeling, M. The liguleless-1 gene acts tissue specifically in maize leaf development. Dev Biol. 141 (1), 220-232 (1990).
- Sylvester, A. W., Cande, W. Z., Freeling, M. Division and differentiation during normal and liguleless-1 maize leaf development. Development. 110 (3), 985-1000 (1990).
- Moreno, M. A., Harper, L. C., Krueger, R. W., Dellaporta, S. L., Freeling, M. liguleless1 encodes a nuclear-localized protein required for induction of ligules and auricles during maize leaf organogenesis. Genes Dev. 11 (5), 616-628 (1997).
- Emerson, R. A. The inheritance of the ligule and auricle of corn leaves. Neb. Agr. Exp. Sta. An. Rep. 25, 81-85 (1912).
- Moon, J., Candela, H., Hake, S. The Liguleless narrow mutation affects proximal-distal signaling and leaf growth. Development. 140 (2), 405-412 (2013).
- Johnston, R., et al. Transcriptomic Analyses Indicate That Maize Ligule Development Recapitulates Gene Expression Patterns That Occur during Lateral Organ Initiation. Plant Cell. 26 (12), 4718-4732 (2014).
- Schmid, M. W., et al. A powerful method for transcriptional profiling of specific cell types in eukaryotes: laser-assisted microdissection and RNA sequencing. PLoS One. 7 (1), e29685 (2012).
- Kerk, N. M., Ceserani, T., Tausta, S. L., Sussex, I. M., Nelson, T. M. Laser capture microdissection of cells from plant tissues. Plant Physiol. 132 (1), 27-35 (2003).
- Nakazono, M., Qiu, F., Borsuk, L. A., Schnable, P. S. Laser-capture microdissection, a tool for the global analysis of gene expression in specific plant cell types: identification of genes expressed differentially in epidermal cells or vascular tissues of maize. Plant Cell. 15 (3), 583-596 (2003).
- Marioni, J. C., Mason, C. E., Mane, S. M., Stephens, M., Gilad, Y. RNA-seq: an assessment of technical reproducibility and comparison with gene expression arrays. Genome Res. 18 (9), 1509-1517 (2008).
- Wang, Z., Gerstein, M., Snyder, M. RNA-Seq: a revolutionary tool for transcriptomics. Nat Rev Genet. 10 (1), 57-63 (2009).
- Brooks, L., et al. Microdissection of shoot meristem functional domains. PLoS Genet. 5 (5), e1000476 (2009).
- Cai, S., Lashbrook, C. C. Stamen abscission zone transcriptome profiling reveals new candidates for abscission control: enhanced retention of floral organs in transgenic plants overexpressing Arabidopsis ZINC FINGER PROTEIN2. Plant Physiol. 146 (3), 1305-1321 (2008).
- Li, P., et al. The developmental dynamics of the maize leaf transcriptome. Nat Genet. 42 (12), 1060-1067 (2010).
- Eveland, A. L., et al. Regulatory modules controlling maize inflorescence architecture. Genome Res. 24 (3), 431-443 (2014).
- Takacs, E. M., et al. Ontogeny of the maize shoot apical meristem. Plant Cell. 24 (8), 3219-3234 (2012).
- Aida, M., Ishida, T., Tasaka, M. Shoot apical meristem and cotyledon formation during Arabidopsis embryogenesis: interaction among the CUP-SHAPED COTYLEDON and SHOOT MERISTEMLESS genes. Development. 126 (8), 1563-1570 (1999).
- Ishida, T., Aida, M., Takada, S., Tasaka, M. Involvement of CUP-SHAPED COTYLEDON genes in gynoecium and ovule development in Arabidopsis thaliana. Plant Cell Physiol. 41 (1), 60-67 (2000).
- Takada, S., Hibara, K., Ishida, T., Tasaka, M. The CUP-SHAPED COTYLEDON1 gene of Arabidopsis regulates shoot apical meristem formation. Development. 128 (7), 1127-1135 (2001).
- Nardmann, J., Ji, J., Werr, W., Scanlon, M. J. The maize duplicate genes narrow sheath1 and narrow sheath2 encode a conserved homeobox gene function in a lateral domain of shoot apical meristems. Development. 131 (12), 2827-2839 (2004).
- Bonner, W. A., Hulett, H. R., Sweet, R. G., Herzenberg, L. A. Fluorescence activated cell sorting. Rev Sci Instrum. 43 (3), 404-409 (1972).
- Birnbaum, K., et al. A gene expression map of the Arabidopsis root. Science. 302 (5652), 1956-1960 (2003).
- Brady, S. M., et al. A high-resolution root spatiotemporal map reveals dominant expression patterns. Science. 318 (5851), 801-806 (2007).
- Carter, A. D., Bonyadi, R., Gifford, M. L. The use of fluorescence-activated cell sorting in studying plant development and environmental responses. Int J Dev Biol. 57 (6-8), 545-552 (2013).
- Ruzin, S. E. . Plant microtechnique and microscopy. , (1999).
- Jackson, D., Veit, B., Hake, S. Expression of maize KNOTTED1 related homeobox genes in the shoot apical meristem predicts patterns of morphogenesis in the vegetative shoot. Development. 120, 405-413 (1994).
- Javelle, M., Marco, C. F., Timmermans, M. In situ hybridization for the precise localization of transcripts in plants. J Vis Exp. (57), e3328 (2011).
- Day, R. C., McNoe, L., Macknight, R. C. Evaluation of global RNA amplification and its use for high-throughput transcript analysis of laser-microdissected endosperm. Int J Plant Genomics. , 61028 (2007).

