Lazer Diseksiyon RNA Sek için Deneysel Tasarım: Mısır Yaprak Kalkınma bir Analiz Dersler
Summary
Many developmentally important genes have cell- or tissue-specific expression patterns. This paper describes LM RNA-seq experiments to identify genes that are differentially expressed at the maize leaf blade-sheath boundary and in lg1-R mutants compared to wild-type. The experimental considerations discussed here apply to transcriptomic analyses of other developmental phenomena.
Abstract
gelişmesinde önemli rolleri olan genler sıklıkla mekansal ve / veya zamansal sınırlı ifade desenleri var. Genellikle bu gen transkript tespit edilmemiştir ya da farklı olarak tüm bitki organlarının transkriptomik analizlerinde (DE) ifade edildiği gibi tanımlanmamıştır. Lazer Diseksiyon RNA Seq (LM RNA Seq) özel gelişimsel alanlarda DE olan genleri tanımlamak için güçlü bir araçtır. Ancak, hücresel alanlarının seçimi microdissect ve karşılaştırmak ve microdissections doğruluğu deneylerin başarısı için çok önemlidir için. Burada, iki örnek transkriptomik deneyler için tasarım konuları göstermektedir; RNA DİZİ analizi, mısır yaprağı yakın-uzak eksen boyunca de genleri belirlemek için bir LM ve ikinci bir deney, DE liguleless1-R (LG1-R) olan genleri tanımlamak için mutantlar vahşi tip ile karşılaştırıldığında. Bu deneylerin başarısına katkıda kilit unsurları histolojik ve si ayrıntılı edilditu melezleme analiz edilecek bölgenin analizleri, eşdeğer gelişim aşamalarında yaprak primordiasında seçimi, morfolojik yerlerinden kullanımı mikrodiseksiyon bölgeleri seçin ve hassas ölçümler etki mikrodisseksiyon için. Bu makale LM RNA Sek tarafından gelişimsel etki analizi için ayrıntılı bir protokol sağlar. Burada sunulan veriler mikrodiseksiyon seçilen bölge elde edilen sonuçları nasıl etkileyeceğini göstermektedir.
Introduction
Genetik diseksiyon (Şekil 1A) için uygun olan bıçak ve kılıf arasında belirgin bir sınır olduğu gibi mısır yaprağı, morfolojilerinden sırasında gelişimsel alanların oluşumunu incelemek için ideal bir model. yaprak gelişimi, küçük hücrelerden oluşan bir lineer grubun ilk aşamalarında, preligule bandı (PLB), pre-bıçak ve ön kılıf etki alanlarına yaprak primordium'u subdivides. Bir saçak gibi ligule ve üçgen kulakcık PLB (Şekil 1A, C, D) gelişir. Genetik ekranlar bıçak kılıf sınırını bozan mutasyonlar saptanmıştır. Örneğin, resesif liguleless1 (LG1) mutasyonları ligule kulakçıkların, 1, 2, 3, 4 (Şekil 1B) silin. İn situ hibridizasyon LG1 transkript P birikir ortayaLB ve ligule gelişme 5, 6 (Şekil 1E) için o mükemmel bir işaretleyici yaparak, ligule ortaya çıkıyor.
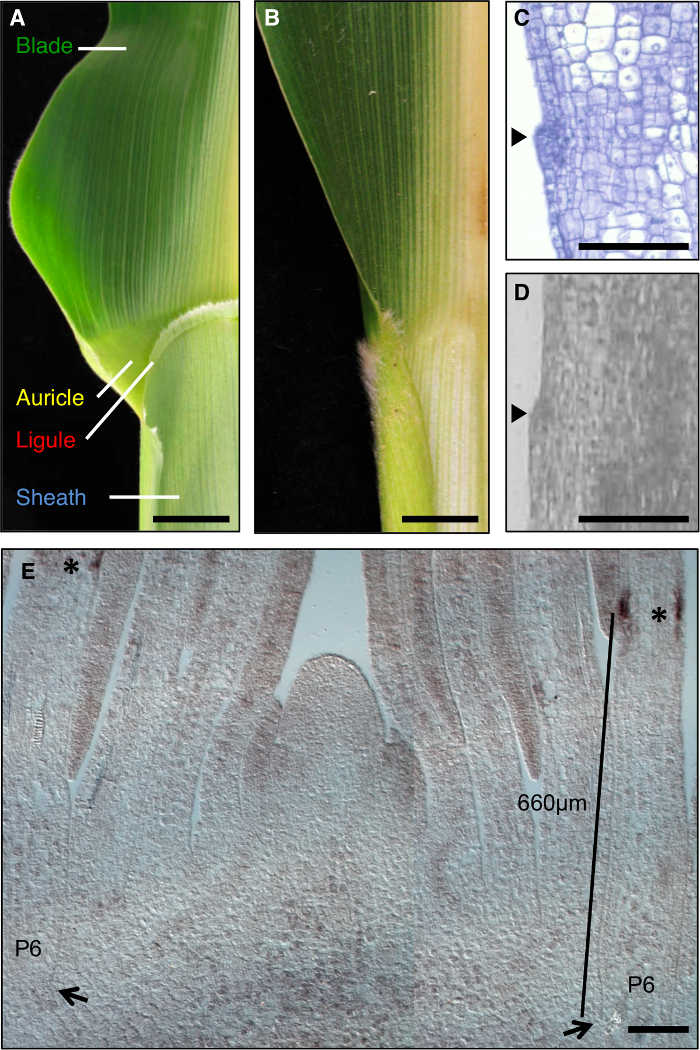
Şekil 1: vahşi tip ve liguleless1-R mısır yaprakları. Ligule ve kulak kepçesi yapıları gösteren, olgun, vahşi tip yaprak (A) Bıçak kaplama sınır bölgesi. (B) ligule ve kulak kepçesi yapıların olgun liguleless1-R yaprak gösteren yokluğunda Bıçak kılıf sınır bölgesi. A ve B Yapraklar yaprak orta damarı boyunca yarıya edilmiştir. Vahşi tip yaprak primordiumunun yoluyla (C) Boyuna kesit. Örnek işleme ve histolojik analiz için boyanmıştır edilmiştir. başlatılması ligule yaprak (ok başı) düzleminden dışarı çıkıntı yapan bir tümsek belirgindir. (D) Boyuna mezhepvahşi tip yaprak primordiumunun yoluyla iyon. metinde tarif edildiği gibi Örnek LM için işlenmiştir. Ok başı ligule başlatılması gösterir. Sürgün ucuna uzunlamasına yanal bölümün in situ hibridizasyon (E) LG1. Yıldız P6 yaprak primordiumunun PLB de LG1 transkript birikimi gösterir. Oklar P6 primordium'un tabanını gösterir. Bar PLB için primordium'un tabanından ölçümünü gösterir. A ve B Ölçek çubukları 20 mm =. CE Ölçek çubukları = 100 mikron. Bu rakam referans 6 (Bitki Biyologlar Telif Amerikan Derneği) modifiye edilmiştir. Bu rakamın büyük halini görmek için lütfen buraya tıklayınız.
Bu çalışmada, LM RNA Seq yaprak primordiumunun ve ide diğer bölgelerine bıçak-kılıf sınır nispetle farklı ifade edilen genlerin bir paketi (DE) tanımlamak için kullanılmıştır vahşi tip kardeşleri göre LG1-R mutantlar DE vardır ntify genler. LM RNA Seq özel hücreler ya da hücre alan 7 transkript birikimi ölçülmesi için bir yöntem olup. LM sistemleri lazer ve bir dijital kamera ile bir mikroskop birleştirir. Kesitli doku slaytlar üzerine monte edilir ve mikroskop aracılığıyla görüntülenebilir. LM yazılım genellikle kullanıcı mikrodiseksiyon seçilen herhangi bir bölgeyi anahat izin çizim araçları içerir. hat boyunca lazer kesim ve seçilen doku slayt kapalı mancınık ve slayt üzerinde asılı bir tüp içine almaktadır. LM kullanıcıya özel hücre katmanları ve hatta tek hücreler 8, 9 da dahil olmak üzere hassas etki, microdissect sağlar. RNA daha sonra microdissected dokusundan elde edilebilir. Daha sonra, RNA-Seq bileşen çıkarılan RNA 10 üretilen cDNA kütüphaneleri sıralamak için yeni nesil dizileme kullanır,= "xref"> 11.
LM RNA seq Anahtar avantajları kesin olarak tanımlanmış alanlarda transkript birikimi ölçmek yeteneği ve tüm transkriptom aynı anda 7 profile kapasite vardır. teknik faiz bölge genellikle mikroskobik erken gelişim olayları sondalama için uygundur. Daha önceki çalışmalar, LM bitkiler 9, 12, 13, gelişim süreçleri incelemek için mikrodizi teknolojisi ile birlikte kullanmıştır. RNA Seq düşük ifade edilen genler de dahil olmak üzere, geniş bir dinamik aralık, arasında transkript miktarının avantajı vardır ve önceki dizi bilgisi, 11 10 gerekli değildir. Ayrıca, LM RNA Seq nedeniyle-of-zarara genetik fazlalık veya öldürücülüğü için mutagenez ekranlarında gözden kaçabilir gelişimsel olarak önemli genlerin vurgulamak için potansiyele sahiptirfonksiyon mutant.
Bu tür dar bir sheath1 (NS1) ve kupa şekilli cotyledon2 (CUC2) halinde gelişimsel olarak önemli genler, genellikle sadece bir spesifik ekspresyon modelleri ya da birkaç hücre 17, 18, 19, 20 sahiptir. Çoğu sadece erken gelişim evrelerinde ve olgun bir organ olarak ifade edilir. Bütün organlar ya da büyük etki incelendiğinde, bu hücreye spesifik transkriptler seyreltilmiş ve daha geleneksel analizlerde tespit edilemez. kesin olarak tanımlanmış etki analiz edilmesine izin vererek, LM RNA Seq bu dokuya özgü genler belirlendi ve miktarları tayin edilmesini sağlar.
Burada tarif edilen deneylerde başarısında önemli faktörlerdir bir analiz için uygun gelişim aşamasında ve etki alanının seçimi güdümlü tam histolojik analiz ve hassas measureme vardıLM hücre dokusunu etki nt. Eş etki her kopya için örneklendi emin olmak için, doku, aynı gelişim aşamasında, yaprak primordia toplandı ve microdissected etki, ortaya çıkan ligule (Şekil 2) gibi morfolojik noktalara göre ölçülmüştür. Bazı genler, yaprak tabanına ucundan gradyanı olarak ifade edilmiştir bilinmektedir. Nedeniyle yaprak proksimal-distal ekseni boyunca farklı yerlerden numune hassas etki, varyasyon ölçerek en az (Şekil 3A) tutuldu. Aynı boyutta etki microdissecting ile nedeniyle değişkenlik hücreye özel transkriptlerin seyreltme (Şekil 3B) indirgenerek diferansiyel. Atış apeksinin yan uzunlamasına kesitleri, tüm microdissections kullanılmıştır. Bunlar (Şekil 4) yaprak orta damarı-margin eksenine dik bölümler vardır. SAM içeren tek bölümleri kullanarak o eşdeğer yanal bölgeleri sağlaryaprak taslakları analiz edilir.
İşlenmiş ve LM için kesitli numunelerde, ligule akıbet ilk morfolojik işareti nedeniyle adaxial epidermis (Şekil 1D, Şekil 2) periclinal hücre bölünmeleri adaxial tarafında bir darbedir. Gelişmekte olan ligule güvenilir plastochron 7 aşamalı yaprak primordiasında tespit edilebilir o belirlenmiştir. Biz gelişmekte olan ligule ve kulak kepçesi oluşturacaktır hemen uzak hücreleri dahil olmak üzere tüm ligule bölgede ifade genler, ilgilenmişlerdir. Eşdeğer doku seçimi yapılırken sağlamak amacıyla, ligule yumru morfolojik işareti olarak kullanıldı ve ligule yumru merkezli 100 um dikdörtgen LM (Şekil 2A, 2B) için seçildi. Ön bıçak ve ön kılıfın aynı büyüklükte dikdörtgenler aynı yaprak primordia seçilmiştir.
liguleless mutant bitkilerin analizi Farklı challe sunulmaktadırnge; LG1-R mutantlar bu nedenle bu morfolojik özellik LM için bölgeyi seçmek için kullanılan olamazdı bir ligule, oluşturmazlar. Bunun yerine, vahşi tip yaprak primordia olarak LG1 transkript birikimi alanı tespit edildi ve bu da kapsamak olan bir bölge tanımlanmıştır. son tahlilde için kullanılan önceki çalışma PLB yeri büyüme koşullarına bağlı olarak değişir göstermiştir beri bu ön analizler, aynı dikimden itibaren fidelerin üzerinde gerçekleştirilmiştir. In situ hibridizasyon LG1 transkript PLB P6 yaprak primordia (Şekil 1 E) birikir olduğunu göstermiştir. Biz LG1 ifadesinin alanını (mor dikdörtgenler, Şekil 2A) kapsıyor ve yabani tip ve LG1-R bitkilerden bu eşdeğer bölgeleri ele yaprak primordiasında tabanından bir alanı 400-900 mikron seçilmiş. transkripsiyonu karşılaştırırken genetik altyapı ve büyüme şartlarına varyasyonu en aza indirmek içinLG1-R ve vahşi tip bitkiler, mutant ve vahşi tipli kardeşlerinden ailelerini ayırma t birikimi kullanılmıştır.
Protocol
Representative Results
Discussion
Deney tasarımı RNA DİZ deneylerinde önemli bir faktördür. Anahtar hususlar kesin etki (ler) ve gelişimsel aşaması (ler) analiz edilecek ve ne karşılaştırmalar yapılacaktır vardır. çıkış iki veya daha fazla durum arasında de genlerin bir liste genellikle, çünkü karşılaştırmalar açısından düşünmek için çok önemlidir. Tüm deneylerde olduğu gibi, aynı anda yalnızca tek bir değişken değiştirmek için önemlidir. Farklı yaprak etki karşılaştırırken Örneğin, aynı koşullar …
Disclosures
The authors have nothing to disclose.
Acknowledgements
Yazarlar işbirliği, devam eden ve ligule gelişimi ile ilgili tartışmalar uyarıcı S. Hake teşekkür ederiz. Bu çalışma, Ulusal Bilim Vakfı Hibe MCB 1052051 ve IOS-1848478 tarafından desteklenmektedir.
Materials
| Diethyl pyrocarbonate | Sigma-Aldrich | 159220 | Used for RNase treatment of solutions |
| Razor blades | Electron Microscopy Sciences | 72000 | |
| RNase Zap | Sigma-Aldrich | R2020-250ML | RNase decontamination solution |
| Ethanol absolute 200 proof | Fisher Scientific | BP28184 | |
| Acetic acid, glacial | Sigma-Aldrich | A6283 | |
| Glass vials – 22mL | VWR | 470206-384 | |
| Xylenes, histological grade | Sigma-Aldrich | 534056-4L | |
| Paraplast plus | Sigma-Aldrich | P3683-1KG | |
| Disposable base molds, 15 x 15 x 5mm | VWR | 15154-072 | |
| Embedding rings | VWR | 15154-303 | |
| Silica gel packets | Electron Microscopy Sciences | 71206-01 | Desiccant for storage of paraffin blocks |
| Oven | Fisher Scientific | 15-103-0503 | Oven must maintain temperature of 60° C |
| Paraffin embedding station | Leica | EG1160 | |
| Microtome | Leica | RM2235 | |
| Slide warmer | Electron Microscopy Sciences | 71317-10 | |
| Coplin jars | Electron Microscopy Sciences | 70316-02 | |
| Laser microdissector | Zeiss | ||
| KIMWIPES™ Delicate Task Wipers | Kimberly-Clark Professional | 34120 | Lint-free wipes for wicking excess solutions from microscope slides |
| Membrane Slide 1.0 PEN | Zeiss | 415190-9041-000 | Slides for laser microdissection |
| Adhesive Cap 200 opaque | Zeiss | 415190-9181-000 | Tubes for laser microdissection |
| PicoPure RNA Isolation Kit | ThermoFisher Scientific | KIT0204 |
References
- Becraft, P. W., Bongard-Pierce, D. K., Sylvester, A. W., Poethig, R. S., Freeling, M. The liguleless-1 gene acts tissue specifically in maize leaf development. Dev Biol. 141 (1), 220-232 (1990).
- Sylvester, A. W., Cande, W. Z., Freeling, M. Division and differentiation during normal and liguleless-1 maize leaf development. Development. 110 (3), 985-1000 (1990).
- Moreno, M. A., Harper, L. C., Krueger, R. W., Dellaporta, S. L., Freeling, M. liguleless1 encodes a nuclear-localized protein required for induction of ligules and auricles during maize leaf organogenesis. Genes Dev. 11 (5), 616-628 (1997).
- Emerson, R. A. The inheritance of the ligule and auricle of corn leaves. Neb. Agr. Exp. Sta. An. Rep. 25, 81-85 (1912).
- Moon, J., Candela, H., Hake, S. The Liguleless narrow mutation affects proximal-distal signaling and leaf growth. Development. 140 (2), 405-412 (2013).
- Johnston, R., et al. Transcriptomic Analyses Indicate That Maize Ligule Development Recapitulates Gene Expression Patterns That Occur during Lateral Organ Initiation. Plant Cell. 26 (12), 4718-4732 (2014).
- Schmid, M. W., et al. A powerful method for transcriptional profiling of specific cell types in eukaryotes: laser-assisted microdissection and RNA sequencing. PLoS One. 7 (1), e29685 (2012).
- Kerk, N. M., Ceserani, T., Tausta, S. L., Sussex, I. M., Nelson, T. M. Laser capture microdissection of cells from plant tissues. Plant Physiol. 132 (1), 27-35 (2003).
- Nakazono, M., Qiu, F., Borsuk, L. A., Schnable, P. S. Laser-capture microdissection, a tool for the global analysis of gene expression in specific plant cell types: identification of genes expressed differentially in epidermal cells or vascular tissues of maize. Plant Cell. 15 (3), 583-596 (2003).
- Marioni, J. C., Mason, C. E., Mane, S. M., Stephens, M., Gilad, Y. RNA-seq: an assessment of technical reproducibility and comparison with gene expression arrays. Genome Res. 18 (9), 1509-1517 (2008).
- Wang, Z., Gerstein, M., Snyder, M. RNA-Seq: a revolutionary tool for transcriptomics. Nat Rev Genet. 10 (1), 57-63 (2009).
- Brooks, L., et al. Microdissection of shoot meristem functional domains. PLoS Genet. 5 (5), e1000476 (2009).
- Cai, S., Lashbrook, C. C. Stamen abscission zone transcriptome profiling reveals new candidates for abscission control: enhanced retention of floral organs in transgenic plants overexpressing Arabidopsis ZINC FINGER PROTEIN2. Plant Physiol. 146 (3), 1305-1321 (2008).
- Li, P., et al. The developmental dynamics of the maize leaf transcriptome. Nat Genet. 42 (12), 1060-1067 (2010).
- Eveland, A. L., et al. Regulatory modules controlling maize inflorescence architecture. Genome Res. 24 (3), 431-443 (2014).
- Takacs, E. M., et al. Ontogeny of the maize shoot apical meristem. Plant Cell. 24 (8), 3219-3234 (2012).
- Aida, M., Ishida, T., Tasaka, M. Shoot apical meristem and cotyledon formation during Arabidopsis embryogenesis: interaction among the CUP-SHAPED COTYLEDON and SHOOT MERISTEMLESS genes. Development. 126 (8), 1563-1570 (1999).
- Ishida, T., Aida, M., Takada, S., Tasaka, M. Involvement of CUP-SHAPED COTYLEDON genes in gynoecium and ovule development in Arabidopsis thaliana. Plant Cell Physiol. 41 (1), 60-67 (2000).
- Takada, S., Hibara, K., Ishida, T., Tasaka, M. The CUP-SHAPED COTYLEDON1 gene of Arabidopsis regulates shoot apical meristem formation. Development. 128 (7), 1127-1135 (2001).
- Nardmann, J., Ji, J., Werr, W., Scanlon, M. J. The maize duplicate genes narrow sheath1 and narrow sheath2 encode a conserved homeobox gene function in a lateral domain of shoot apical meristems. Development. 131 (12), 2827-2839 (2004).
- Bonner, W. A., Hulett, H. R., Sweet, R. G., Herzenberg, L. A. Fluorescence activated cell sorting. Rev Sci Instrum. 43 (3), 404-409 (1972).
- Birnbaum, K., et al. A gene expression map of the Arabidopsis root. Science. 302 (5652), 1956-1960 (2003).
- Brady, S. M., et al. A high-resolution root spatiotemporal map reveals dominant expression patterns. Science. 318 (5851), 801-806 (2007).
- Carter, A. D., Bonyadi, R., Gifford, M. L. The use of fluorescence-activated cell sorting in studying plant development and environmental responses. Int J Dev Biol. 57 (6-8), 545-552 (2013).
- Ruzin, S. E. . Plant microtechnique and microscopy. , (1999).
- Jackson, D., Veit, B., Hake, S. Expression of maize KNOTTED1 related homeobox genes in the shoot apical meristem predicts patterns of morphogenesis in the vegetative shoot. Development. 120, 405-413 (1994).
- Javelle, M., Marco, C. F., Timmermans, M. In situ hybridization for the precise localization of transcripts in plants. J Vis Exp. (57), e3328 (2011).
- Day, R. C., McNoe, L., Macknight, R. C. Evaluation of global RNA amplification and its use for high-throughput transcript analysis of laser-microdissected endosperm. Int J Plant Genomics. , 61028 (2007).

