Experimentell design för Laser Microdissection RNA-Seq: Lektionerna från en analys av majs Leaf utveckling
Summary
Many developmentally important genes have cell- or tissue-specific expression patterns. This paper describes LM RNA-seq experiments to identify genes that are differentially expressed at the maize leaf blade-sheath boundary and in lg1-R mutants compared to wild-type. The experimental considerations discussed here apply to transcriptomic analyses of other developmental phenomena.
Abstract
Gener med viktiga roller i utvecklingen har ofta rumsligt och / eller tidsmässigt begränsade uttrycksmönster. Ofta är dessa gentranskript inte upptäcks eller inte identifieras som differentiellt uttryckt (DE) i transcriptomic analyser av hela växtorgan. Laser Microdissection RNA-Seq (LM RNA-Seq) är ett kraftfullt verktyg för att identifiera gener som är DE särskilda utvecklingsområden. Men valet av cellulära domäner microdissect och jämföra, och noggrannheten av microdissections är avgörande för framgången för experimenten. Här, två exempel illustrerar design överväganden för transkriptomik experiment; en LM-RNA-seq analys för att identifiera gener som är de längs majsblad proximala-distala axeln, och ett andra experiment för att identifiera gener som är DE i liguleless1-R (LG1-R) mutanter jämfört med vildtypen. Viktiga delar som bidragit till framgången för dessa experiment detaljerade histologiska och situ hybridisering analyser av regionen som skall analyseras, val av blad primordia vid motsvarande utvecklingsstadier, användning av morfologiska landmärken för att välja regioner för microdissection, och microdissection av exakt uppmätta domäner. Detta dokument ger en detaljerad protokoll för analys av utvecklings domäner genom LM RNA-Seq. De data som presenteras här visar hur ut för microdissection regionen kommer att påverka resultaten.
Introduction
Bladet majs är en idealisk modell för att studera bildandet av utvecklingsområden under morfogenes, eftersom den har en tydlig gräns mellan bladet och manteln som är mottaglig för genetisk dissektion (Figur 1A). Under de tidiga stadierna av bladutveckling, en linjär band av mindre celler, den preligule bandet (PLB), delar upp blad primordium in i pre-bladiga och pre-slida domäner. En frans-liknande ligule och triangulära flikar utvecklas från PLB (Figur 1A, C, D). Genetiska skärmar har identifierat mutationer som stör blad manteln gränsen. Till exempel, recessiv liguleless1 (LG1) mutationer radera ligule och förmaksöronen 1, 2, 3, 4 (Figur 1B). In situ hybridisering visade att LG1 avskrift ackumuleras i PLB och nya ligule, vilket gör den till en utmärkt markör för ligule utveckling 5, 6 (figur 1E).
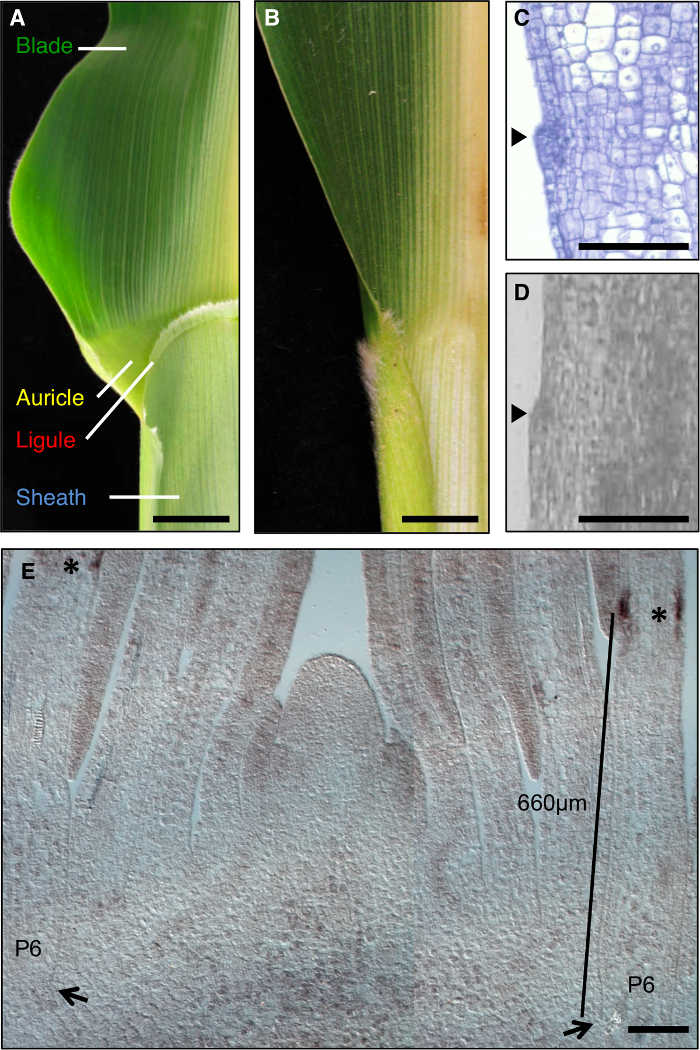
Figur 1: Vildtyp och liguleless1-R majsblad. (A) Blade-mantel gränsområdet av mogen vildtyp blad visar ligule och ytteröra strukturer. (B) Blade-mantel gränsområdet av mogen liguleless1-R blad som visar frånvaro av ligule och ytteröra strukturer. Bladen i A och B har halverats längs Mittnerven. (C) Längdsnitt genom vildtyp leaf primordium. Prov har behandlats och färgades för histologisk analys. Den initierande ligule framgår som en bula som skjuter ut från planet för blad (pilspets). (D) Longitudinell sektjon genom vildtyp leaf primordium. Provet har bearbetats för LM som beskrivs i texten. Pilspets indikerar initiering ligule. (E) LG1 in situ hybridisering av skott apex sidolängdsnitt. Asterisker indikerar LG1 avskrift ansamlingen på PLB av P6 blad primordium. Pilar anger basen av P6 primordium. Bar indikerar mätning från basen av primordium till PLB. Skala barer i A och B = 20 mm. Skala barer i CE = 100 pm. Denna siffra har modifierats referens 6 (Copyright American Society of Plant Biologists). Klicka här för att se en större version av denna siffra.
I denna studie var LM RNA-Seq användas för att identifiera en uppsättning gener som är differentiellt uttryckta (DE) vid bladets-mantelgräns i förhållande till andra delar av blad primordium och till IDE ntify gener som är DE LG1-R-mutanter i förhållande till vildtyp syskon. LM RNA-Seq är en metod för att kvantifiera avskrift ackumulering i specifika celler eller cellulära domäner 7. LM system kombinerar en laser och ett mikroskop med en digital kamera. Sektionerade vävnaden monteras på objektglas och betraktas genom mikroskopet. LM Programvaran innehåller typiskt ritverktyg som gör det möjligt för användaren att beskriva någon utvald region för microdissection. Laser skär längs linjen, och den valda vävnaden slungades från glaset och in i ett rör upphängd ovanför bilden. LM tillåter användaren att microdissect exakta domäner, inklusive specifika cellskikt och även enstaka celler 8, 9. RNA kan sedan extraheras från microdissected vävnaden. Därefter använder RNA-Seq komponent nästa generations sekvensering för att sekvensera cDNA-bibliotek som genereras från det extraherade RNA 10,= "xref"> 11.
Viktigaste fördelarna med LM RNA-punkter är förmågan att kvantifiera avskrift ackumulering i exakt definierade domäner och förmågan att profilera hela transkriptom samtidigt 7. Tekniken är särskilt lämpad för att sondera tidiga utvecklings evenemang där regionen av intresse är ofta mikroskopiska. Tidigare studier har använt LM kombinerat med microarray-teknik för att studera utvecklingsprocesser i växter 9, 12, 13. RNA-Seq har fördelen att kvantifiera transkript över ett brett dynamiskt omfång, inklusive lågt uttryckt gener och före sekvensinformation krävs inte 10, 11. Dessutom har LM RNA-Seq potential att lyfta fram utvecklings viktiga gener som kan missas i mutagenes skärmar på grund av genetiska redundans eller för dödlighet av förlusten-of-funktion mutanten.
Utvecklings viktiga gener, såsom smala sheath1 (NS1) och skålformad cotyledon2 (CUC2), har ofta specifika uttrycksmönster för bara en eller ett fåtal celler 17, 18, 19, 20. Många uttrycks endast under tidiga utvecklingsstadier och inte i den mogna organet. När hela organ eller stora domäner analyseras, är dessa cellspecifika transkript utspädd och kan inte upptäckas i mer konventionella analyser. Genom att tillåta analyser av exakt definierade domäner, LM RNA-Seq möjliggör dessa vävnadsspecifika gener kan identifieras och kvantifieras.
Avgörande faktorer för framgång de experiment som beskrivs här var en grundlig histologisk analys som styrt valet av lämplig utvecklingsstadiet och domän för analys, och exakt MeasureMent av cellvävnads domäner för LM. För att säkerställa att motsvarande domäner samplades för alla replikat, ades vävnad uppsamlades från blad primordia vid samma utvecklingsstadium och de microdissected domänerna mättes i förhållande till morfologiska landmärken, såsom den framväxande ligule (Figur 2). Det är känt att vissa gener uttrycks i en gradient från spetsen till basen av bladet. Genom att mäta exakta domäner, variation på grund av provtagning från olika platser längs blad proximal-distal axel hölls till ett minimum (figur 3A). Genom microdissecting domäner av samma storlek, för att variation på grund differentiell utspädning av cellspecifika transkript minskade också (figur 3B). Laterala längsgående sektioner av shoot apex användes för alla microdissections. Dessa är delar som är vinkelräta mot Mittnerven marginal axeln (Figur 4). Genom att bara använda delar som inkluderar SAM säkerställer att motsvarande sidoområdena avblad primordia analyseras.
I prover som behandlas och sektione för LM, är den första morfologiska tecken på ligule utväxt en bula på adaxial sidan på grund av periclinal celldelningar i adaxial epidermis (Figur 1D, Figur 2). Det konstaterades att den framväxande ligule kan tillförlitligt identifieras på plastochron 7 skede blad primordia. Vi var intresserade av gener som uttrycks i hela ligule regionen, inklusive den framväxande ligule och cellerna omedelbart distala som ska ligga ytterörat. I syfte att säkerställa att motsvarande vävnads urval gjordes, var ligule bula som används som en morfologisk landmärke och en 100 | j, m rektangel centrerad på ligule bump valdes för LM (figur 2A, 2B). Ekvivalenta stora rektanglar av pre-bladet och pre-mantel valdes från samma blad primordia.
Analyser av liguleless muterade växter presenterade en annan ChalleNBE; LG1-R-mutanter inte bildar en ligule, därför denna morfologiska funktionen inte kan användas för att välja region för LM. I stället var domänen för LG1 avskrift ackumulering i vild-typ blad primordia bestämdes och en region som skulle omfatta detta område definierades. Dessa preliminära analyser utfördes på plantor från samma plantering som användes för den slutliga analysen, eftersom tidigare arbete har visat att placeringen av PLB varierar beroende på odlingsbetingelser. In situ hybridisering indikerade att LG1 transkript ackumuleras i PLB av P6 blad primordia (figur 1E). Vi valde en domän 400-900 pm från basen av bladet primordia som omfattade domän LG1 uttryck (lila rektanglar, figur 2A) och fångade dessa motsvarande regioner från vild-typ och LG1-R växter. För att minimera variationer i genetisk bakgrund och tillväxtbetingelser när man jämför transcript ackumulering i LG1-R och vildtypsväxter, segregerande familjer mutanter och vildtyp syskon användes.
Protocol
Representative Results
Discussion
Experimentell design är en kritisk faktor i RNA-seq experiment. Viktiga överväganden är den exakta domän (er) och utvecklingsstadium (er) som skall analyseras, och vilka jämförelser kommer att göras. Det är viktigt att tänka i termer av jämförelser, eftersom produktionen är typiskt en lista av gener som är de mellan två eller flera villkor. Som med alla experiment, är det viktigt att ändra endast en variabel i taget. Till exempel, när man jämför olika blad domäner, blad av samma ålder och utvecklin…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Författarna tackar S. Hake för pågående samarbete och stimulera diskussioner om ligule utveckling. Detta arbete stöds av National Science Foundation MCB 1052051 och IOS-1.848.478.
Materials
| Diethyl pyrocarbonate | Sigma-Aldrich | 159220 | Used for RNase treatment of solutions |
| Razor blades | Electron Microscopy Sciences | 72000 | |
| RNase Zap | Sigma-Aldrich | R2020-250ML | RNase decontamination solution |
| Ethanol absolute 200 proof | Fisher Scientific | BP28184 | |
| Acetic acid, glacial | Sigma-Aldrich | A6283 | |
| Glass vials – 22mL | VWR | 470206-384 | |
| Xylenes, histological grade | Sigma-Aldrich | 534056-4L | |
| Paraplast plus | Sigma-Aldrich | P3683-1KG | |
| Disposable base molds, 15 x 15 x 5mm | VWR | 15154-072 | |
| Embedding rings | VWR | 15154-303 | |
| Silica gel packets | Electron Microscopy Sciences | 71206-01 | Desiccant for storage of paraffin blocks |
| Oven | Fisher Scientific | 15-103-0503 | Oven must maintain temperature of 60° C |
| Paraffin embedding station | Leica | EG1160 | |
| Microtome | Leica | RM2235 | |
| Slide warmer | Electron Microscopy Sciences | 71317-10 | |
| Coplin jars | Electron Microscopy Sciences | 70316-02 | |
| Laser microdissector | Zeiss | ||
| KIMWIPES™ Delicate Task Wipers | Kimberly-Clark Professional | 34120 | Lint-free wipes for wicking excess solutions from microscope slides |
| Membrane Slide 1.0 PEN | Zeiss | 415190-9041-000 | Slides for laser microdissection |
| Adhesive Cap 200 opaque | Zeiss | 415190-9181-000 | Tubes for laser microdissection |
| PicoPure RNA Isolation Kit | ThermoFisher Scientific | KIT0204 |
References
- Becraft, P. W., Bongard-Pierce, D. K., Sylvester, A. W., Poethig, R. S., Freeling, M. The liguleless-1 gene acts tissue specifically in maize leaf development. Dev Biol. 141 (1), 220-232 (1990).
- Sylvester, A. W., Cande, W. Z., Freeling, M. Division and differentiation during normal and liguleless-1 maize leaf development. Development. 110 (3), 985-1000 (1990).
- Moreno, M. A., Harper, L. C., Krueger, R. W., Dellaporta, S. L., Freeling, M. liguleless1 encodes a nuclear-localized protein required for induction of ligules and auricles during maize leaf organogenesis. Genes Dev. 11 (5), 616-628 (1997).
- Emerson, R. A. The inheritance of the ligule and auricle of corn leaves. Neb. Agr. Exp. Sta. An. Rep. 25, 81-85 (1912).
- Moon, J., Candela, H., Hake, S. The Liguleless narrow mutation affects proximal-distal signaling and leaf growth. Development. 140 (2), 405-412 (2013).
- Johnston, R., et al. Transcriptomic Analyses Indicate That Maize Ligule Development Recapitulates Gene Expression Patterns That Occur during Lateral Organ Initiation. Plant Cell. 26 (12), 4718-4732 (2014).
- Schmid, M. W., et al. A powerful method for transcriptional profiling of specific cell types in eukaryotes: laser-assisted microdissection and RNA sequencing. PLoS One. 7 (1), e29685 (2012).
- Kerk, N. M., Ceserani, T., Tausta, S. L., Sussex, I. M., Nelson, T. M. Laser capture microdissection of cells from plant tissues. Plant Physiol. 132 (1), 27-35 (2003).
- Nakazono, M., Qiu, F., Borsuk, L. A., Schnable, P. S. Laser-capture microdissection, a tool for the global analysis of gene expression in specific plant cell types: identification of genes expressed differentially in epidermal cells or vascular tissues of maize. Plant Cell. 15 (3), 583-596 (2003).
- Marioni, J. C., Mason, C. E., Mane, S. M., Stephens, M., Gilad, Y. RNA-seq: an assessment of technical reproducibility and comparison with gene expression arrays. Genome Res. 18 (9), 1509-1517 (2008).
- Wang, Z., Gerstein, M., Snyder, M. RNA-Seq: a revolutionary tool for transcriptomics. Nat Rev Genet. 10 (1), 57-63 (2009).
- Brooks, L., et al. Microdissection of shoot meristem functional domains. PLoS Genet. 5 (5), e1000476 (2009).
- Cai, S., Lashbrook, C. C. Stamen abscission zone transcriptome profiling reveals new candidates for abscission control: enhanced retention of floral organs in transgenic plants overexpressing Arabidopsis ZINC FINGER PROTEIN2. Plant Physiol. 146 (3), 1305-1321 (2008).
- Li, P., et al. The developmental dynamics of the maize leaf transcriptome. Nat Genet. 42 (12), 1060-1067 (2010).
- Eveland, A. L., et al. Regulatory modules controlling maize inflorescence architecture. Genome Res. 24 (3), 431-443 (2014).
- Takacs, E. M., et al. Ontogeny of the maize shoot apical meristem. Plant Cell. 24 (8), 3219-3234 (2012).
- Aida, M., Ishida, T., Tasaka, M. Shoot apical meristem and cotyledon formation during Arabidopsis embryogenesis: interaction among the CUP-SHAPED COTYLEDON and SHOOT MERISTEMLESS genes. Development. 126 (8), 1563-1570 (1999).
- Ishida, T., Aida, M., Takada, S., Tasaka, M. Involvement of CUP-SHAPED COTYLEDON genes in gynoecium and ovule development in Arabidopsis thaliana. Plant Cell Physiol. 41 (1), 60-67 (2000).
- Takada, S., Hibara, K., Ishida, T., Tasaka, M. The CUP-SHAPED COTYLEDON1 gene of Arabidopsis regulates shoot apical meristem formation. Development. 128 (7), 1127-1135 (2001).
- Nardmann, J., Ji, J., Werr, W., Scanlon, M. J. The maize duplicate genes narrow sheath1 and narrow sheath2 encode a conserved homeobox gene function in a lateral domain of shoot apical meristems. Development. 131 (12), 2827-2839 (2004).
- Bonner, W. A., Hulett, H. R., Sweet, R. G., Herzenberg, L. A. Fluorescence activated cell sorting. Rev Sci Instrum. 43 (3), 404-409 (1972).
- Birnbaum, K., et al. A gene expression map of the Arabidopsis root. Science. 302 (5652), 1956-1960 (2003).
- Brady, S. M., et al. A high-resolution root spatiotemporal map reveals dominant expression patterns. Science. 318 (5851), 801-806 (2007).
- Carter, A. D., Bonyadi, R., Gifford, M. L. The use of fluorescence-activated cell sorting in studying plant development and environmental responses. Int J Dev Biol. 57 (6-8), 545-552 (2013).
- Ruzin, S. E. . Plant microtechnique and microscopy. , (1999).
- Jackson, D., Veit, B., Hake, S. Expression of maize KNOTTED1 related homeobox genes in the shoot apical meristem predicts patterns of morphogenesis in the vegetative shoot. Development. 120, 405-413 (1994).
- Javelle, M., Marco, C. F., Timmermans, M. In situ hybridization for the precise localization of transcripts in plants. J Vis Exp. (57), e3328 (2011).
- Day, R. C., McNoe, L., Macknight, R. C. Evaluation of global RNA amplification and its use for high-throughput transcript analysis of laser-microdissected endosperm. Int J Plant Genomics. , 61028 (2007).

