बाध्यकारी microscale thermophoresis का उपयोग करते हुए एटीपी पर एक Aptamer की साइट मानचित्रण
Summary
MicroScale Thermophoresis (MST) is a sensitive technology to characterize aptamer-target interactions. This manuscript describes an MST protocol to characterize aptamer-small molecule interactions.
Abstract
Characterization of molecular interactions in terms of basic binding parameters such as binding affinity, stoichiometry, and thermodynamics is an essential step in basic and applied science. MicroScale Thermophoresis (MST) is a sensitive biophysical method to obtain this important information. Relying on a physical effect called thermophoresis, which describes the movement of molecules through temperature gradients, this technology allows for the fast and precise determination of binding parameters in solution and allows the free choice of buffer conditions (from buffer to lysates/sera). MST uses the fact that an unbound molecule displays a different thermophoretic movement than a molecule that is in complex with a binding partner. The thermophoretic movement is altered in the moment of molecular interaction due to changes in size, charge, and hydration shell. By comparing the movement profiles of different molecular ratios of the two binding partners, quantitative information such as binding affinity (pM to mM) can be determined. Even challenging interactions between molecules of small sizes, such as aptamers and small compounds, can be studied by MST. Using the well-studied model interaction between the DH25.42 DNA aptamer and ATP, this manuscript provides a protocol to characterize aptamer-small molecule interactions. This study demonstrates that MST is highly sensitive and permits the mapping of the binding site of the 7.9 kDa DNA aptamer to the adenine of ATP.
Introduction
अणुओं के बीच बातचीत प्रकृति का आधार है। इसलिए, बुनियादी और अनुप्रयुक्त अनुसंधान के कई क्षेत्रों में वैज्ञानिकों ने विभिन्न प्रकार के आणविक बातचीत के मौलिक सिद्धांतों को समझने की कोशिश करते हैं। Microscale thermophoresis (एमएसटी) बफ़र्स के एक मुक्त विकल्प के साथ, समाधान में तेजी, सटीक, लागत, कुशल, और गुणवत्ता नियंत्रण आणविक बातचीत के लक्षण वर्णन प्रदर्शन करने के लिए वैज्ञानिकों को सक्षम बनाता है। वहां पहले से ही अकेले मासिक सीजन टिकट का उपयोग कर, 2016 से, पुस्तकालय चोकर सहित विश्लेषण के विभिन्न प्रकार, का वर्णन, कई बंधन भागीदारों 1-8 के साथ घटना सत्यापन, प्रतियोगिता assays, और प्रयोगों बाध्यकारी 1,000 से अधिक प्रकाशन कर रहे हैं। सामान्य में, इस तरह के मासिक सीजन टिकट बंधन आत्मीयता (मिमी बजे), stoichiometry, और ऊष्मा, आणविक बातचीत की किसी भी तरह के रूप में शास्त्रीय बाध्यकारी मापदंडों के अध्ययन के लिए परमिट। एमएसटी का एक बड़ा लाभ बातचीत भागीदारों के आकार के स्वतंत्र बाध्यकारी घटनाओं का अध्ययन करने की क्षमता है। यहाँ तक कि chalइस तरह के छोटे अणुओं, दवाओं, एंटीबायोटिक दवाओं, या चयापचयों के रूप में छोटे न्यूक्लिक एसिड aptamers के बीच बातचीत lenging (15-30 NT) और लक्ष्य मात्रा निर्धारित किया जा सकता है।
वर्तमान राज्य के अत्याधुनिक प्रौद्योगिकियों को चिह्नित aptamer लक्ष्य बातचीत या तो प्रयोगशाला तीव्र और अत्यधिक जटिल कर रहे हैं या यों तो aptamer-छोटे अणु बातचीत 9,10 विफल करने के लिए। सतह plasmon अनुनाद (एसपीआर) आधारित assays 11,12 और ऐसे इज़ोटेर्माल अनुमापन calorimetry (आईटीसी) 13-15, isocratic क्षालन 16, संतुलन फाई ltration 17,18 के रूप में वास्तव लेबल मुक्त उष्मापन दृष्टिकोण,, में लाइन की जांच कर रही 19, gel- assays बदलाव, stopped- FL ओउ FL uorescence स्पेक्ट्रोस्कोपी 20,21, प्रतिदीप्ति anisotropy (एफए) 22,23, एकल अणु फ्लोरिडा uorescence इमेजिंग 24,25, और बायो-परत इंटरफेरोमेट्री (BLI) 26 भी या तो imprecise या aptamer-छोटे अणु के साथ असंगत हैं बातचीत। अन्य principaइन तरीकों में से एल मुद्दों कम संवेदनशीलता, उच्च नमूना खपत, स्थिरीकरण, सतहों पर जन परिवहन सीमाओं, और / या बफर प्रतिबंध है। इन प्रौद्योगिकियों के केवल कुछ ही एकत्रीकरण और सोखना प्रभाव के लिए एकीकृत नियंत्रण प्रदान करते हैं।
एमएसटी वैज्ञानिकों ऐसे प्रोटीन 30-33 के रूप में aptamers और छोटे अणुओं 27-29 के बीच बातचीत, साथ ही अन्य ठिकानों अध्ययन करने के लिए इस सीमा को पार करने के लिए एक शक्तिशाली उपकरण का प्रतिनिधित्व करता है। प्रौद्योगिकी तापमान ढ़ाल के माध्यम से अणुओं के आंदोलन पर निर्भर करता है। यह निर्देशित आंदोलन, तथाकथित "thermophoresis," आकार, प्रभारी, और अणु 34,35 के हाइड्रेशन खोल पर निर्भर करता है। अणु को एक ligand के बंधन सीधे इन मानकों में से कम से कम एक बदल जाएगा, एक बदल thermophoretic गतिशीलता में जिसके परिणामस्वरूप। छोटे आकार के साथ ligands अनबाउंड से ही राज्य को आकार परिवर्तन के मामले में काफी प्रभाव नहीं हो सकता है, लेकिन वे डॉ हो सकता है हाइड्रेशन खोल और / या आरोप पर amatic प्रभाव। बाध्यकारी साथी के साथ बातचीत के बाद अणुओं के thermophoretic आंदोलन में परिवर्तन के बुनियादी मानकों को बाध्यकारी 2,7,34,36,37 की मात्रा का ठहराव सक्षम बनाता है।
जैसा कि चित्र में दिखाया गया है 1 ए, एमएसटी डिवाइस प्रतिदीप्ति का पता लगाने के लिए के रूप में ही प्रकाशिकी का उपयोग कांच केशिकाओं के भीतर नमूना पर ध्यान केंद्रित एक अवरक्त लेजर के होते हैं। जबकि लेजर एक तापमान ढाल (2-6 डिग्री सेल्सियस के ΔT) स्थापित करता है tryptophans 6 या एक fluorescently लेबल बातचीत साथी 3,8 की आंतरिक FL uorescence के माध्यम से प्रोटीन के thermophoretic आंदोलन पर नजर रखी जा सकती है। अंतरिक्ष, ΔT, जिसके परिणामस्वरूप तापमान अंतर कमी या ऊंचा तापमान के क्षेत्र में अणुओं का संचय, जो Soret द्वारा मात्रा निर्धारित किया जा सकता है की ओर जाता है coef फाई cient (एस टी):
जी "/>
सी गर्म गर्म क्षेत्र में एकाग्रता का प्रतिनिधित्व करता है, और सी ठंड प्रारंभिक ठंड क्षेत्र में एकाग्रता है।
चित्रा 1 बी, एक मासिक सीजन टिकट आंदोलन प्रोफ़ाइल (समय का पता लगाने), अलग-अलग चरणों है, जो अपने संबंधित timescales के द्वारा अलग किया जा सकता से मिलकर में एक ठेठ एमएसटी प्रयोग के परिणाम में दिखाया गया है। प्रारंभिक प्रतिदीप्ति तापमान ढाल के अभाव में पहले 5 एस में मापा जाता है सटीक शुरू प्रतिदीप्ति परिभाषित करने के लिए और photobleaching या photoenhancement के लिए जाँच करें। तापमान कूद (टी-कूद) चरण में जो thermophoretic आंदोलन से पहले प्रतिदीप्ति परिवर्तन का प्रतिनिधित्व करता है। प्रतिदीप्ति में यह प्रारंभिक कमी क्वांटम उपज uorophore फ्लोरिडा की गर्मी पर निर्भर परिवर्तन पर निर्भर करता है। thermophoresis चरण इस प्रकार है, जिसमें रोशनी कम हो जाती है (या बढ़ जाती है) स्थिर राज्य वितरण तक के अणुओं की thermophoretic आंदोलन की वजह से पहुँच जाता है।के रूप में चित्रा 1 बी में संकेत लेजर के बाद बंद है रिवर्स TJump और फ्लोरिडा uorescent अणुओं के सहवर्ती वापस प्रसार मनाया जा सकता है। बुनियादी बाध्यकारी मापदंडों का उपयोग करने के लिए, बातचीत के भागीदारों के विभिन्न दाढ़ अनुपात का विश्लेषण किया और तुलना कर रहे हैं। आमतौर पर, 16 विभिन्न अनुपात, एक मासिक सीजन टिकट प्रयोग में अध्ययन कर रहे हैं, जबकि ऑप्टिकल दिखाई अणु स्थिर रखा जाता है और लेबल हटाया गया ligand के एक बढ़ती हुई राशि के साथ आपूर्ति की है। दो बंधन भागीदारों के बीच बातचीत thermophoresis में परिवर्तन लाती है, और इस तरह सामान्यीकृत FL uorescence में, एफ आदर्श है, जो निम्नलिखित के रूप में गणना की है:
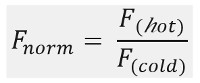
एफ गर्म और ठंडे एफ एमएसटी निशान के डी फाई नेड समय बिंदुओं पर फ्लोरिडा uorescence तीव्रता के औसत का प्रतिनिधित्व करते हैं। बाध्यकारी समानताएं (कश्मीर डी या चुनाव आयोग 50 मान) curv द्वारा गणना की जा सकती हैई फिटिंग (चित्रा 1 सी)।
कुल मिलाकर, मासिक सीजन टिकट किसी भी प्रकार की आणविक बातचीत का अध्ययन करने के लिए एक शक्तिशाली उपकरण है। और 25-NT कम ssDNA aptamer DH25.42 (7.9 केडीए); इस पांडुलिपि छोटे अणु adenosine triphosphate (0.5 केडीए एटीपी) के बीच चुनौतीपूर्ण बातचीत को चिह्नित करने के लिए एक प्रोटोकॉल प्रदान करता है। पांडुलिपि के दौरान, एटीपी अणु पर aptamer के बंधन साइट एटीपी के adenine समूह के लिए नीचे मैप किया गया है।
Protocol
Representative Results
Discussion
गुणवत्ता नियंत्रण:
Unspecific चिपके / सतहों के लिए नमूना सामग्री, साथ ही एकत्रीकरण प्रभाव के सोखना, आत्मीयता के डेटा की गुणवत्ता पर एक नाटकीय प्रभाव है। हालांकि, केवल कुछ ही राज्य के अत्याधुनिक प्रौ?…
Disclosures
The authors have nothing to disclose.
Acknowledgements
लेखकों कोई स्वीकृतियां है।
Materials
| Aptamer binding buffer | 20 mM Tris pH7.6; 300 mM NaCl; 5 mM MgCl2; 0.01%Tween-20 | ||
| Fluorescently labeled ATP aptamer | IDT, Leuven, Belgium | sequence: DH25.42 50-Cy5-CCTGGGGGAGT-ATTGCGGAGGAAGG-3 | |
| ATP | Sigma Aldrich, Germany | A2383 | 10 mM stock solutions stored at – 20 °C |
| ADP | Sigma Aldrich, Germany | A2754 | 10 mM stock solutions stored at – 20 °C |
| AMP | Sigma Aldrich, Germany | A2252 | 10 mM stock solutions stored at – 20 °C |
| Adenine | Sigma Aldrich, Germany | A8626 | 10 mM stock solutions stored at – 20 °C |
| SAM | Sigma Aldrich, Germany | A7007 | 10 mM stock solutions stored at – 20 °C |
| dATP | Sigma Aldrich, Germany | 11934511001 | 10 mM stock solutions stored at – 20 °C |
| CTP | Sigma Aldrich, Germany | C1506 | 10 mM stock solutions stored at – 20 °C |
| GTP | Sigma Aldrich, Germany | G8877 | 10 mM stock solutions stored at – 20 °C |
| Monolith NT.115 | NanoTemper Technologies, Munich, Germany | MO-G008 | Blue/Red Channel MST device with standard detector, Monolith NT115 pico is MST device with high sensitivity detector |
| Monolith NT.115 capillaries Standard | NanoTemper Technologies, Munich, Germany | MO-K002 | |
| Eppendorf PCR tubes | Eppendorf, Germany | 30124537 | |
| Monolith control software. 2.1.33, pre-installed on the device | NanoTemper Technologies, Munich, Germany | ||
| MO.affinity analysis v2.1.1 | NanoTemper Technologies, Munich, Germany | ||
| Kaleidagraph 4.5.2 | Synergy Software |
References
- Linke, P., et al. An Automated Microscale Thermophoresis Screening Approach for Fragment-Based Lead Discovery. J Biomol Screen. 21 (4), 414-421 (2015).
- Jerabek-Willemsen, M., et al. MicroScale Thermophoresis: Interaction analysis and beyond. Journal of Molecular Structure. 1077, 101-113 (2014).
- Zillner, K., et al. Microscale thermophoresis as a sensitive method to quantify protein: nucleic acid interactions in solution. Methods Mol Biol. 815, 241-252 (2012).
- Zhang, W., Duhr, S., Baaske, P., Laue, E. Microscale thermophoresis for the assessment of nuclear protein-binding affinities. Methods Mol Biol. 1094, 269-276 (2014).
- Wienken, C. J., Baaske, P., Rothbauer, U., Braun, D., Duhr, S. Protein-binding assays in biological liquids using microscale thermophoresis. Nat Commun. 1, 100 (2010).
- Seidel, S. A., et al. Label-free microscale thermophoresis discriminates sites and affinity of protein-ligand binding. Angew Chem Int Ed Engl. 51 (42), 10656-10659 (2012).
- Seidel, S. A., et al. Microscale thermophoresis quantifies biomolecular interactions under previously challenging conditions. Methods. 59 (3), 301-315 (2013).
- Schubert, T., et al. Df31 protein and snoRNAs maintain accessible higher-order structures of chromatin. Mol Cell. 48 (3), 434-444 (2012).
- McKeague, M., Derosa, M. C. Challenges and opportunities for small molecule aptamer development. J Nucleic Acids. 2012, 748913 (2012).
- Ruscito, A., DeRosa, M. C. Small-Molecule Binding Aptamers: Selection Strategies, Characterization, and Applications. Front Chem. 4, 14 (2016).
- Chang, A. L., McKeague, M., Liang, J. C., Smolke, C. D. Kinetic and equilibrium binding characterization of aptamers to small molecules using a label-free, sensitive, and scalable platform. Anal Chem. 86 (7), 3273-3278 (2014).
- Chang, A. L., McKeague, M., Smolke, C. D. Facile characterization of aptamer kinetic and equilibrium binding properties using surface plasmon resonance. Methods Enzymol. 549, 451-466 (2014).
- Jing, M., Bowser, M. T. Methods for measuring aptamer-protein equilibria: a review. Anal Chim Acta. 686 (1-2), 9-18 (2011).
- Sokoloski, J. E., Dombrowski, S. E., Bevilacqua, P. C. Thermodynamics of ligand binding to a heterogeneous RNA population in the malachite green aptamer. Biochemistry. 51 (1), 565-572 (2012).
- Burnouf, D., et al. kinITC: a new method for obtaining joint thermodynamic and kinetic data by isothermal titration calorimetry. J Am Chem Soc. 134 (1), 559-565 (2012).
- Mannironi, C., Scerch, C., Fruscoloni, P., Tocchini-Valentini, G. P. Molecular recognition of amino acids by RNA aptamers: the evolution into an L-tyrosine binder of a dopamine-binding RNA motif. RNA. 6 (4), 520-527 (2000).
- Jenison, R. D., Gill, S. C., Pardi, A., Polisky, B. High-resolution molecular discrimination by RNA. Science. 263 (5152), 1425-1429 (1994).
- Huizenga, D. E., Szostak, J. W. A DNA aptamer that binds adenosine and ATP. Biochemistry. 34 (2), 656-665 (1995).
- Lee, E. R., Baker, J. L., Weinberg, Z., Sudarsan, N., Breaker, R. R. An allosteric self-splicing ribozyme triggered by a bacterial second messenger. Science. 329 (5993), 845-848 (2010).
- Wickiser, J. K., Cheah, M. T., Breaker, R. R., Crothers, D. M. The kinetics of ligand binding by an adenine-sensing riboswitch. Biochemistry. 44 (40), 13404-13414 (2005).
- Jucker, F. M., Phillips, R. M., McCallum, S. A., Pardi, A. Role of a heterogeneous free state in the formation of a specific RNA-theophylline complex. Biochemistry. 42 (9), 2560-2567 (2003).
- Zhao, Q., Lv, Q., Wang, H. Aptamer fluorescence anisotropy sensors for adenosine triphosphate by comprehensive screening tetramethylrhodamine labeled nucleotides. Biosens Bioelectron. 70, 188-193 (2015).
- Zhang, D., et al. A sensitive fluorescence anisotropy method for detection of lead (II) ion by a G-quadruplex-inducible DNA aptamer. Anal Chim Acta. 812, 161-167 (2014).
- Elenko, M. P., Szostak, J. W., van Oijen, A. M. Single-molecule imaging of an in vitro-evolved RNA aptamer reveals homogeneous ligand binding kinetics. J Am Chem Soc. 131 (29), 9866-9867 (2009).
- Elenko, M. P., Szostak, J. W., van Oijen, A. M. Single-molecule binding experiments on long time scales. Rev Sci Instrum. 81 (8), 083705 (2010).
- Zichel, R., Chearwae, W., Pandey, G. S., Golding, B., Sauna, Z. E. Aptamers as a sensitive tool to detect subtle modifications in therapeutic proteins. PLoS One. 7 (2), 31948 (2012).
- Baaske, P., Wienken, C. J., Reineck, P., Duhr, S., Braun, D. Optical thermophoresis for quantifying the buffer dependence of aptamer binding. Angew Chem Int Ed Engl. 49 (12), 2238-2241 (2010).
- Entzian, C., Schubert, T. Studying small molecule-aptamer interactions using MicroScale Thermophoresis (MST). Methods. 97, 27-34 (2016).
- Valenzano, S., et al. Screening and Identification of DNA Aptamers to Tyramine Using in Vitro Selection and High-Throughput Sequencing. ACS Comb Sci. 18 (6), 302-313 (2016).
- Jauset Rubio, M., et al. beta-Conglutin dual aptamers binding distinct aptatopes. Anal Bioanal Chem. 408 (3), 875-884 (2016).
- Breitsprecher, D., et al. Aptamer Binding Studies Using MicroScale Thermophoresis. Methods Mol Biol. 1380, 99-111 (2016).
- Stoltenburg, R., Schubert, T., Strehlitz, B. In vitro Selection and Interaction Studies of a DNA Aptamer Targeting Protein A. PLoS One. 10 (7), 0134403 (2015).
- Kinghorn, A. B., et al. Aptamer Affinity Maturation by Resampling and Microarray Selection. Anal Chem. 88 (14), 6981-6985 (2016).
- Duhr, S., Braun, D. Why molecules move along a temperature gradient. Proc Natl Acad Sci U S A. 103 (52), 19678-19682 (2006).
- Braun, D., Libchaber, A. Trapping of DNA by thermophoretic depletion and convection. Phys Rev Lett. 89 (18), 188103 (2002).
- Duhr, S., Arduini, S., Braun, D. Thermophoresis of DNA determined by microfluidic fluorescence. Eur Phys J E Soft Matter. 15 (3), 277-286 (2004).
- Jerabek-Willemsen, M., Wienken, C. J., Braun, D., Baaske, P., Duhr, S. Molecular interaction studies using microscale thermophoresis. Assay Drug Dev Technol. 9 (4), 342-353 (2011).
- He, K., Dragnea, V., Bauer, C. E. Adenylate Charge Regulates Sensor Kinase CheS3 To Control Cyst Formation in Rhodospirillum centenum. MBio. 6 (3), 00546 (2015).
- Brvar, M., et al. Structure-based discovery of substituted 4,5′-bithiazoles as novel DNA gyrase inhibitors. J Med Chem. 55 (14), 6413-6426 (2012).
- Pogorelcnik, B., et al. 4,6-Substituted-1,3,5-triazin-2(1H)-ones as monocyclic catalytic inhibitors of human DNA topoisomerase IIalpha targeting the ATP binding site. Bioorg Med Chem. 23 (15), 4218-4229 (2015).
- Jhaveri, S., Rajendran, M., Ellington, A. D. In vitro selection of signaling aptamers. Nat Biotechnol. 18 (12), 1293-1297 (2000).
- Khavrutskii, L., et al. Protein purification-free method of binding affinity determination by microscale thermophoresis. J Vis Exp. (78), (2013).
- Ramakrishnan, M., et al. Probing cocaine-antibody interactions in buffer and human serum. PLoS One. 7 (7), 40518 (2012).
- Chen, M., et al. Antiviral activity and interaction mechanisms study of novel glucopyranoside derivatives. Bioorg Med Chem Lett. 25 (18), 3840-3844 (2015).
- Wan, C., et al. Insights into the molecular recognition of the granuphilin C2A domain with PI(4,5)P2. Chem Phys Lipids. 186 (4,5), 61-67 (2015).
- Harazi, A., et al. The Interaction of UDP-N-Acetylglucosamine 2-Epimerase/N-Acetylmannosamine Kinase (GNE) and Alpha-Actinin 2 Is Altered in GNE Myopathy M743T Mutant. Mol Neurobiol. , (2016).

