In kaart brengen van de bindingsplaats van een aptameer op ATP gebruik van microschaal Thermophoresis
Summary
MicroScale Thermophoresis (MST) is a sensitive technology to characterize aptamer-target interactions. This manuscript describes an MST protocol to characterize aptamer-small molecule interactions.
Abstract
Characterization of molecular interactions in terms of basic binding parameters such as binding affinity, stoichiometry, and thermodynamics is an essential step in basic and applied science. MicroScale Thermophoresis (MST) is a sensitive biophysical method to obtain this important information. Relying on a physical effect called thermophoresis, which describes the movement of molecules through temperature gradients, this technology allows for the fast and precise determination of binding parameters in solution and allows the free choice of buffer conditions (from buffer to lysates/sera). MST uses the fact that an unbound molecule displays a different thermophoretic movement than a molecule that is in complex with a binding partner. The thermophoretic movement is altered in the moment of molecular interaction due to changes in size, charge, and hydration shell. By comparing the movement profiles of different molecular ratios of the two binding partners, quantitative information such as binding affinity (pM to mM) can be determined. Even challenging interactions between molecules of small sizes, such as aptamers and small compounds, can be studied by MST. Using the well-studied model interaction between the DH25.42 DNA aptamer and ATP, this manuscript provides a protocol to characterize aptamer-small molecule interactions. This study demonstrates that MST is highly sensitive and permits the mapping of the binding site of the 7.9 kDa DNA aptamer to the adenine of ATP.
Introduction
Interactie tussen moleculen is de basis van de natuur. Vandaar dat wetenschappers op vele terreinen van fundamenteel en toegepast onderzoek proberen om de fundamentele principes van de moleculaire interacties van verschillende soorten te begrijpen. Microschaal Thermophoresis (MST) kunnen wetenschappers de snelle, nauwkeurige, kostenefficiënte en op kwaliteit gecontroleerde karakterisering van moleculaire interacties uit te voeren in een oplossing, met een vrije keuze van buffers. Er zijn al meer dan 1.000 publicaties met behulp van MST, van 2016 alleen al, de beschrijving van verschillende soorten analyses, met inbegrip van de bibliotheek screenings, bindende event validaties, de concurrentie assays, en experimenten met meerdere bindende partners 1-8. Over het algemeen MST maakt de studie van de klassieke bindingsparameters, zoals bindingsaffiniteit (pM tot mM), stoichiometrie en thermodynamica, van elke soort moleculaire interactie. Een groot voordeel van MST is het vermogen binding gebeurtenissen onafhankelijk van de grootte van de interactiepartners bestuderen. zelfs challenging interacties tussen kleine nucleïnezuur aptameren (15-30 nt) en targets zoals kleine moleculen, geneesmiddelen, antibiotica, of metabolieten kunnen worden gekwantificeerd.
De huidige state-of-the-art technologieën te karakteriseren aptameer-target interacties ofwel lab-intense en zeer complexe of niet te kwantificeren aptameer-klein molecuul interacties 9,10. Surface Plasmon Resonance (SPR) gebaseerde assays 11,12 en echt label-free calorimetrische benaderingen, zoals Isotherme Titratie Calorimetrie (ITC) 13-15, isocratische elutie 16, evenwicht filtratie 17,18, in-line sonderen 19, gel- shift assays, stoppunt fl ow fluorescentie-spectroscopie 20,21, fluorescentie anisotropie (FA) 22,23, single-molecule fluorescentie beeldvorming 24,25, en Bio-layer interferometrie (BLI) 26 zijn ook ofwel onnauwkeurig of niet verenigbaar met aptameer-klein molecuul interacties. andere principal vraagstukken van deze methoden zijn lage gevoeligheid, hoge monster verbruik, immobilisatie, massatransport beperkingen op oppervlakken, en / of buffer beperkingen. Slechts een paar van deze technologie te bieden geïntegreerde schakelaars voor aggregatie en adsorptie effecten.
MST is een krachtig hulpmiddel voor wetenschappers om deze beperking op de interactie tussen aptameren en kleine moleculen 27-29, alsmede andere doelen zoals eiwitten 30-33 bestuderen overwonnen. De technologie is gebaseerd op de beweging van moleculen door temperatuurgradiënten. Deze gerichte beweging, genaamd "thermophoresis," afhankelijk van de grootte, lading en hydratatie shell van het molecuul 34,35. De binding van een ligand aan het molecuul direct ten minste één van deze parameters verandert, wat resulteert in een veranderde mobiliteit thermophoretic. Liganden met kleine maten kan geen aanzienlijke gevolgen in termen van grootte veranderen van de niet-gebonden aan de gebonden toestand, maar ze kunnen dr hebben amatic effecten op de hydratatie shell en / of kosten. De veranderingen in de thermophoretic beweging van moleculen na interactie met de bindende partner maakt de kwantificering van fundamentele bindingsparameters 2,7,34,36,37.
Zoals weergegeven in figuur 1A, de MST inrichting bestaat uit een infrarood laser gefocust op het monster in de glazen capillairen met dezelfde optiek als voor fluorescentiedetectie. De thermophoretic beweging van eiwitten via de intrinsieke fluorescentie van tryptofaan 6 of een fluorescent gelabelde interactiepartner 3,8 kan worden bewaakt terwijl de laser wordt een temperatuurgradiënt (AT van 2-6 ° C). Het resulterende temperatuurverschil in de ruimte, AT, leidt tot de uitputting of accumulatie van moleculen in het gebied van verhoogde temperatuur, die kan worden gekwantificeerd door de Soret coëfficiënt (S T):
g "/>
c hete geeft de concentratie in het verwarmde gebied, en c koud de concentratie in de oorspronkelijke koud gebied.
Zoals getoond in figuur 1B, een typische MST experiment resulteert in een MST bewegingsprofiel (tijddiagram), bestaande uit verschillende fasen, die gescheiden kunnen worden door hun respectieve tijdschema. De initiële fluorescentie gemeten in de eerste 5 s bij afwezigheid van de temperatuurgradiënt de precieze start fluorescentie definiëren en controleren fotobleken of photoenhancement. De temperatuursprong (T-Jump) vertegenwoordigt de fase waarin de fluorescentieveranderingen voor thermophoretic beweging. Deze initiële afname in fluorescentie afhankelijk warmte-afhankelijke veranderingen van fl uorophore kwantumopbrengst. De thermophoresis fase volgt, waarbij de fluorescentie afneemt (of verhogingen) door de thermophoretic beweging van de molecules tot de steady-state verdeling wordt bereikt.Het omgekeerde TJump en gelijktijdige terugdiffusie van fluorescerende moleculen zoals aangegeven in figuur 1B nadat de laser is uitgeschakeld worden waargenomen. Om bij fundamentele bindingsparameters worden verschillende molaire verhoudingen van de interactiepartners geanalyseerd en vergeleken. Gewoonlijk worden 16 verschillende ratio's bestudeerd in een MST experiment, terwijl de optische zichtbaar molecuul constant wordt gehouden en wordt geleverd met een toenemende hoeveelheid van de niet-gelabelde ligand. De interactie tussen de twee bindingspartners induceert veranderingen in de thermophoresis, en dus de genormaliseerde fluorescentie, F norm, die wordt berekend als volgt:
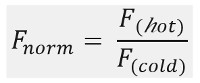
F warme en koude F vertegenwoordigen gemiddelde fluorescentie-intensiteiten in de gedefinieerde tijdstippen van het MST sporen. Bindende affiniteiten (Kd of EC 50 waarden) kan worden berekend door curve fitting (figuur 1C).
Over het geheel genomen MST is een krachtig hulpmiddel om de moleculaire interacties van welke aard dan ook te bestuderen. Dit manuscript biedt een protocol bij de uitdagende interactie te karakteriseren tussen de kleine molecule adenosine trifosfaat (ATP; 0,5 kDa) en de 25-nt korte ssDNA aptameer DH25.42 (7,9 kDa). In de loop van het manuscript wordt de bindingsplaats van de aptameer aan het ATP molecuul in kaart gebracht naar de adenine groep van ATP.
Protocol
Representative Results
Discussion
Kwaliteitscontroles:
Aspecifieke plakken / adsorptie van monstermateriaal op oppervlakken en aggregatie effecten, een wezenlijke invloed op de kwaliteit van de affiniteit gegevens. Slechts enkele state-of-the-art technologieën bieden een nauwkeurige en snelle opties te controleren en deze te vermijden. MST biedt geïntegreerde kwaliteitscontroles die detecteren en helpen om deze problemen te overwinnen, waardoor de stapsgewijze optimalisatie van de technische installatie. Belangrijke informati…
Disclosures
The authors have nothing to disclose.
Acknowledgements
De auteurs hebben geen bevestigingen.
Materials
| Aptamer binding buffer | 20 mM Tris pH7.6; 300 mM NaCl; 5 mM MgCl2; 0.01%Tween-20 | ||
| Fluorescently labeled ATP aptamer | IDT, Leuven, Belgium | sequence: DH25.42 50-Cy5-CCTGGGGGAGT-ATTGCGGAGGAAGG-3 | |
| ATP | Sigma Aldrich, Germany | A2383 | 10 mM stock solutions stored at – 20 °C |
| ADP | Sigma Aldrich, Germany | A2754 | 10 mM stock solutions stored at – 20 °C |
| AMP | Sigma Aldrich, Germany | A2252 | 10 mM stock solutions stored at – 20 °C |
| Adenine | Sigma Aldrich, Germany | A8626 | 10 mM stock solutions stored at – 20 °C |
| SAM | Sigma Aldrich, Germany | A7007 | 10 mM stock solutions stored at – 20 °C |
| dATP | Sigma Aldrich, Germany | 11934511001 | 10 mM stock solutions stored at – 20 °C |
| CTP | Sigma Aldrich, Germany | C1506 | 10 mM stock solutions stored at – 20 °C |
| GTP | Sigma Aldrich, Germany | G8877 | 10 mM stock solutions stored at – 20 °C |
| Monolith NT.115 | NanoTemper Technologies, Munich, Germany | MO-G008 | Blue/Red Channel MST device with standard detector, Monolith NT115 pico is MST device with high sensitivity detector |
| Monolith NT.115 capillaries Standard | NanoTemper Technologies, Munich, Germany | MO-K002 | |
| Eppendorf PCR tubes | Eppendorf, Germany | 30124537 | |
| Monolith control software. 2.1.33, pre-installed on the device | NanoTemper Technologies, Munich, Germany | ||
| MO.affinity analysis v2.1.1 | NanoTemper Technologies, Munich, Germany | ||
| Kaleidagraph 4.5.2 | Synergy Software |
References
- Linke, P., et al. An Automated Microscale Thermophoresis Screening Approach for Fragment-Based Lead Discovery. J Biomol Screen. 21 (4), 414-421 (2015).
- Jerabek-Willemsen, M., et al. MicroScale Thermophoresis: Interaction analysis and beyond. Journal of Molecular Structure. 1077, 101-113 (2014).
- Zillner, K., et al. Microscale thermophoresis as a sensitive method to quantify protein: nucleic acid interactions in solution. Methods Mol Biol. 815, 241-252 (2012).
- Zhang, W., Duhr, S., Baaske, P., Laue, E. Microscale thermophoresis for the assessment of nuclear protein-binding affinities. Methods Mol Biol. 1094, 269-276 (2014).
- Wienken, C. J., Baaske, P., Rothbauer, U., Braun, D., Duhr, S. Protein-binding assays in biological liquids using microscale thermophoresis. Nat Commun. 1, 100 (2010).
- Seidel, S. A., et al. Label-free microscale thermophoresis discriminates sites and affinity of protein-ligand binding. Angew Chem Int Ed Engl. 51 (42), 10656-10659 (2012).
- Seidel, S. A., et al. Microscale thermophoresis quantifies biomolecular interactions under previously challenging conditions. Methods. 59 (3), 301-315 (2013).
- Schubert, T., et al. Df31 protein and snoRNAs maintain accessible higher-order structures of chromatin. Mol Cell. 48 (3), 434-444 (2012).
- McKeague, M., Derosa, M. C. Challenges and opportunities for small molecule aptamer development. J Nucleic Acids. 2012, 748913 (2012).
- Ruscito, A., DeRosa, M. C. Small-Molecule Binding Aptamers: Selection Strategies, Characterization, and Applications. Front Chem. 4, 14 (2016).
- Chang, A. L., McKeague, M., Liang, J. C., Smolke, C. D. Kinetic and equilibrium binding characterization of aptamers to small molecules using a label-free, sensitive, and scalable platform. Anal Chem. 86 (7), 3273-3278 (2014).
- Chang, A. L., McKeague, M., Smolke, C. D. Facile characterization of aptamer kinetic and equilibrium binding properties using surface plasmon resonance. Methods Enzymol. 549, 451-466 (2014).
- Jing, M., Bowser, M. T. Methods for measuring aptamer-protein equilibria: a review. Anal Chim Acta. 686 (1-2), 9-18 (2011).
- Sokoloski, J. E., Dombrowski, S. E., Bevilacqua, P. C. Thermodynamics of ligand binding to a heterogeneous RNA population in the malachite green aptamer. Biochemistry. 51 (1), 565-572 (2012).
- Burnouf, D., et al. kinITC: a new method for obtaining joint thermodynamic and kinetic data by isothermal titration calorimetry. J Am Chem Soc. 134 (1), 559-565 (2012).
- Mannironi, C., Scerch, C., Fruscoloni, P., Tocchini-Valentini, G. P. Molecular recognition of amino acids by RNA aptamers: the evolution into an L-tyrosine binder of a dopamine-binding RNA motif. RNA. 6 (4), 520-527 (2000).
- Jenison, R. D., Gill, S. C., Pardi, A., Polisky, B. High-resolution molecular discrimination by RNA. Science. 263 (5152), 1425-1429 (1994).
- Huizenga, D. E., Szostak, J. W. A DNA aptamer that binds adenosine and ATP. Biochemistry. 34 (2), 656-665 (1995).
- Lee, E. R., Baker, J. L., Weinberg, Z., Sudarsan, N., Breaker, R. R. An allosteric self-splicing ribozyme triggered by a bacterial second messenger. Science. 329 (5993), 845-848 (2010).
- Wickiser, J. K., Cheah, M. T., Breaker, R. R., Crothers, D. M. The kinetics of ligand binding by an adenine-sensing riboswitch. Biochemistry. 44 (40), 13404-13414 (2005).
- Jucker, F. M., Phillips, R. M., McCallum, S. A., Pardi, A. Role of a heterogeneous free state in the formation of a specific RNA-theophylline complex. Biochemistry. 42 (9), 2560-2567 (2003).
- Zhao, Q., Lv, Q., Wang, H. Aptamer fluorescence anisotropy sensors for adenosine triphosphate by comprehensive screening tetramethylrhodamine labeled nucleotides. Biosens Bioelectron. 70, 188-193 (2015).
- Zhang, D., et al. A sensitive fluorescence anisotropy method for detection of lead (II) ion by a G-quadruplex-inducible DNA aptamer. Anal Chim Acta. 812, 161-167 (2014).
- Elenko, M. P., Szostak, J. W., van Oijen, A. M. Single-molecule imaging of an in vitro-evolved RNA aptamer reveals homogeneous ligand binding kinetics. J Am Chem Soc. 131 (29), 9866-9867 (2009).
- Elenko, M. P., Szostak, J. W., van Oijen, A. M. Single-molecule binding experiments on long time scales. Rev Sci Instrum. 81 (8), 083705 (2010).
- Zichel, R., Chearwae, W., Pandey, G. S., Golding, B., Sauna, Z. E. Aptamers as a sensitive tool to detect subtle modifications in therapeutic proteins. PLoS One. 7 (2), 31948 (2012).
- Baaske, P., Wienken, C. J., Reineck, P., Duhr, S., Braun, D. Optical thermophoresis for quantifying the buffer dependence of aptamer binding. Angew Chem Int Ed Engl. 49 (12), 2238-2241 (2010).
- Entzian, C., Schubert, T. Studying small molecule-aptamer interactions using MicroScale Thermophoresis (MST). Methods. 97, 27-34 (2016).
- Valenzano, S., et al. Screening and Identification of DNA Aptamers to Tyramine Using in Vitro Selection and High-Throughput Sequencing. ACS Comb Sci. 18 (6), 302-313 (2016).
- Jauset Rubio, M., et al. beta-Conglutin dual aptamers binding distinct aptatopes. Anal Bioanal Chem. 408 (3), 875-884 (2016).
- Breitsprecher, D., et al. Aptamer Binding Studies Using MicroScale Thermophoresis. Methods Mol Biol. 1380, 99-111 (2016).
- Stoltenburg, R., Schubert, T., Strehlitz, B. In vitro Selection and Interaction Studies of a DNA Aptamer Targeting Protein A. PLoS One. 10 (7), 0134403 (2015).
- Kinghorn, A. B., et al. Aptamer Affinity Maturation by Resampling and Microarray Selection. Anal Chem. 88 (14), 6981-6985 (2016).
- Duhr, S., Braun, D. Why molecules move along a temperature gradient. Proc Natl Acad Sci U S A. 103 (52), 19678-19682 (2006).
- Braun, D., Libchaber, A. Trapping of DNA by thermophoretic depletion and convection. Phys Rev Lett. 89 (18), 188103 (2002).
- Duhr, S., Arduini, S., Braun, D. Thermophoresis of DNA determined by microfluidic fluorescence. Eur Phys J E Soft Matter. 15 (3), 277-286 (2004).
- Jerabek-Willemsen, M., Wienken, C. J., Braun, D., Baaske, P., Duhr, S. Molecular interaction studies using microscale thermophoresis. Assay Drug Dev Technol. 9 (4), 342-353 (2011).
- He, K., Dragnea, V., Bauer, C. E. Adenylate Charge Regulates Sensor Kinase CheS3 To Control Cyst Formation in Rhodospirillum centenum. MBio. 6 (3), 00546 (2015).
- Brvar, M., et al. Structure-based discovery of substituted 4,5′-bithiazoles as novel DNA gyrase inhibitors. J Med Chem. 55 (14), 6413-6426 (2012).
- Pogorelcnik, B., et al. 4,6-Substituted-1,3,5-triazin-2(1H)-ones as monocyclic catalytic inhibitors of human DNA topoisomerase IIalpha targeting the ATP binding site. Bioorg Med Chem. 23 (15), 4218-4229 (2015).
- Jhaveri, S., Rajendran, M., Ellington, A. D. In vitro selection of signaling aptamers. Nat Biotechnol. 18 (12), 1293-1297 (2000).
- Khavrutskii, L., et al. Protein purification-free method of binding affinity determination by microscale thermophoresis. J Vis Exp. (78), (2013).
- Ramakrishnan, M., et al. Probing cocaine-antibody interactions in buffer and human serum. PLoS One. 7 (7), 40518 (2012).
- Chen, M., et al. Antiviral activity and interaction mechanisms study of novel glucopyranoside derivatives. Bioorg Med Chem Lett. 25 (18), 3840-3844 (2015).
- Wan, C., et al. Insights into the molecular recognition of the granuphilin C2A domain with PI(4,5)P2. Chem Phys Lipids. 186 (4,5), 61-67 (2015).
- Harazi, A., et al. The Interaction of UDP-N-Acetylglucosamine 2-Epimerase/N-Acetylmannosamine Kinase (GNE) and Alpha-Actinin 2 Is Altered in GNE Myopathy M743T Mutant. Mol Neurobiol. , (2016).

