La mappatura del sito di legame di un Aptamero su ATP Utilizzando MicroScale termoforesi
Summary
MicroScale Thermophoresis (MST) is a sensitive technology to characterize aptamer-target interactions. This manuscript describes an MST protocol to characterize aptamer-small molecule interactions.
Abstract
Characterization of molecular interactions in terms of basic binding parameters such as binding affinity, stoichiometry, and thermodynamics is an essential step in basic and applied science. MicroScale Thermophoresis (MST) is a sensitive biophysical method to obtain this important information. Relying on a physical effect called thermophoresis, which describes the movement of molecules through temperature gradients, this technology allows for the fast and precise determination of binding parameters in solution and allows the free choice of buffer conditions (from buffer to lysates/sera). MST uses the fact that an unbound molecule displays a different thermophoretic movement than a molecule that is in complex with a binding partner. The thermophoretic movement is altered in the moment of molecular interaction due to changes in size, charge, and hydration shell. By comparing the movement profiles of different molecular ratios of the two binding partners, quantitative information such as binding affinity (pM to mM) can be determined. Even challenging interactions between molecules of small sizes, such as aptamers and small compounds, can be studied by MST. Using the well-studied model interaction between the DH25.42 DNA aptamer and ATP, this manuscript provides a protocol to characterize aptamer-small molecule interactions. This study demonstrates that MST is highly sensitive and permits the mapping of the binding site of the 7.9 kDa DNA aptamer to the adenine of ATP.
Introduction
L'interazione tra le molecole è la base della natura. Quindi, gli scienziati in molti campi della ricerca di base e applicata cercano di capire i principi fondamentali delle interazioni molecolari di diversi tipi. MicroScale termoforesi (MST) consente agli scienziati di effettuare il veloce, preciso, caratterizzazione economicamente efficiente e di qualità controllata delle interazioni molecolari in soluzione, con una scelta di buffer. Ci sono già più di 1.000 pubblicazioni utilizzando MST, a partire dal 2016 da solo, che descrive i diversi tipi di analisi, tra cui proiezioni di libreria, convalide di eventi, analisi della concorrenza, e gli esperimenti di legame con partner multipli vincolanti 1-8. In generale, MST consente lo studio dei parametri vincolanti classici, come affinità di legame (pM a mM), stechiometria e termodinamica, di qualsiasi tipo di interazione molecolare. Un grande vantaggio di MST è la capacità di studiare eventi di legame indipendenti dalla dimensione dei partner di interazione. anche Chalinterazioni lenging tra piccole aptameri di acidi nucleici (15-30 nt) e target, quali piccole molecole, farmaci, antibiotici, o metaboliti possono essere quantificati.
Le attuali tecnologie state-of-the-art di caratterizzare le interazioni aptamer bersaglio sono o laboratorio-intenso e molto complesso o non riescono a quantificare aptameri-piccola molecola Interazioni 9,10. Risonanza plasmonica di superficie (SPR) a base di test di 11,12 e approcci calorimetrica veramente libero-label, come Calorimetria isotermica di titolazione (ITC) 13-15, eluizione isocratica 16, equilibrio filtrazione 17,18, in linea di sondaggio 19, gel- spostare saggi, stopped- flusso fluorescenza spettroscopia 20,21, anisotropia di fluorescenza (FA) 22,23, singola molecola fluorescenza di imaging 24,25, e Bio-strato di interferometria (BLI) 26 siano anche imprecisi o incompatibile con la molecola aptameri-piccole interazioni. Altro Principal temi di questi metodi sono bassa sensibilità, elevato consumo di campione, l'immobilizzazione, limitazioni di trasporto di massa su superfici e / o limitazioni del buffer. Solo pochi di queste tecnologie forniscono controlli integrati per aggregazione e di adsorbimento effetti.
MST rappresenta un potente strumento per gli scienziati di superare questa limitazione per studiare le interazioni tra aptameri e piccole molecole 27-29, nonché altri obiettivi come le proteine 30-33. La tecnologia si basa sul movimento delle molecole attraverso gradienti di temperatura. Questo movimento diretto, chiamato "termoforesi," dipende dalle dimensioni, carica, e shell idratazione della molecola 34,35. Il legame di un ligando alla molecola modificherà direttamente almeno uno di questi parametri, con un conseguente mobilità thermophoretic cambiato. Ligandi con le piccole dimensioni non possono avere un notevole impatto in termini di cambio formato dal non associata alla stato legato, ma possono avere dr effetti Amatic sul guscio di idratazione e / o carica. I cambiamenti nel movimento delle molecole thermophoretic dopo interazioni con il partner legante consente la quantificazione dei parametri vincolanti di base 2,7,34,36,37.
Come illustrato in figura 1A, il dispositivo MST costituito da un laser a infrarossi focalizzato sul campione entro i capillari di vetro con la stessa ottica come per la rilevazione della fluorescenza. Il movimento thermophoretic delle proteine tramite fluorescenza intrinseca di triptofani 6 o di un 3,8 socio interazione fluorescente può essere monitorato mentre il laser stabilisce un gradiente di temperatura (DT di 2-6 ° C). La differenza di temperatura risultante nello spazio, DT, porta alla deplezione o accumulo di molecole nella zona di temperatura elevata, che può essere quantificato dal Soret coefficiente (S T):
g "/>
c rappresenta caldo la concentrazione nella regione riscaldata, ec freddo è la concentrazione nella regione fredda iniziale.
Come mostrato nella Figura 1B, un tipico esperimento MST ottiene un profilo di movimento MST (traccia temporale), costituito da diverse fasi, che possono essere separati dai rispettivi tempi. La fluorescenza iniziale è misurata nei primi 5 s in assenza del gradiente di temperatura per definire la precisione di fluorescenza iniziale e per controllare photobleaching o photoenhancement. Il salto di temperatura (T-Jump) rappresenta la fase in cui le variazioni di fluorescenza prima del movimento thermophoretic. Questa diminuzione iniziale della fluorescenza dipende da sbalzi termici-dipendente di fl uorophore resa quantica. La fase termoforesi segue, in cui diminuisce fluorescenza (o aumenta) dovuti al movimento thermophoretic delle molecole fino alla distribuzione dello stato stazionario viene raggiunto.Il tjump retromarcia e retrodiffusione concomitante di molecole fl uorescenti possono essere osservati come indicato in Figura 1B dopo il laser è spento. Per accedere ai parametri di base vincolanti, diversi rapporti molari dei partner di interazione sono analizzati e confrontati. Tipicamente, 16 differenti rapporti sono studiati in un esperimento MST, considerando che la molecola visibile ottica viene mantenuta costante ed è fornita con una quantità crescente di ligando non marcato. L'interazione tra i due partner di legame induce cambiamenti nella termoforesi, e quindi nella fluorescenza normalizzato, F norma, che viene calcolato come segue:
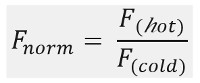
F calda e fredda F rappresentano una media di fl intensità fluorescenza a de fi nite momenti delle tracce MST. Affinità di legame (K D o valori di EC 50) possono essere calcolati curve montaggio (Figura 1C).
Nel complesso, MST è un potente strumento per studiare le interazioni molecolari di qualsiasi tipo. Questo manoscritto offre un protocollo per caratterizzare l'interazione sfida tra la piccola molecola di adenosina trifosfato (ATP; 0,5 kDa) e il 25-nt breve ssDNA aptameri DH25.42 (7,9 kDa). Nel corso del manoscritto, il sito di legame del aptamer sulla molecola ATP viene mappato verso il gruppo adenina dell'ATP.
Protocol
Representative Results
Discussion
Controlli di qualità:
Aspecifico sticking / adsorbimento di materiale campione a superfici, così come gli effetti di aggregazione, hanno una influenza notevole sulla qualità dei dati di affinità. Tuttavia, solo un paio di tecnologie state-of-the-art offrono opzioni precise e rapide per monitorare ed evitare questi effetti. MST offre controlli di qualità integrati che consentono di rilevare e aiutano a superare questi problemi, consentendo l'ottimizzazione graduale della configurazione …
Disclosures
The authors have nothing to disclose.
Acknowledgements
Gli autori non hanno riconoscimenti.
Materials
| Aptamer binding buffer | 20 mM Tris pH7.6; 300 mM NaCl; 5 mM MgCl2; 0.01%Tween-20 | ||
| Fluorescently labeled ATP aptamer | IDT, Leuven, Belgium | sequence: DH25.42 50-Cy5-CCTGGGGGAGT-ATTGCGGAGGAAGG-3 | |
| ATP | Sigma Aldrich, Germany | A2383 | 10 mM stock solutions stored at – 20 °C |
| ADP | Sigma Aldrich, Germany | A2754 | 10 mM stock solutions stored at – 20 °C |
| AMP | Sigma Aldrich, Germany | A2252 | 10 mM stock solutions stored at – 20 °C |
| Adenine | Sigma Aldrich, Germany | A8626 | 10 mM stock solutions stored at – 20 °C |
| SAM | Sigma Aldrich, Germany | A7007 | 10 mM stock solutions stored at – 20 °C |
| dATP | Sigma Aldrich, Germany | 11934511001 | 10 mM stock solutions stored at – 20 °C |
| CTP | Sigma Aldrich, Germany | C1506 | 10 mM stock solutions stored at – 20 °C |
| GTP | Sigma Aldrich, Germany | G8877 | 10 mM stock solutions stored at – 20 °C |
| Monolith NT.115 | NanoTemper Technologies, Munich, Germany | MO-G008 | Blue/Red Channel MST device with standard detector, Monolith NT115 pico is MST device with high sensitivity detector |
| Monolith NT.115 capillaries Standard | NanoTemper Technologies, Munich, Germany | MO-K002 | |
| Eppendorf PCR tubes | Eppendorf, Germany | 30124537 | |
| Monolith control software. 2.1.33, pre-installed on the device | NanoTemper Technologies, Munich, Germany | ||
| MO.affinity analysis v2.1.1 | NanoTemper Technologies, Munich, Germany | ||
| Kaleidagraph 4.5.2 | Synergy Software |
References
- Linke, P., et al. An Automated Microscale Thermophoresis Screening Approach for Fragment-Based Lead Discovery. J Biomol Screen. 21 (4), 414-421 (2015).
- Jerabek-Willemsen, M., et al. MicroScale Thermophoresis: Interaction analysis and beyond. Journal of Molecular Structure. 1077, 101-113 (2014).
- Zillner, K., et al. Microscale thermophoresis as a sensitive method to quantify protein: nucleic acid interactions in solution. Methods Mol Biol. 815, 241-252 (2012).
- Zhang, W., Duhr, S., Baaske, P., Laue, E. Microscale thermophoresis for the assessment of nuclear protein-binding affinities. Methods Mol Biol. 1094, 269-276 (2014).
- Wienken, C. J., Baaske, P., Rothbauer, U., Braun, D., Duhr, S. Protein-binding assays in biological liquids using microscale thermophoresis. Nat Commun. 1, 100 (2010).
- Seidel, S. A., et al. Label-free microscale thermophoresis discriminates sites and affinity of protein-ligand binding. Angew Chem Int Ed Engl. 51 (42), 10656-10659 (2012).
- Seidel, S. A., et al. Microscale thermophoresis quantifies biomolecular interactions under previously challenging conditions. Methods. 59 (3), 301-315 (2013).
- Schubert, T., et al. Df31 protein and snoRNAs maintain accessible higher-order structures of chromatin. Mol Cell. 48 (3), 434-444 (2012).
- McKeague, M., Derosa, M. C. Challenges and opportunities for small molecule aptamer development. J Nucleic Acids. 2012, 748913 (2012).
- Ruscito, A., DeRosa, M. C. Small-Molecule Binding Aptamers: Selection Strategies, Characterization, and Applications. Front Chem. 4, 14 (2016).
- Chang, A. L., McKeague, M., Liang, J. C., Smolke, C. D. Kinetic and equilibrium binding characterization of aptamers to small molecules using a label-free, sensitive, and scalable platform. Anal Chem. 86 (7), 3273-3278 (2014).
- Chang, A. L., McKeague, M., Smolke, C. D. Facile characterization of aptamer kinetic and equilibrium binding properties using surface plasmon resonance. Methods Enzymol. 549, 451-466 (2014).
- Jing, M., Bowser, M. T. Methods for measuring aptamer-protein equilibria: a review. Anal Chim Acta. 686 (1-2), 9-18 (2011).
- Sokoloski, J. E., Dombrowski, S. E., Bevilacqua, P. C. Thermodynamics of ligand binding to a heterogeneous RNA population in the malachite green aptamer. Biochemistry. 51 (1), 565-572 (2012).
- Burnouf, D., et al. kinITC: a new method for obtaining joint thermodynamic and kinetic data by isothermal titration calorimetry. J Am Chem Soc. 134 (1), 559-565 (2012).
- Mannironi, C., Scerch, C., Fruscoloni, P., Tocchini-Valentini, G. P. Molecular recognition of amino acids by RNA aptamers: the evolution into an L-tyrosine binder of a dopamine-binding RNA motif. RNA. 6 (4), 520-527 (2000).
- Jenison, R. D., Gill, S. C., Pardi, A., Polisky, B. High-resolution molecular discrimination by RNA. Science. 263 (5152), 1425-1429 (1994).
- Huizenga, D. E., Szostak, J. W. A DNA aptamer that binds adenosine and ATP. Biochemistry. 34 (2), 656-665 (1995).
- Lee, E. R., Baker, J. L., Weinberg, Z., Sudarsan, N., Breaker, R. R. An allosteric self-splicing ribozyme triggered by a bacterial second messenger. Science. 329 (5993), 845-848 (2010).
- Wickiser, J. K., Cheah, M. T., Breaker, R. R., Crothers, D. M. The kinetics of ligand binding by an adenine-sensing riboswitch. Biochemistry. 44 (40), 13404-13414 (2005).
- Jucker, F. M., Phillips, R. M., McCallum, S. A., Pardi, A. Role of a heterogeneous free state in the formation of a specific RNA-theophylline complex. Biochemistry. 42 (9), 2560-2567 (2003).
- Zhao, Q., Lv, Q., Wang, H. Aptamer fluorescence anisotropy sensors for adenosine triphosphate by comprehensive screening tetramethylrhodamine labeled nucleotides. Biosens Bioelectron. 70, 188-193 (2015).
- Zhang, D., et al. A sensitive fluorescence anisotropy method for detection of lead (II) ion by a G-quadruplex-inducible DNA aptamer. Anal Chim Acta. 812, 161-167 (2014).
- Elenko, M. P., Szostak, J. W., van Oijen, A. M. Single-molecule imaging of an in vitro-evolved RNA aptamer reveals homogeneous ligand binding kinetics. J Am Chem Soc. 131 (29), 9866-9867 (2009).
- Elenko, M. P., Szostak, J. W., van Oijen, A. M. Single-molecule binding experiments on long time scales. Rev Sci Instrum. 81 (8), 083705 (2010).
- Zichel, R., Chearwae, W., Pandey, G. S., Golding, B., Sauna, Z. E. Aptamers as a sensitive tool to detect subtle modifications in therapeutic proteins. PLoS One. 7 (2), 31948 (2012).
- Baaske, P., Wienken, C. J., Reineck, P., Duhr, S., Braun, D. Optical thermophoresis for quantifying the buffer dependence of aptamer binding. Angew Chem Int Ed Engl. 49 (12), 2238-2241 (2010).
- Entzian, C., Schubert, T. Studying small molecule-aptamer interactions using MicroScale Thermophoresis (MST). Methods. 97, 27-34 (2016).
- Valenzano, S., et al. Screening and Identification of DNA Aptamers to Tyramine Using in Vitro Selection and High-Throughput Sequencing. ACS Comb Sci. 18 (6), 302-313 (2016).
- Jauset Rubio, M., et al. beta-Conglutin dual aptamers binding distinct aptatopes. Anal Bioanal Chem. 408 (3), 875-884 (2016).
- Breitsprecher, D., et al. Aptamer Binding Studies Using MicroScale Thermophoresis. Methods Mol Biol. 1380, 99-111 (2016).
- Stoltenburg, R., Schubert, T., Strehlitz, B. In vitro Selection and Interaction Studies of a DNA Aptamer Targeting Protein A. PLoS One. 10 (7), 0134403 (2015).
- Kinghorn, A. B., et al. Aptamer Affinity Maturation by Resampling and Microarray Selection. Anal Chem. 88 (14), 6981-6985 (2016).
- Duhr, S., Braun, D. Why molecules move along a temperature gradient. Proc Natl Acad Sci U S A. 103 (52), 19678-19682 (2006).
- Braun, D., Libchaber, A. Trapping of DNA by thermophoretic depletion and convection. Phys Rev Lett. 89 (18), 188103 (2002).
- Duhr, S., Arduini, S., Braun, D. Thermophoresis of DNA determined by microfluidic fluorescence. Eur Phys J E Soft Matter. 15 (3), 277-286 (2004).
- Jerabek-Willemsen, M., Wienken, C. J., Braun, D., Baaske, P., Duhr, S. Molecular interaction studies using microscale thermophoresis. Assay Drug Dev Technol. 9 (4), 342-353 (2011).
- He, K., Dragnea, V., Bauer, C. E. Adenylate Charge Regulates Sensor Kinase CheS3 To Control Cyst Formation in Rhodospirillum centenum. MBio. 6 (3), 00546 (2015).
- Brvar, M., et al. Structure-based discovery of substituted 4,5′-bithiazoles as novel DNA gyrase inhibitors. J Med Chem. 55 (14), 6413-6426 (2012).
- Pogorelcnik, B., et al. 4,6-Substituted-1,3,5-triazin-2(1H)-ones as monocyclic catalytic inhibitors of human DNA topoisomerase IIalpha targeting the ATP binding site. Bioorg Med Chem. 23 (15), 4218-4229 (2015).
- Jhaveri, S., Rajendran, M., Ellington, A. D. In vitro selection of signaling aptamers. Nat Biotechnol. 18 (12), 1293-1297 (2000).
- Khavrutskii, L., et al. Protein purification-free method of binding affinity determination by microscale thermophoresis. J Vis Exp. (78), (2013).
- Ramakrishnan, M., et al. Probing cocaine-antibody interactions in buffer and human serum. PLoS One. 7 (7), 40518 (2012).
- Chen, M., et al. Antiviral activity and interaction mechanisms study of novel glucopyranoside derivatives. Bioorg Med Chem Lett. 25 (18), 3840-3844 (2015).
- Wan, C., et al. Insights into the molecular recognition of the granuphilin C2A domain with PI(4,5)P2. Chem Phys Lipids. 186 (4,5), 61-67 (2015).
- Harazi, A., et al. The Interaction of UDP-N-Acetylglucosamine 2-Epimerase/N-Acetylmannosamine Kinase (GNE) and Alpha-Actinin 2 Is Altered in GNE Myopathy M743T Mutant. Mol Neurobiol. , (2016).

