Kartlegging av bindingssetet på en Aptamer på ATP Bruke mikro termodiffusjon
Summary
MicroScale Thermophoresis (MST) is a sensitive technology to characterize aptamer-target interactions. This manuscript describes an MST protocol to characterize aptamer-small molecule interactions.
Abstract
Characterization of molecular interactions in terms of basic binding parameters such as binding affinity, stoichiometry, and thermodynamics is an essential step in basic and applied science. MicroScale Thermophoresis (MST) is a sensitive biophysical method to obtain this important information. Relying on a physical effect called thermophoresis, which describes the movement of molecules through temperature gradients, this technology allows for the fast and precise determination of binding parameters in solution and allows the free choice of buffer conditions (from buffer to lysates/sera). MST uses the fact that an unbound molecule displays a different thermophoretic movement than a molecule that is in complex with a binding partner. The thermophoretic movement is altered in the moment of molecular interaction due to changes in size, charge, and hydration shell. By comparing the movement profiles of different molecular ratios of the two binding partners, quantitative information such as binding affinity (pM to mM) can be determined. Even challenging interactions between molecules of small sizes, such as aptamers and small compounds, can be studied by MST. Using the well-studied model interaction between the DH25.42 DNA aptamer and ATP, this manuscript provides a protocol to characterize aptamer-small molecule interactions. This study demonstrates that MST is highly sensitive and permits the mapping of the binding site of the 7.9 kDa DNA aptamer to the adenine of ATP.
Introduction
Interaksjon mellom molekylene er grunnlaget for naturen. Derfor forskere i mange felt av grunnleggende og anvendt forskning prøve å forstå de grunnleggende prinsippene i molekylære interaksjoner av ulike slag. Mikro termodiffusjon (MST) gjør det mulig for forskere å utføre raske, presise, kostnadseffektive og kvalitetssikret karakterisering av molekylære interaksjoner i løsning, med et fritt valg av buffere. Det er allerede mer enn 1000 publikasjoner bruke MST, fra 2016 alene, som beskriver ulike typer analyser, inkludert bibliotek screenings, bindende event valideringer, konkurranseanalyser, og eksperimenter med flere bindingspartnere 1-8. Generelt tillater MST studiet av de klassiske bindende parametere, slik som bindingsaffinitet (pM til mM), støkiometri, og termodynamikk, av en hvilken som helst form for molekylær interaksjon. En stor fordel med MST er evnen til å studere bindings hendelser uavhengig av størrelsen på interaksjonspartnere. selv utfordrende interaksjoner mellom små nukleinsyre aptamerer (15-30 nt) og mål som små molekyler, narkotika, antibiotika, eller metabolitter kan kvantifiseres.
Nåværende state-of-the-art teknologi for å karakterisere aptamer-målet interaksjoner er enten lab-intens og svært komplekse eller unnlater å kvantifisere aptamer-lite molekyl interaksjoner 9,10. Surface plasmonresonans (SPR) -baserte analyser 11,12 og virkelig label-free kalori tilnærminger, for eksempel Isoterm Titrering kalorimetri (ITC) 13-15, isokratisk eluering 16, likevekt fi filtrering 17,18, in-line sondering 19, gel- skifte analyser, stopped- fl ow fl uorescence spektroskopi 20,21, fluorescens anisotropi (FA) 22,23, single-molekyl fl uorescence bildebehandling 24,25, og Bio-lag interferometri (BLI) 26 er også enten upresis eller uforenlig med aptamer-lite molekyl interaksjoner. andre Principal utgaver av disse metodene er lav følsomhet, høy prøven forbruk, immobilisering, massetransportbegrensninger på overflater, og / eller buffer restriksjoner. Bare et fåtall av disse teknologiene gir integrerte kontroller for aggregering og adsorpsjon effekter.
MST representerer et kraftig verktøy for forskere å overvinne denne begrensningen for å studere interaksjoner mellom aptamerer og små molekyler 27-29, samt andre mål, for eksempel proteiner 30-33. Teknologien baserer seg på bevegelse av molekyler gjennom temperaturgradienter. Dette rettet bevegelse, kalt "termodiffusjon", avhenger av størrelse, ladning, og hydrering skall av molekylet 34,35. Binding av en ligand til molekylet vil direkte endre i det minste én av disse parametre, noe som resulterer i en endret termoforetiske mobilitet. Ligander med små størrelser kan ikke ha store konsekvenser i forhold til størrelse endring fra ubundet til bundet tilstand, men de kan ha dr amatic effekter på hydrering skall og / eller kostnad. Endringene i den termoforetiske bevegelighet av molekylene etter interaksjon med bindingspartneren muliggjør kvantifisering av grunnleggende bindingsparametere 2,7,34,36,37.
Som vist i figur 1A, MST enheten består av en infrarød laser fokusert på prøven innenfor glasskapillærer ved hjelp av de samme optikk som for fluorescensdeteksjon. Den termo bevegelse av proteiner via den iboende fl uorescence av tryptofaner 6 eller av et fluorescensmerket interaksjon partner 3,8 kan overvåkes mens laseren etablerer en temperaturgradient (aT på 2-6 ° C). Den resulterende temperaturforskjell på plass, AT, fører til uttømming, eller oppsamling av molekyler i området av forhøyet temperatur, som kan kvantifiseres ved Soret COEF fi sient (S T):
g "/>
c varm utgjør den konsentrasjon i det oppvarmede område, og c kulde er konsentrasjonen i den innledende kald region.
Som vist i figur 1B, en typisk MST eksperimentresultatene i en MST bevegelse profil (tid kurve), som består av forskjellige faser, som kan separeres ved deres respektive tidsrammer. Den innledende fluorescensen måles i de første 5 s i fravær av temperaturgradienten for å definere den nøyaktige utgangs fluorescensen og for å se etter fotobleking eller photoenhancement. Temperatur Jump (T-Jump) representerer den fasen hvor fluorescens endringene før termo bevegelse. Denne første nedgang i fluorescens er avhengig av varmeavhengige endringer av fl uorophore kvanteutbytte. Den termodiffusjon fase følger, hvor fluorescens-lavere (eller høyere) på grunn av den termoforetiske bevegelighet av molekylene inntil steady-state fordeling er oppnådd.Det motsatte TJump og samtidig tilbakediffusjon av fl uorescent molekyler kan observeres som vist i figur 1B etter at laseren er skrudd av. For å få tilgang til basisbindingsparametere, blir forskjellige molare forhold av interaksjonspartnere analysert og sammenlignet. Vanligvis er 16 forskjellige forhold studert i en MST eksperiment, mens den optiske synlig molekylet holdes konstant og er utstyrt med en økende mengde av umerket ligand. Samspillet mellom de to bindingspartnere induserer endringer i termodiffusjon, og således i den normaliserte fl uorescence, F norm, som beregnes som følger:
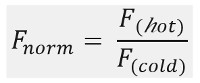
F varm og F kald representerer gjennomsnitt fl uorescence intensiteter på de fi nert tidspunkter av MST spor. Bindingsaffiniteter (K d eller EC 50 verdier) kan beregnes ved curve montering (figur 1C).
Samlet er MST et kraftig verktøy for å studere molekylære interaksjoner av noe slag. Dette manuskriptet har en protokoll for å karakterisere utfordrende samspill mellom den lille molekylet adenosintrifosfat (ATP, 0,5 kDa) og 25-nt kort ssDNA aptamer DH25.42 (7,9 kDa). I løpet av manuskriptet er bindingssetet for aptamer på den ATP-molekylet tilordnet ned til adenin gruppen av ATP.
Protocol
Representative Results
Discussion
Kvalitetskontroller:
Uspesifikke klebing / adsorpsjon av prøvemateriale til overflatene, i tillegg til aggregering effekter, ha en dramatisk innvirkning på kvaliteten av de data affinitet. Men bare noen få state-of-the-art teknologi tilbyr nøyaktige og raske alternativer for å overvåke og unngå disse effektene. MST tilbyr integrerte kvalitetskontroller som gjenkjenner og bidrar til å overvinne disse problemene, slik at for trinnvis optimalisering av tekniske oppsettet. Viktig informasjo…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Forfatterne har ingen bekreftelser.
Materials
| Aptamer binding buffer | 20 mM Tris pH7.6; 300 mM NaCl; 5 mM MgCl2; 0.01%Tween-20 | ||
| Fluorescently labeled ATP aptamer | IDT, Leuven, Belgium | sequence: DH25.42 50-Cy5-CCTGGGGGAGT-ATTGCGGAGGAAGG-3 | |
| ATP | Sigma Aldrich, Germany | A2383 | 10 mM stock solutions stored at – 20 °C |
| ADP | Sigma Aldrich, Germany | A2754 | 10 mM stock solutions stored at – 20 °C |
| AMP | Sigma Aldrich, Germany | A2252 | 10 mM stock solutions stored at – 20 °C |
| Adenine | Sigma Aldrich, Germany | A8626 | 10 mM stock solutions stored at – 20 °C |
| SAM | Sigma Aldrich, Germany | A7007 | 10 mM stock solutions stored at – 20 °C |
| dATP | Sigma Aldrich, Germany | 11934511001 | 10 mM stock solutions stored at – 20 °C |
| CTP | Sigma Aldrich, Germany | C1506 | 10 mM stock solutions stored at – 20 °C |
| GTP | Sigma Aldrich, Germany | G8877 | 10 mM stock solutions stored at – 20 °C |
| Monolith NT.115 | NanoTemper Technologies, Munich, Germany | MO-G008 | Blue/Red Channel MST device with standard detector, Monolith NT115 pico is MST device with high sensitivity detector |
| Monolith NT.115 capillaries Standard | NanoTemper Technologies, Munich, Germany | MO-K002 | |
| Eppendorf PCR tubes | Eppendorf, Germany | 30124537 | |
| Monolith control software. 2.1.33, pre-installed on the device | NanoTemper Technologies, Munich, Germany | ||
| MO.affinity analysis v2.1.1 | NanoTemper Technologies, Munich, Germany | ||
| Kaleidagraph 4.5.2 | Synergy Software |
References
- Linke, P., et al. An Automated Microscale Thermophoresis Screening Approach for Fragment-Based Lead Discovery. J Biomol Screen. 21 (4), 414-421 (2015).
- Jerabek-Willemsen, M., et al. MicroScale Thermophoresis: Interaction analysis and beyond. Journal of Molecular Structure. 1077, 101-113 (2014).
- Zillner, K., et al. Microscale thermophoresis as a sensitive method to quantify protein: nucleic acid interactions in solution. Methods Mol Biol. 815, 241-252 (2012).
- Zhang, W., Duhr, S., Baaske, P., Laue, E. Microscale thermophoresis for the assessment of nuclear protein-binding affinities. Methods Mol Biol. 1094, 269-276 (2014).
- Wienken, C. J., Baaske, P., Rothbauer, U., Braun, D., Duhr, S. Protein-binding assays in biological liquids using microscale thermophoresis. Nat Commun. 1, 100 (2010).
- Seidel, S. A., et al. Label-free microscale thermophoresis discriminates sites and affinity of protein-ligand binding. Angew Chem Int Ed Engl. 51 (42), 10656-10659 (2012).
- Seidel, S. A., et al. Microscale thermophoresis quantifies biomolecular interactions under previously challenging conditions. Methods. 59 (3), 301-315 (2013).
- Schubert, T., et al. Df31 protein and snoRNAs maintain accessible higher-order structures of chromatin. Mol Cell. 48 (3), 434-444 (2012).
- McKeague, M., Derosa, M. C. Challenges and opportunities for small molecule aptamer development. J Nucleic Acids. 2012, 748913 (2012).
- Ruscito, A., DeRosa, M. C. Small-Molecule Binding Aptamers: Selection Strategies, Characterization, and Applications. Front Chem. 4, 14 (2016).
- Chang, A. L., McKeague, M., Liang, J. C., Smolke, C. D. Kinetic and equilibrium binding characterization of aptamers to small molecules using a label-free, sensitive, and scalable platform. Anal Chem. 86 (7), 3273-3278 (2014).
- Chang, A. L., McKeague, M., Smolke, C. D. Facile characterization of aptamer kinetic and equilibrium binding properties using surface plasmon resonance. Methods Enzymol. 549, 451-466 (2014).
- Jing, M., Bowser, M. T. Methods for measuring aptamer-protein equilibria: a review. Anal Chim Acta. 686 (1-2), 9-18 (2011).
- Sokoloski, J. E., Dombrowski, S. E., Bevilacqua, P. C. Thermodynamics of ligand binding to a heterogeneous RNA population in the malachite green aptamer. Biochemistry. 51 (1), 565-572 (2012).
- Burnouf, D., et al. kinITC: a new method for obtaining joint thermodynamic and kinetic data by isothermal titration calorimetry. J Am Chem Soc. 134 (1), 559-565 (2012).
- Mannironi, C., Scerch, C., Fruscoloni, P., Tocchini-Valentini, G. P. Molecular recognition of amino acids by RNA aptamers: the evolution into an L-tyrosine binder of a dopamine-binding RNA motif. RNA. 6 (4), 520-527 (2000).
- Jenison, R. D., Gill, S. C., Pardi, A., Polisky, B. High-resolution molecular discrimination by RNA. Science. 263 (5152), 1425-1429 (1994).
- Huizenga, D. E., Szostak, J. W. A DNA aptamer that binds adenosine and ATP. Biochemistry. 34 (2), 656-665 (1995).
- Lee, E. R., Baker, J. L., Weinberg, Z., Sudarsan, N., Breaker, R. R. An allosteric self-splicing ribozyme triggered by a bacterial second messenger. Science. 329 (5993), 845-848 (2010).
- Wickiser, J. K., Cheah, M. T., Breaker, R. R., Crothers, D. M. The kinetics of ligand binding by an adenine-sensing riboswitch. Biochemistry. 44 (40), 13404-13414 (2005).
- Jucker, F. M., Phillips, R. M., McCallum, S. A., Pardi, A. Role of a heterogeneous free state in the formation of a specific RNA-theophylline complex. Biochemistry. 42 (9), 2560-2567 (2003).
- Zhao, Q., Lv, Q., Wang, H. Aptamer fluorescence anisotropy sensors for adenosine triphosphate by comprehensive screening tetramethylrhodamine labeled nucleotides. Biosens Bioelectron. 70, 188-193 (2015).
- Zhang, D., et al. A sensitive fluorescence anisotropy method for detection of lead (II) ion by a G-quadruplex-inducible DNA aptamer. Anal Chim Acta. 812, 161-167 (2014).
- Elenko, M. P., Szostak, J. W., van Oijen, A. M. Single-molecule imaging of an in vitro-evolved RNA aptamer reveals homogeneous ligand binding kinetics. J Am Chem Soc. 131 (29), 9866-9867 (2009).
- Elenko, M. P., Szostak, J. W., van Oijen, A. M. Single-molecule binding experiments on long time scales. Rev Sci Instrum. 81 (8), 083705 (2010).
- Zichel, R., Chearwae, W., Pandey, G. S., Golding, B., Sauna, Z. E. Aptamers as a sensitive tool to detect subtle modifications in therapeutic proteins. PLoS One. 7 (2), 31948 (2012).
- Baaske, P., Wienken, C. J., Reineck, P., Duhr, S., Braun, D. Optical thermophoresis for quantifying the buffer dependence of aptamer binding. Angew Chem Int Ed Engl. 49 (12), 2238-2241 (2010).
- Entzian, C., Schubert, T. Studying small molecule-aptamer interactions using MicroScale Thermophoresis (MST). Methods. 97, 27-34 (2016).
- Valenzano, S., et al. Screening and Identification of DNA Aptamers to Tyramine Using in Vitro Selection and High-Throughput Sequencing. ACS Comb Sci. 18 (6), 302-313 (2016).
- Jauset Rubio, M., et al. beta-Conglutin dual aptamers binding distinct aptatopes. Anal Bioanal Chem. 408 (3), 875-884 (2016).
- Breitsprecher, D., et al. Aptamer Binding Studies Using MicroScale Thermophoresis. Methods Mol Biol. 1380, 99-111 (2016).
- Stoltenburg, R., Schubert, T., Strehlitz, B. In vitro Selection and Interaction Studies of a DNA Aptamer Targeting Protein A. PLoS One. 10 (7), 0134403 (2015).
- Kinghorn, A. B., et al. Aptamer Affinity Maturation by Resampling and Microarray Selection. Anal Chem. 88 (14), 6981-6985 (2016).
- Duhr, S., Braun, D. Why molecules move along a temperature gradient. Proc Natl Acad Sci U S A. 103 (52), 19678-19682 (2006).
- Braun, D., Libchaber, A. Trapping of DNA by thermophoretic depletion and convection. Phys Rev Lett. 89 (18), 188103 (2002).
- Duhr, S., Arduini, S., Braun, D. Thermophoresis of DNA determined by microfluidic fluorescence. Eur Phys J E Soft Matter. 15 (3), 277-286 (2004).
- Jerabek-Willemsen, M., Wienken, C. J., Braun, D., Baaske, P., Duhr, S. Molecular interaction studies using microscale thermophoresis. Assay Drug Dev Technol. 9 (4), 342-353 (2011).
- He, K., Dragnea, V., Bauer, C. E. Adenylate Charge Regulates Sensor Kinase CheS3 To Control Cyst Formation in Rhodospirillum centenum. MBio. 6 (3), 00546 (2015).
- Brvar, M., et al. Structure-based discovery of substituted 4,5′-bithiazoles as novel DNA gyrase inhibitors. J Med Chem. 55 (14), 6413-6426 (2012).
- Pogorelcnik, B., et al. 4,6-Substituted-1,3,5-triazin-2(1H)-ones as monocyclic catalytic inhibitors of human DNA topoisomerase IIalpha targeting the ATP binding site. Bioorg Med Chem. 23 (15), 4218-4229 (2015).
- Jhaveri, S., Rajendran, M., Ellington, A. D. In vitro selection of signaling aptamers. Nat Biotechnol. 18 (12), 1293-1297 (2000).
- Khavrutskii, L., et al. Protein purification-free method of binding affinity determination by microscale thermophoresis. J Vis Exp. (78), (2013).
- Ramakrishnan, M., et al. Probing cocaine-antibody interactions in buffer and human serum. PLoS One. 7 (7), 40518 (2012).
- Chen, M., et al. Antiviral activity and interaction mechanisms study of novel glucopyranoside derivatives. Bioorg Med Chem Lett. 25 (18), 3840-3844 (2015).
- Wan, C., et al. Insights into the molecular recognition of the granuphilin C2A domain with PI(4,5)P2. Chem Phys Lipids. 186 (4,5), 61-67 (2015).
- Harazi, A., et al. The Interaction of UDP-N-Acetylglucosamine 2-Epimerase/N-Acetylmannosamine Kinase (GNE) and Alpha-Actinin 2 Is Altered in GNE Myopathy M743T Mutant. Mol Neurobiol. , (2016).

