Mikro Termoforesis kullanarak ATP bir aptamer Bağlama Sitesi Haritalama
Summary
MicroScale Thermophoresis (MST) is a sensitive technology to characterize aptamer-target interactions. This manuscript describes an MST protocol to characterize aptamer-small molecule interactions.
Abstract
Characterization of molecular interactions in terms of basic binding parameters such as binding affinity, stoichiometry, and thermodynamics is an essential step in basic and applied science. MicroScale Thermophoresis (MST) is a sensitive biophysical method to obtain this important information. Relying on a physical effect called thermophoresis, which describes the movement of molecules through temperature gradients, this technology allows for the fast and precise determination of binding parameters in solution and allows the free choice of buffer conditions (from buffer to lysates/sera). MST uses the fact that an unbound molecule displays a different thermophoretic movement than a molecule that is in complex with a binding partner. The thermophoretic movement is altered in the moment of molecular interaction due to changes in size, charge, and hydration shell. By comparing the movement profiles of different molecular ratios of the two binding partners, quantitative information such as binding affinity (pM to mM) can be determined. Even challenging interactions between molecules of small sizes, such as aptamers and small compounds, can be studied by MST. Using the well-studied model interaction between the DH25.42 DNA aptamer and ATP, this manuscript provides a protocol to characterize aptamer-small molecule interactions. This study demonstrates that MST is highly sensitive and permits the mapping of the binding site of the 7.9 kDa DNA aptamer to the adenine of ATP.
Introduction
moleküller arasındaki etkileşim doğasının temelidir. Bu nedenle, temel ve uygulamalı araştırma birçok alanda bilim farklı moleküler etkileşimlerin temel ilkelerini anlamaya çalışın. Mikro Termoforesis (MST) tamponların bir özgür seçim ile, çözelti içinde moleküler etkileşimlerin hızlı, hassas, düşük maliyetli ve kalite kontrollü karakterizasyonu gerçekleştirmek için bilim adamları sağlar. Birden fazla bağlanma ortakları 1-8 ile olay doğrulamaları, rekabet deneyleri ve deneyleri bağlayıcı, kütüphane taramaları da dahil olmak üzere analizler farklı türde anlatan, yalnız 2016 den, MST kullanarak 1000'den fazla yayın zaten vardır. Genel olarak, MST, moleküler etkileşimin herhangi bir bağlanma afinitesi (mM'ye pM), stokiyometri ve termodinamik, klasik bağlanma parametreleri, çalışmasına izin verir. MST büyük bir avantajı etkileşimi mümkün boyutundan bağımsız bağlayıcı olayları incelemek için yeteneğidir. hatta çal'ıBu küçük moleküller, ilaçlar, antibiyotikler, veya metabolitler olarak zorlu bir yöntem, küçük nükleik asit aptamerler arasındaki etkileşimler (nt 15-30) ve hedefler belirlenebilir.
Güncel state-of-the-art teknolojileri aptamer hedef etkileşimleri laboratuvar yoğun ve son derece karmaşık ya vardır karakterize ya da aptamer-küçük molekül 9,10 etkileşimleri ölçmek için başarısız. Plasmon Rezonans (SPR) deneyleri 11,12 ve İzotermal Titrasyon Kalorimetre (ITC) 13-15, izokratik elüsyon 16, denge fi ltration 17,18 olarak gerçekten etiket içermeyen Kalorimetre yaklaşımları, tabanlı Surface, in-line 19 Jel- sondalama deneyleri vardiya, Durduğunuz akış fl uorescence spektroskopisi 20,21, floresan anizotropi (FA) 22,23, tek-molekül fl uorescence görüntüleme 24,25 ve Bio-katmanlı enterforemetre (BLI) 26 de aptamer-küçük molekül ile kesin olmayan veya uyumsuz ya vardır etkileşimleri. diğer principaBu yöntemlerin l sorunları düşük duyarlılık, yüksek numune tüketimi, hareketsizlik, yüzeylerde toplu taşıma sınırlamalar ve / veya tampon kısıtlamaları vardır. Bu teknolojilerin sadece birkaç toplama ve adsorpsiyon efektleri için entegre kontroller sağlar.
MST bilim adamları bu tür proteinlerin 30-33 olarak aptamers ve küçük moleküllerin 27-29 arasındaki etkileşimleri, yanı sıra diğer hedefleri incelemek için bu sınırlamayı aşmak için için güçlü bir araç temsil eder. Teknoloji sıcaklık değişimleri ile moleküllerin hareketine dayanır. Olarak adlandırılan bu yönlendirilmiş hareket, "Termoforosis," molekülün 34,35 büyüklüğüne, ücret ve hidrasyon kabuğu bağlıdır. moleküle bir ligand bağlanmasının doğrudan değiştirildi thermophoretic hareket ile sonuçlanan, aşağıdaki parametrelerden en azından birinin değiştirecektir. Küçük boyutları ile ligandlar bağlı duruma bağlanmamış gelen boyut değişim açısından önemli bir etkiye sahip olmayabilir, ama onlar dr olabilir hidrasyon kabuğu ve / veya şarj amatic etkiler. Bağlama ortağı ile etkileşim sonrası moleküllerin thermophoretic hareketinde değişiklik temel bağlanma parametreleri 2,7,34,36,37 ölçülmesini sağlar.
Şekil 1A 'de gösterildiği gibi, MST cihazı flüoresan tespiti için aynı optik kullanılarak cam kılcal olan numune üzerine odaklanmış bir kızıl ötesi lazer oluşur. Lazer sıcaklık gradyanı (2-6 ° C DT) kurarken tryptophans 6 ya da flüoresan bir etkileşim, ortak olarak 3,8 arasında içsel fl uorescence ile proteinlerin thermophoretic hareketi izlenebilir. Boşluk, DT, içinde sıcaklık farkı Soret ile belirlenebilir yüksek bir sıcaklıkta alanında azalması veya molekül birikmesine yol açar, Katsayı (St):
g "/>
C sıcak ısıtılmış bölgede konsantrasyonunu temsil eder, ve C, soğuk ilk olarak soğuk bölge konsantrasyonudur.
Şekil 1B, ilgili zaman dilimlerinde ayrılabilir farklı aşamalarında oluşan MST hareket profilinin (zaman iz), tipik bir MST deney sonuçları gösterildiği gibi. başlangıç floresans kesin bir başlangıç floresans tanımlamak ve ışıkla ağartma ya photoenhancement kontrol etmek için sıcaklık gradyanı yokluğunda yer alan birinci 5 saniye cinsinden ölçülür. Sıcaklık Jump (T-Jump) hangi floresan değişiklikleri thermophoretic hareketinden önce safhasını oluşturuyor. floresanstaki bu ilk azalma kuantum verimi uorophore fl ısıya bağlı değişikliklere bağlıdır. Termoforosis aşaması nedeniyle sabit durum dağılım kadar molekül thermophoretic hareketine flüoresans azalır (veya artar) ulaşıldığında edildiği izler.Lazer kapatıldıktan sonra Şekil 1B gösterildiği gibi ters TJump ve BM uorescent moleküllerin beraberinde geri difüzyon gözlenebilir. Temel bağlanma parametreleri erişmek için, etkileşim ortakları farklı molar oranları incelenmiş ve karşılaştırılmıştır. Optik görebilir molekülü sabit tutulur ve etiketlenmemiş ligandın artan bir miktarı ile temin edilir ise tipik olarak, 16 farklı oranlar bir MST deneyde incelenmiştir. İki bağlanma ortakları arasındaki etkileşim Termoforosis değişiklikleri uyarır ve bu nedenle normalize fl uorescence olarak aşağıdaki şekilde hesaplanır F norm:
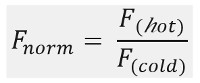
F sıcak ve F soğuk MST izleri de fi ned zaman noktalarında fl uorescence yoğunlukları ortalama temsil eder. Bağlanma ilgileri (Kd ve EC50 değerleri) Curv hesaplanabilire uydurma (Şekil 1C).
Genel olarak, MST her türlü moleküler etkileşimleri çalışmak için güçlü bir araçtır. ve 25-nt kısa ssDNA aptamer DH25.42 (7.9 kDa); Bu yazının küçük molekül adenozin trifosfat (0.5 kDa ATP) arasındaki zorlu etkileşimi karakterize bir protokol sunar. Yazının boyunca ATP molekülü üzerindeki aptamer bağlanma yeri ATP adenin grubuna aşağı eşleştirilir.
Protocol
Representative Results
Discussion
Kalite kontrolleri:
Spesifik olmayan yapışma / yüzeylere örnek malzeme, hem de çekiş etkileri adsorpsiyon, afinite verinin kalitesi üzerinde önemli bir etkisi vardır. Ancak, sadece bir kaç state-of-the-art teknolojileri izlemek ve bu etkilerden kaçınmak için doğru ve hızlı seçenekler sunuyoruz. MST algılar ve teknik kurulum adım adım optimizasyonu sağlayan, bu sorunların üstesinden gelmek için yardımcı entegre kalite kontrolleri sunmaktadır. toplama / yağış etkiler…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Yazarlar hiçbir onayları var.
Materials
| Aptamer binding buffer | 20 mM Tris pH7.6; 300 mM NaCl; 5 mM MgCl2; 0.01%Tween-20 | ||
| Fluorescently labeled ATP aptamer | IDT, Leuven, Belgium | sequence: DH25.42 50-Cy5-CCTGGGGGAGT-ATTGCGGAGGAAGG-3 | |
| ATP | Sigma Aldrich, Germany | A2383 | 10 mM stock solutions stored at – 20 °C |
| ADP | Sigma Aldrich, Germany | A2754 | 10 mM stock solutions stored at – 20 °C |
| AMP | Sigma Aldrich, Germany | A2252 | 10 mM stock solutions stored at – 20 °C |
| Adenine | Sigma Aldrich, Germany | A8626 | 10 mM stock solutions stored at – 20 °C |
| SAM | Sigma Aldrich, Germany | A7007 | 10 mM stock solutions stored at – 20 °C |
| dATP | Sigma Aldrich, Germany | 11934511001 | 10 mM stock solutions stored at – 20 °C |
| CTP | Sigma Aldrich, Germany | C1506 | 10 mM stock solutions stored at – 20 °C |
| GTP | Sigma Aldrich, Germany | G8877 | 10 mM stock solutions stored at – 20 °C |
| Monolith NT.115 | NanoTemper Technologies, Munich, Germany | MO-G008 | Blue/Red Channel MST device with standard detector, Monolith NT115 pico is MST device with high sensitivity detector |
| Monolith NT.115 capillaries Standard | NanoTemper Technologies, Munich, Germany | MO-K002 | |
| Eppendorf PCR tubes | Eppendorf, Germany | 30124537 | |
| Monolith control software. 2.1.33, pre-installed on the device | NanoTemper Technologies, Munich, Germany | ||
| MO.affinity analysis v2.1.1 | NanoTemper Technologies, Munich, Germany | ||
| Kaleidagraph 4.5.2 | Synergy Software |
References
- Linke, P., et al. An Automated Microscale Thermophoresis Screening Approach for Fragment-Based Lead Discovery. J Biomol Screen. 21 (4), 414-421 (2015).
- Jerabek-Willemsen, M., et al. MicroScale Thermophoresis: Interaction analysis and beyond. Journal of Molecular Structure. 1077, 101-113 (2014).
- Zillner, K., et al. Microscale thermophoresis as a sensitive method to quantify protein: nucleic acid interactions in solution. Methods Mol Biol. 815, 241-252 (2012).
- Zhang, W., Duhr, S., Baaske, P., Laue, E. Microscale thermophoresis for the assessment of nuclear protein-binding affinities. Methods Mol Biol. 1094, 269-276 (2014).
- Wienken, C. J., Baaske, P., Rothbauer, U., Braun, D., Duhr, S. Protein-binding assays in biological liquids using microscale thermophoresis. Nat Commun. 1, 100 (2010).
- Seidel, S. A., et al. Label-free microscale thermophoresis discriminates sites and affinity of protein-ligand binding. Angew Chem Int Ed Engl. 51 (42), 10656-10659 (2012).
- Seidel, S. A., et al. Microscale thermophoresis quantifies biomolecular interactions under previously challenging conditions. Methods. 59 (3), 301-315 (2013).
- Schubert, T., et al. Df31 protein and snoRNAs maintain accessible higher-order structures of chromatin. Mol Cell. 48 (3), 434-444 (2012).
- McKeague, M., Derosa, M. C. Challenges and opportunities for small molecule aptamer development. J Nucleic Acids. 2012, 748913 (2012).
- Ruscito, A., DeRosa, M. C. Small-Molecule Binding Aptamers: Selection Strategies, Characterization, and Applications. Front Chem. 4, 14 (2016).
- Chang, A. L., McKeague, M., Liang, J. C., Smolke, C. D. Kinetic and equilibrium binding characterization of aptamers to small molecules using a label-free, sensitive, and scalable platform. Anal Chem. 86 (7), 3273-3278 (2014).
- Chang, A. L., McKeague, M., Smolke, C. D. Facile characterization of aptamer kinetic and equilibrium binding properties using surface plasmon resonance. Methods Enzymol. 549, 451-466 (2014).
- Jing, M., Bowser, M. T. Methods for measuring aptamer-protein equilibria: a review. Anal Chim Acta. 686 (1-2), 9-18 (2011).
- Sokoloski, J. E., Dombrowski, S. E., Bevilacqua, P. C. Thermodynamics of ligand binding to a heterogeneous RNA population in the malachite green aptamer. Biochemistry. 51 (1), 565-572 (2012).
- Burnouf, D., et al. kinITC: a new method for obtaining joint thermodynamic and kinetic data by isothermal titration calorimetry. J Am Chem Soc. 134 (1), 559-565 (2012).
- Mannironi, C., Scerch, C., Fruscoloni, P., Tocchini-Valentini, G. P. Molecular recognition of amino acids by RNA aptamers: the evolution into an L-tyrosine binder of a dopamine-binding RNA motif. RNA. 6 (4), 520-527 (2000).
- Jenison, R. D., Gill, S. C., Pardi, A., Polisky, B. High-resolution molecular discrimination by RNA. Science. 263 (5152), 1425-1429 (1994).
- Huizenga, D. E., Szostak, J. W. A DNA aptamer that binds adenosine and ATP. Biochemistry. 34 (2), 656-665 (1995).
- Lee, E. R., Baker, J. L., Weinberg, Z., Sudarsan, N., Breaker, R. R. An allosteric self-splicing ribozyme triggered by a bacterial second messenger. Science. 329 (5993), 845-848 (2010).
- Wickiser, J. K., Cheah, M. T., Breaker, R. R., Crothers, D. M. The kinetics of ligand binding by an adenine-sensing riboswitch. Biochemistry. 44 (40), 13404-13414 (2005).
- Jucker, F. M., Phillips, R. M., McCallum, S. A., Pardi, A. Role of a heterogeneous free state in the formation of a specific RNA-theophylline complex. Biochemistry. 42 (9), 2560-2567 (2003).
- Zhao, Q., Lv, Q., Wang, H. Aptamer fluorescence anisotropy sensors for adenosine triphosphate by comprehensive screening tetramethylrhodamine labeled nucleotides. Biosens Bioelectron. 70, 188-193 (2015).
- Zhang, D., et al. A sensitive fluorescence anisotropy method for detection of lead (II) ion by a G-quadruplex-inducible DNA aptamer. Anal Chim Acta. 812, 161-167 (2014).
- Elenko, M. P., Szostak, J. W., van Oijen, A. M. Single-molecule imaging of an in vitro-evolved RNA aptamer reveals homogeneous ligand binding kinetics. J Am Chem Soc. 131 (29), 9866-9867 (2009).
- Elenko, M. P., Szostak, J. W., van Oijen, A. M. Single-molecule binding experiments on long time scales. Rev Sci Instrum. 81 (8), 083705 (2010).
- Zichel, R., Chearwae, W., Pandey, G. S., Golding, B., Sauna, Z. E. Aptamers as a sensitive tool to detect subtle modifications in therapeutic proteins. PLoS One. 7 (2), 31948 (2012).
- Baaske, P., Wienken, C. J., Reineck, P., Duhr, S., Braun, D. Optical thermophoresis for quantifying the buffer dependence of aptamer binding. Angew Chem Int Ed Engl. 49 (12), 2238-2241 (2010).
- Entzian, C., Schubert, T. Studying small molecule-aptamer interactions using MicroScale Thermophoresis (MST). Methods. 97, 27-34 (2016).
- Valenzano, S., et al. Screening and Identification of DNA Aptamers to Tyramine Using in Vitro Selection and High-Throughput Sequencing. ACS Comb Sci. 18 (6), 302-313 (2016).
- Jauset Rubio, M., et al. beta-Conglutin dual aptamers binding distinct aptatopes. Anal Bioanal Chem. 408 (3), 875-884 (2016).
- Breitsprecher, D., et al. Aptamer Binding Studies Using MicroScale Thermophoresis. Methods Mol Biol. 1380, 99-111 (2016).
- Stoltenburg, R., Schubert, T., Strehlitz, B. In vitro Selection and Interaction Studies of a DNA Aptamer Targeting Protein A. PLoS One. 10 (7), 0134403 (2015).
- Kinghorn, A. B., et al. Aptamer Affinity Maturation by Resampling and Microarray Selection. Anal Chem. 88 (14), 6981-6985 (2016).
- Duhr, S., Braun, D. Why molecules move along a temperature gradient. Proc Natl Acad Sci U S A. 103 (52), 19678-19682 (2006).
- Braun, D., Libchaber, A. Trapping of DNA by thermophoretic depletion and convection. Phys Rev Lett. 89 (18), 188103 (2002).
- Duhr, S., Arduini, S., Braun, D. Thermophoresis of DNA determined by microfluidic fluorescence. Eur Phys J E Soft Matter. 15 (3), 277-286 (2004).
- Jerabek-Willemsen, M., Wienken, C. J., Braun, D., Baaske, P., Duhr, S. Molecular interaction studies using microscale thermophoresis. Assay Drug Dev Technol. 9 (4), 342-353 (2011).
- He, K., Dragnea, V., Bauer, C. E. Adenylate Charge Regulates Sensor Kinase CheS3 To Control Cyst Formation in Rhodospirillum centenum. MBio. 6 (3), 00546 (2015).
- Brvar, M., et al. Structure-based discovery of substituted 4,5′-bithiazoles as novel DNA gyrase inhibitors. J Med Chem. 55 (14), 6413-6426 (2012).
- Pogorelcnik, B., et al. 4,6-Substituted-1,3,5-triazin-2(1H)-ones as monocyclic catalytic inhibitors of human DNA topoisomerase IIalpha targeting the ATP binding site. Bioorg Med Chem. 23 (15), 4218-4229 (2015).
- Jhaveri, S., Rajendran, M., Ellington, A. D. In vitro selection of signaling aptamers. Nat Biotechnol. 18 (12), 1293-1297 (2000).
- Khavrutskii, L., et al. Protein purification-free method of binding affinity determination by microscale thermophoresis. J Vis Exp. (78), (2013).
- Ramakrishnan, M., et al. Probing cocaine-antibody interactions in buffer and human serum. PLoS One. 7 (7), 40518 (2012).
- Chen, M., et al. Antiviral activity and interaction mechanisms study of novel glucopyranoside derivatives. Bioorg Med Chem Lett. 25 (18), 3840-3844 (2015).
- Wan, C., et al. Insights into the molecular recognition of the granuphilin C2A domain with PI(4,5)P2. Chem Phys Lipids. 186 (4,5), 61-67 (2015).
- Harazi, A., et al. The Interaction of UDP-N-Acetylglucosamine 2-Epimerase/N-Acetylmannosamine Kinase (GNE) and Alpha-Actinin 2 Is Altered in GNE Myopathy M743T Mutant. Mol Neurobiol. , (2016).

