디지털 홀로그램 현미경 (DHM)를 사용 하 여 낮은 농도에서 미생물 측정
Summary
디지털 홀로그램 현미경 (DHM) 이미징 샘플 50-100 X 초점 비교 해상도, 명시 야 현미경 보다 두꺼운 수행 후 처리를 허용 하는 체적 기술입니다. 여기 DHM 사용 됩니다 식별, 계산, 그리고 미생물에서 매우 낮은 밀도 추적과 광학 밀도 측정, 판 수, 및 직접 카운트와 비교.
Abstract
정확 하 게 감지 하 고 스파스 세균 샘플 세 의료 진단, 및 로봇 식 임무를 다른 행 성과 태양계의 위성에 의해 생활 탐지에 대 한 음식, 음료, 제약 공업, 많은 응용 프로그램 있다. 현재, 스파스 세균 샘플은 문화 도금 또는 epifluorescence 현미경 검사 법에 의해 계산 됩니다. 문화 접시 필요 오래 부 화 시간 (주 일) 하며 epifluorescence 현미경 광범위 한 얼룩 및 샘플의 농도. 여기, 꺼짐-축 디지털 홀로그램 현미경 (DHM)를 사용 하 여 매우 희석 문화 (100-104 셀/mL)에 박테리아를 열거 하는 방법을 보여 줍니다. 첫째, 사용자 지정 DHM의 건설 건물 저가 악기에 자세한 지침과 함께 설명 되어 있습니다. 홀로 그래피의 원리를 설명 하 고 통계 모델 동영상 해야 얼마나 오래 추정 하는 데 사용은 악기의 광학 성능 특성 및 세균 솔루션 (표 2)의 농도에 따라 셀을 검출 하 . 10 셀의 비디오 검색5, 104, 103및 100 셀/mL 유엔 재건된 홀로그램을 사용 하 여 실시간으로에서 설명 된다. 진폭 및 위상 이미지의 개조는 오픈 소스 소프트웨어 패키지를 사용 하 여 보여 줍니다.
Introduction
매우 희석 샘플에 정확한 세균의 결정은 많은 응용 프로그램에서 중요 한: 몇 가지 예는 물과 식품 품질 분석1,,23; 혈액, 중추 신 경계, 또는 래4,5;에 병원 균의 검출 메 마른 물6;를 포함 하 여 제약 제품의 생산 그리고 오픈 바다와 퇴적 물7,,89같은 oligotrophic 환경에서 환경 지역 사회 분석. 또한 목성과 토성, 특히 유로 파10,11 , Enceladus12,13, 의 얼음 위성에 가능한 현존 미생물 생명의 검출에 대 한 관심 증가 14, 표면 액체 바다를 알려져 있습니다. 1978 년에서 바이킹 이후 아무 미션이 다른 행성에 현존 생활을 찾을 하려고, 때문에 기술 및 세균성 식별 및 공간 임무15동안 계산을 위한 계기의 한정 된 개발이 되었습니다.
판 수의 전통적인 방법의 찾을 culturable 셀만, 때로는 환경 긴장에 종의 소수를 표현할 수 있는 < 116. 플레이트 일 또는 외피의 주 변형에 따라 최대 성공을 위해 필요 합니다. Epifluorescence 현미경 검사 법은 신속 하 고 정확한 미생물 열거형에 대 한 황금 표준으로 크게 접시 계산을 대체 했다. 핵 산 라벨 형광 염료 4, 6-diamidino-2-phenylindole dihydrochloride (DAPI), SYBR 녹색 또는 acridine 오렌지 같은 핵 산을 바인딩하는 일반적인 염료 사용17,,1819 하지만 많은 연구를 사용 하 여 그램의 형광 표시기 서명20,,2122,,2324. ML 당 10 ~5 셀의 검출 (Lod)의 한계를 이끌어 전 농도 단계 없이 이러한 방법을 사용 하 여. LoD 향상 된 여과 사용 하 여 가능 하다입니다. 액체 샘플 막, 일반적으로 폴 리 카보 네이트를 이상적으로 줄이기 위해 배경 검정에 진공 필터링입니다. 저 배경 염료와 같은 위에서 언급 한 DNA 얼룩 필터25에 직접 적용할 수 있습니다. 눈으로 정확한 계산에 대 한 10 ~5 셀 필터, mL 당 10 ~5 셀 보다 더 희석 샘플에 대 한 즉 당 필요한, 중요 한 샘플 볼륨을 수집 하 고 필터링 해야 합니다. 레이저 검사 장치 필터의 모든 영역을 체계적으로 탐구 하 고 따라서 mL26당 10 ~2 셀 아래로 탐지의 한계를 밀고, 계산에 필요한 셀의 수를 줄이기 위해 개발 되었습니다. 그러나, 이들은 대부분 실험실에서 사용할 수 있습니다 그리고 정교한 하드웨어 뿐 아니라 소프트웨어 전문가 확인 입자를 관찰 하는 허용 하 고 박테리아와 파편 하지.
참고로, 패 혈 증으로 성인 보통 시작에서 증상을 보여주는 < 셀/100ml의 혈액, 및에서 유아 < 10 셀/mL. 성인에서 혈액 무승부 소요 10 mL, 유아, 1 mL에서. PCR 기반 방법 인간과 비 병원 성 식물 DNA의 존재에 의해 및 혈액27,28구성 요소 PCR 억제 저해 됩니다. 다양 한 새로운 기술에도 불구 하 고 문화 전원 지역 또는 개발 도상 국가에서 특히 혈 류 감염의 진단에 대 한 황금 표준 남아 있습니다. 다른 행성에 생활의 탐지, 열역학 계산 생활을 따라서 예상된 가능한 바이오 매스 에너지 예산을 추정할 수 있다. 1-100 셀/mL 유로 파29에 열역학으로 합리적인 것으로 예상 된다. 그것은 쉽게 이러한 숫자에서 볼는 수성 해결책의 다량에 있는 세포의 아주 작은 숫자의 발견은 중요 한 미해결된 문제를 수 있습니다.
이 논문에서는, 10의 농도에서 Serratia marcescens 및 Shewanella oneidensis (야생 형 및 비 운동 돌연변이)의 탐지 설명5, 104, 103및 꺼짐-축을 사용 하 여 100 셀/mL 디지털 홀로그램 현미경 (DHM) DHM의 주요 장점은 전통적인 가벼운 현미경 검사 법은 높은 해상도에서 두꺼운 샘플 볼륨의 동시 영상-이 구현에서 샘플 챔버는 0.8 m m 두께. 이러한 샘플 챔버는 소프트-리소 그래피입니다 (PDMS) 정밀 가공 알루미늄 몰드에서의 허용 오차는 ± 50 µ m에 의해 건설 되었다. 이 고 전력 가벼운 현미경 검사 법을 통해 필드의 깊이에 약 100 개선을 나타냅니다. 또한 DHM 광학 경로 길이 (굴절률과 두께의 제품)의 측정에 대 한 허용 하 양적 위상 정보를 제공 합니다. DHM와 유사한 기술을 사용 되었습니다 모니터링 박테리아와 효 모 세포 주기 및 세균 건조 질량30,,3132;의 계산 산란 차이점도 세균성 긴장33을 차별화 하는 데 사용할 수 있습니다.
우리가 사용 하는 악기 이전 게시34,35, 미생물와 함께 사용 사용자 이며 그것의 디자인 및 건축 시연 및 설명. 수성 솔루션 통해 주사기 펌프; 0.25 µ L 볼륨 샘플 챔버에 지속적으로 공급 된다 흐름 율은 전체 샘플 볼륨의 이미지를 보장 하기 위해 카메라 프레임 속도 의해 결정 됩니다. 통계 계산 주어진된 농도에서 세포의 상당수를 탐지 하기 위해 몇 군데 해야 합니다 샘플 볼륨의 수를 예측 합니다.
셀-감지 응용 프로그램 진폭 및 위상 이미지에 홀로그램의 재건은 필요; 분석은 원시 홀로그램에서 수행 되었다. 이 저장 계산 리소스 및 디스크 공간: 500mb 홀로그램 비디오 1-2 결핵 때 재건 될 것입니다. 그러나, 우리는 홀로그램 원하는 종 나타내는 확인 하는 샘플의 깊이 통해 재건 논의 할. DHM의 중요 한 기능 강도 및 이미지의 단계를 모니터링 하는 기능입니다. 강도 (예: 대부분의 생물 세포)에 거의 투명 한 유기 단계에서 명확 하 게 표시 됩니다. 그것은 레이블 없는 기술, 아니 염료 사용 됩니다. 이 한가능한 우주 비행 용, 염료는 임무의 조건을 살아날 수 있습니다 이후 dvantage 및-더 중요 한 것은-외계 생물, 인코딩에 대 한 DNA 또는 RNA를 사용 하지 않을 수 있습니다 작업으로 간주 될 수 없습니다. 북극과 남극, 염료 원격 위치를 데 려 어려울 수 있습니다 및 저장 시 저하 될 수 있습니다 같은 극한 환경에서 작업에 대 한 장점 이기도 합니다. 위상 및 진폭으로 이미지의 개조는 우리가 만든 사용할 수 GitHub (샴푸)에 오픈 소스 소프트웨어 패키지를 사용 하 여 또는 ImageJ를 사용 하 여 수행 됩니다.
Protocol
Representative Results
Discussion
홀로그램의 수치 재건: 홀로그램의 숫자 재건, 각도 스펙트럼 방법 (ASM) 사용 됩니다. 이 DHM의 그린의 기능으로 홀로그램의 회선을 포함 한다. 특정 초점면에서 이미지의 복잡 한 wavefront 푸리에 회선 정리를 다음과 같이 채용 하 여 계산할 수 있다:
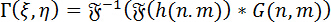 (1)
(1)
어디 <img alt="Equation 3" src="/files/ftp_upload/56343/…
Disclosures
The authors have nothing to disclose.
Acknowledgements
저자는 고 든과 베티 무어 재단 보조금 4037 4038 자금이 작품에 대 한 캘리포니아 기술 연구소를 인정 합니다.
Materials
| Bacto Yeast | BD Biosciences | 212750 | |
| Bacto Tryptone | BD Biosciences | 211705 | |
| Sodium chloride | Sigma-Aldrich | 7710 | Many options for purchase |
| Bacto Agar | BD Biosciences | 214010 | |
| 10 cm Petri dishes | VWR | 10053-704 | |
| 15 mL culture tubes | Falcon (Corning Life Sciences) | 352002 | Loose-capped |
| Petroff-Hauser chamber | Electron Microscopy Sciences | 3920 | |
| 10 mL syringes | BD Biosciences | 309604 | Luer-Lok tip not necessary |
| Male Luer to 1/16” barbed fitting | McMaster-Carr | 51525K291 | |
| Autoclavable 1/16” ID PVC tubing for flow | Nalgene | 8000-0004 | Sold by length, purchase accordingly |
| Syringe pump | Harvard Apparatus | PHD 2000 | |
| Sample Chamber | Custom | n/a | See Materials Section |
| Holographic Microscope | Custom | n/a | See Wallace et al. |
| Open-source software | Custom | https://github.com/bmorris3/shampoo |
References
- Akineden, O., Weirich, S., Abdulmawjood, A., Failing, K., Buelte, M. Application of a Fluorescence Microscopy Technique for Detecting Viable Mycobacterium avium ssp. paratuberculosis Cells in Milk. Food Anal. Methods. 8, 499-506 (2015).
- Deibl, J., Paulsen, P., Bauer, F. Rapid enumeration of total aerobic microbial counts on meat and in meat products by means of the direct epifluorescence filter technique. Wien Tierarztl Monat. 85, 327-333 (1998).
- Huang, J., Li, Y., Slavik, M. F., Tao, Y., Huff, G. R. Identification and enumeration of Salmonella on sample slides of poultry carcass wash water using image analysis with fluorescent microscopy. Transactions of the Asae. 42, 267-273 (1999).
- Durtschi, J. D., et al. Increased sensitivity of bacterial detection in cerebrospinal fluid by fluorescent staining on low-fluorescence membrane filters. J Med Microbiol. 54, 843-850 (2005).
- Panicker, R. O., Soman, B., Saini, G., Rajan, J. A Review of Automatic Methods Based on Image Processing Techniques for Tuberculosis Detection from Microscopic Sputum Smear Images. J Med Syst. 40, (2016).
- Riepl, M., et al. Applicability of solid-phase cytometry and epifluorescence microscopy for rapid assessment of the microbiological quality of dialysis water. Nephrol Dial Transplant. 26, 3640-3645 (2011).
- Hwang, C. Y., Cho, B. C. Virus-infected bacteria in oligotrophic open waters of the East Sea, Korea. Aquat Microb Ecol. 30, 1-9 (2002).
- Kepner, R. L., Pratt, J. R. Use of Fluorochromes for Direct Enumeration of Total Bacteria in Environmental-Samples – Past and Present. Microbiol Rev. 58, 603-615 (1994).
- Queric, N. V., Soltwedel, T., Arntz, W. E. Application of a rapid direct viable count method to deep-sea sediment bacteria. J Microbiol Meth. 57, 351-367 (2004).
- Prieto-Ballesteros, O., Vorobyova, E., Parro, V., Rodriguez Manfredi, J. A., Gomez, F. Strategies for detection of putative life on Europa. Adv Space Res. 48, 678-688 (2011).
- Gleeson, D. F., et al. Biosignature Detection at an Arctic Analog to Europa. Astrobiology. 12, 135-150 (2012).
- Carr, C. E., Zuber, M. T., Ruvkun, G., Ieee, . 2013 Ieee Aerospace Conference IEEE Aerospace Conference Proceedings. , (2013).
- Konstantinidis, K., et al. A lander mission to probe subglacial water on Saturn’s moon Enceladus for life. Acta Astronautica. 106, 63-89 (2015).
- McKay, C. P., Anbar, A. D., Porco, C., Tsou, P. Follow the Plume: The Habitability of Enceladus. Astrobiology. 14, 352-355 (2014).
- Hoover, R. B., Hoover, R. B., Levin, G. V., Rozanov, A. Y., Wickramasinghe, N. C. . Instruments, Methods, and Missions for Astrobiology Xvii. , (2015).
- Handelsman, J. Metagenomics: application of genomics to uncultured microorganisms. Microbiol Mol Biol Rev: MMBR. 68, 669-685 (2004).
- Lebaron, P., Parthuisot, N., Catala, P. Comparison of blue nucleic acid dyes for flow cytometric enumeration of bacteria in aquatic systems. Appl Environ Microbiol. 64, 1725-1730 (1998).
- Marie, D., Partensky, F., Jacquet, S., Vaulot, D. Enumeration and cell cycle analysis of natural populations of marine picoplankton by flow cytometry using the nucleic acid stain SYBR Green I. Appl Environ Microbiol. 63, 186-193 (1997).
- Noble, R. T., Fuhrman, J. A. Use of SYBR Green I for rapid epifluorescence counts of marine viruses and bacteria. Aquat Microb Ecol. 14, 113-118 (1998).
- Forster, S., Snape, J. R., Lappin-Scott, H. M., Porter, J. Simultaneous fluorescent gram staining and activity assessment of activated sludge bacteria. Appl Environ Microbiol. 68, 4772-4779 (2002).
- Lauer, B. A., Reller, L. B., Mirrett, S. Comparison Of Acridine-Orange And Gram Stains For Detection Of Microorganisms In Cerebrospinal-Fluid And Other Clinical Specimens. J Clin Microbiol. 14, 201-205 (1981).
- Mason, D. J., Shanmuganathan, S., Mortimer, F. C., Gant, V. A. A fluorescent gram stain for flow cytometry and epifluorescence microscopy. Appl Environ Microbiol. 64, 2681-2685 (1998).
- Saida, H., Ytow, N., Seki, H. Photometric application of the Gram stain method to characterize natural bacterial populations in aquatic environments. Appl Environ Microbiol. 64, 742-747 (1998).
- Sizemore, R. K., Caldwell, J. J., Kendrick, A. S. Alternate Gram Staining Technique Using A Fluorescent Lectin. Appl Environ Microbiol. 56, 2245-2247 (1990).
- Bitton, G., Dutton, R. J., Foran, J. A. New Rapid Technique For Counting Microorganisms Directly On Membrane Filters. Stain Technology. 58, 343-346 (1983).
- Broadaway, S. C., Barton, S. A., Pyle, B. H. Rapid staining and enumeration of small numbers of total bacteria in water by solid-phase laser cytometry. Appl Environ Microbiol. 69, 4272-4273 (2003).
- Yagupsky, P., Nolte, F. S. Quantitative aspects of septicemia. Clin Microbiol Rev. 3, 269-279 (1990).
- Kang, D. K., et al. Rapid detection of single bacteria in unprocessed blood using Integrated Comprehensive Droplet Digital Detection. Nat Commun. 5, 5427 (2014).
- Hand, K. P. . Report of the Europa Lander Science Definition Team. , (2017).
- Popescu, G., et al. Optical imaging of cell mass and growth dynamics. Am J Physiol-Cell Physiol. 295, C538-C544 (2008).
- Mir, M., et al. Optical measurement of cycle-dependent cell growth. Proc Natl Acad Sci U S A. 108, 13124-13129 (2011).
- Rappaz, B., et al. Noninvasive characterization of the fission yeast cell cycle by monitoring dry mass with digital holographic microscopy. J.Biomed Opt. 14, 034049 (2009).
- Jo, Y., et al. Label-free identification of individual bacteria using Fourier transform light scattering. Opt. Express. 23, 15792-15805 (2015).
- Wallace, J. K., et al. Robust, compact implementation of an off-axis digital holographic microscope. Opt. Express. 23, 17367-17378 (2015).
- Lindensmith, C. A., et al. A Submersible, Off-Axis Holographic Microscope for Detection of Microbial Motility and Morphology in Aqueous and Icy Environments. Plos One. 11, e0147700 (2016).
- Dumas, E. M., Ozenne, V., Mielke, R. E., Nadeau, J. L. Toxicity of CdTe Quantum Dots in Bacterial Strains. IEEE Trans. NanoBiosci. 8, 58-64 (2009).
- Frisk, A., Jyot, J., Arora, S. K., Ramphal, R. Identification and functional characterization of flgM, a gene encoding the anti-sigma 28 factor in Pseudomonas aeruginosa. J Bacteriol. 184, 1514-1521 (2002).
- Adler, J., Templeton, B. The effect of environmental conditions on the motility of Escherichia coli. Journal Gen Microbiol. 46, 175-184 (1967).
- Piedrahita-Quintero, P., Castaneda, R., Garcia-Sucerquia, J. Numerical wave propagation in Image J. Appl Opt. 54, 6410-6415 (2015).
- Schnars, U., Jüptner, W. P. Digital recording and numerical reconstruction of holograms. Meas. Sci. Technol. 13, R85 (2002).

