Kwantificeren van micro-organismen in lage concentraties, met behulp van digitale holografische microscopie (DHM)
Summary
Digitale holografische microscopie (DHM) is een volumetrische techniek waarmee imaging monsters die 50-100 X dikker dan helderveld microscopie bij vergelijkbare resolutie, met scherpstellen uitgevoerd na verwerking. Hier DHM wordt gebruikt voor het identificeren, tellen, en bijhouden van micro-organismen bij zeer lage dichtheden en vergeleken met metingen van de optische dichtheid, kiemgetal, en directe.
Abstract
Nauwkeurig detecteren en tellen sparse bacteriële monsters kent vele toepassingen in de voedingsmiddelen, dranken en farmaceutische verwerkingsindustrie, in de medische diagnostiek, en voor de detectie van het leven door robotic missies naar andere planeten en manen van het zonnestelsel. Momenteel worden schaars bacteriële monsters geteld door cultuur beplating of epifluorescence microscopie. Cultuur platen vereisen lange incubatie tijd (dagen tot weken), en epifluorescence microscopie vereist uitgebreide kleuring en concentratie van het monster. Hier laten we zien hoe off-axis digitale holografische microscopie (DHM) te gebruiken bij het inventariseren van bacteriën in zeer verdunde culturen (100-104 cellen/mL). Eerst, de bouw van de aangepaste DHM is besproken, samen met gedetailleerde instructies voor het bouwen van een low cost-instrument. De beginselen van holografie worden besproken, en een statistisch model is gebruikt om te schatten hoe lang video’s moeten worden om op te sporen cellen, op basis van de kenmerken van de optische prestaties van het instrument en de concentratie van de bacteriële oplossing (tabel 2) . Video detectie van cellen op 105, 104, 103, en 100 cellen/mL wordt gedemonstreerd in real time via un-gereconstrueerde hologrammen. Wederopbouw van amplitude en fase beelden is aangetoond door middel van een open-source softwarepakket.
Introduction
Bepaling van nauwkeurige bacteriële graven in zeer verdunde monsters is van cruciaal belang in vele toepassingen: een paar voorbeelden zijn water en voedsel kwaliteit analyse1,2,3; opsporing van ziekteverwekkers in het bloed, cerebrospinaal vocht of sputum4,5; productie van farmaceutische producten, met inbegrip van steriel water6; en milieu Gemeenschap analyse in oligotrofe omgevingen zoals de open oceaan en sedimenten7,8,9. Er is ook steeds meer belangstelling in opsporing van mogelijke bestaande microbiële leven op de ijzige manen van Jupiter en Saturnus, met name Europa10,11 en Enceladus12,13, 14, waarvan bekend is dat hebben ondergrond vloeibare oceanen. Omdat er geen missie sinds Viking in 1978 heeft geprobeerd te vinden van overgeleverde leven op een andere planeet, is er beperkte ontwikkeling van technologieën en instrumenten voor bacteriële identificatie en tellen tijdens ruimte missies15.
Traditionele methoden van kiemgetal vinden alleen culturable cellen, die een minderheid van soorten in milieu stammen, soms kunnen vertegenwoordigen < 1%16. Platen vereisen dagen of weken van incubatie voor maximaal succes, afhankelijk van de stam. Epifluorescence microscopie heeft grotendeels plaat graven als de gouden standaard voor snelle en accurate microbiële opsomming vervangen. Nucleic-zuur-labeling fluorescente kleurstoffen zoals 4 ‘, 6-diamidino-2-phenylindole-dihydrochloride (DAPI), SYBR Green of acridine oranje die zich aan nucleic zuren binden zijn de typische kleurstoffen gebruikt17,18,19 , hoewel vele studies gebruiken fluorescerende indicatoren van Gram ondertekenen20,21,22,23,24. Met behulp van deze methoden zonder pre concentratie stappen leidt tot detectiegrenzen (LoDs) van ~ 105 cellen per mL. Er zijn verbeteringen in LoD mogelijk gebruik van filtratie. Een vloeibare monster is vacuüm-gefilterd op een membraan, meestal polycarbonaat en ideaal zwart te verminderen achtergrond. Low-achtergrond kleurstoffen zoals de vlekken van DNA bovengenoemde kunnen rechtstreeks worden toegepast op de filter-25. Voor het nauwkeurig tellen met het blote oog, ~ 105 cellen zijn vereist per filter, wat dat voor monsters meer verdund dan ~ 105 cellen per mL betekent, significante steekproef volumes worden verzameld en gefilterd. Laser-scannen apparaten zijn ontwikkeld om systematisch Ontdek alle regio’s van de filter en dus vermindering van het aantal cellen die nodig zijn voor het tellen, verlegging van de grenzen van de detectie tot ~ 102 cellen per mL26. Echter, deze zijn niet beschikbaar in de meeste laboratoria, en vereisen geavanceerde hardware zowel als software waarmee deskundige bevestiging dat deeltjes waargenomen zijn bacteriën en niet puin.
Ter referentie, volwassenen met sepsis beginnen meestal met symptomen bij < 100 cellen/mL bloed en zuigelingen op < 10 cellen/mL. Een bloed trekken uit een volwassene neemt 10 mL, en van een kind, 1 mL. PCR gebaseerde methoden worden geremd door de aanwezigheid van menselijke en niet-pathogene flora DNA en door PCR-remmende componenten in het bloed27,28. Ondanks een verscheidenheid van technieken in opkomst blijven culturen de gouden standaard voor de diagnose van infecties van de bloedbaan, vooral in de meer landelijke gebieden of ontwikkelingslanden. Voor de detectie van het leven op andere planeten, kunnen thermodynamische berekeningen schatten het energiebudget voor leven en dus ook de verwachte mogelijk biomassa. 1 – 100 cellen/mL verwachting thermodynamisch redelijk op Europa29. Het kan worden gemakkelijk afgeleid uit deze nummers dat detectie van zeer kleine aantallen cellen in grote hoeveelheden waterige oplossing een belangrijk onopgelost probleem is.
In deze paper, tonen we detectie van Serratia marcescens en Shewanella oneidensis (wild-type en niet-sporenvormende mutant) bij concentraties van 105, 104, 103en 100 cellen/mL met behulp van een off-axis digitale holografische Microscoop (DHM). Het belangrijkste voordeel van DHM over traditionele lichte microscopie is de gelijktijdige beeldvorming van een dikke monstervolume op hoge resolutie — in deze uitvoering was de monsterkamer 0.8 mm dik. Deze steekproef kamers werden gebouwd door de zacht-litho Polydimethylsiloxaan (PDMS) van een precisie-gefreesd aluminium schimmel met een tolerantie van ± 50 µm. Dit is een ongeveer 100-fold verbetering in de diepte van veld opzichte high-power licht microscopie. DHM biedt ook kwantitatieve fase informatie, waardoor metingen van optische weglengte (product van de brekingsindex en dikte). DHM en soortgelijke technieken werden gebruikt voor het toezicht op bacteriële en gist celcyclus en berekening van bacteriële droge massa30,31,32; verstrooiing verschillen kunnen zelfs worden gebruikt om te onderscheiden van de bacteriestammen33.
Het instrument dat wij gebruiken is custom-built specifiek voor gebruik met micro-organismen, als eerder gepubliceerde34,35, en het ontwerp en de constructie worden gedemonstreerd en besproken. Waterige oplossingen worden doorlopend geleverd aan een 0,25 µL volume monsterkamer via spuitpomp; het debiet wordt bepaald door de framesnelheid van de camera met het oog op de beeldvorming van het gehele monstervolume. Een statistische berekening voorspelt het aantal monster volumes die image moet worden gemaakt om op te sporen van een groot aantal cellen met een bepaalde concentratie.
Voor cel-detectie toepassingen was wederopbouw van de hologrammen in amplitude en fase beelden niet vereist; analyse werd uitgevoerd op het ruwe hologram. Dit bespaart aanzienlijke rekenkracht en schijfruimte: een 500 Mb hologram video zal worden 1-2 Tb wanneer gereconstrueerd. We echter wederopbouw door de diepte van het monster te bevestigen dat de hologrammen de gewenste soort vertegenwoordigt bespreken. Een belangrijk kenmerk van DHM is haar vermogen om te controleren zowel de intensiteit en de fase van de beelden. Organismen die bijna transparant in intensiteit (zoals de meeste biologische cellen zijn) worden duidelijk weergegeven in fase. Zoals het is een label-vrije techniek, worden geen kleurstoffen gebruikt. Dit is een eendvantage voor mogelijke ruimtevaart toepassingen, omdat kleurstoffen niet op de voorwaarden van een missie overleven kunnen en — belangrijker — niet kan worden aangenomen om te werken met buitenaardse organismen, die mag niet DNA of RNA voor codering gebruiken. Het is ook een voordeel voor werk in extreme omgevingen zoals de arctische en Antarctische, waar kleurstoffen kunnen moeilijk te brengen naar de externe locatie en kunnen afnemen bij opslag. Reconstructie van beelden in fase en amplitude wordt uitgevoerd met behulp van een open-source softwarepakket dat we beschikbaar hebben gesteld op GitHub (SHAMPOO) of met behulp van ImageJ.
Protocol
Representative Results
Discussion
Numerieke wederopbouw van hologrammen: voor de numerieke wederopbouw van hologrammen, de hoekige spectrum methode (ASM) wordt gebruikt. Het gaat hierbij om de convolutie van het hologram met de Greense functie voor de DHM. De complexe wavefront van de afbeelding op een bepaalde brandvlak kan worden berekend door gebruik te maken van de Fourier convolutie stelling als volgt:
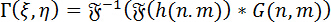 (1)
(1)
Waar <i…
Disclosures
The authors have nothing to disclose.
Acknowledgements
De auteurs erkennen de Gordon en Betty Moore Foundation subsidies 4037 en 4038 aan het California Institute of Technology voor financiering van dit werk.
Materials
| Bacto Yeast | BD Biosciences | 212750 | |
| Bacto Tryptone | BD Biosciences | 211705 | |
| Sodium chloride | Sigma-Aldrich | 7710 | Many options for purchase |
| Bacto Agar | BD Biosciences | 214010 | |
| 10 cm Petri dishes | VWR | 10053-704 | |
| 15 mL culture tubes | Falcon (Corning Life Sciences) | 352002 | Loose-capped |
| Petroff-Hauser chamber | Electron Microscopy Sciences | 3920 | |
| 10 mL syringes | BD Biosciences | 309604 | Luer-Lok tip not necessary |
| Male Luer to 1/16” barbed fitting | McMaster-Carr | 51525K291 | |
| Autoclavable 1/16” ID PVC tubing for flow | Nalgene | 8000-0004 | Sold by length, purchase accordingly |
| Syringe pump | Harvard Apparatus | PHD 2000 | |
| Sample Chamber | Custom | n/a | See Materials Section |
| Holographic Microscope | Custom | n/a | See Wallace et al. |
| Open-source software | Custom | https://github.com/bmorris3/shampoo |
References
- Akineden, O., Weirich, S., Abdulmawjood, A., Failing, K., Buelte, M. Application of a Fluorescence Microscopy Technique for Detecting Viable Mycobacterium avium ssp. paratuberculosis Cells in Milk. Food Anal. Methods. 8, 499-506 (2015).
- Deibl, J., Paulsen, P., Bauer, F. Rapid enumeration of total aerobic microbial counts on meat and in meat products by means of the direct epifluorescence filter technique. Wien Tierarztl Monat. 85, 327-333 (1998).
- Huang, J., Li, Y., Slavik, M. F., Tao, Y., Huff, G. R. Identification and enumeration of Salmonella on sample slides of poultry carcass wash water using image analysis with fluorescent microscopy. Transactions of the Asae. 42, 267-273 (1999).
- Durtschi, J. D., et al. Increased sensitivity of bacterial detection in cerebrospinal fluid by fluorescent staining on low-fluorescence membrane filters. J Med Microbiol. 54, 843-850 (2005).
- Panicker, R. O., Soman, B., Saini, G., Rajan, J. A Review of Automatic Methods Based on Image Processing Techniques for Tuberculosis Detection from Microscopic Sputum Smear Images. J Med Syst. 40, (2016).
- Riepl, M., et al. Applicability of solid-phase cytometry and epifluorescence microscopy for rapid assessment of the microbiological quality of dialysis water. Nephrol Dial Transplant. 26, 3640-3645 (2011).
- Hwang, C. Y., Cho, B. C. Virus-infected bacteria in oligotrophic open waters of the East Sea, Korea. Aquat Microb Ecol. 30, 1-9 (2002).
- Kepner, R. L., Pratt, J. R. Use of Fluorochromes for Direct Enumeration of Total Bacteria in Environmental-Samples – Past and Present. Microbiol Rev. 58, 603-615 (1994).
- Queric, N. V., Soltwedel, T., Arntz, W. E. Application of a rapid direct viable count method to deep-sea sediment bacteria. J Microbiol Meth. 57, 351-367 (2004).
- Prieto-Ballesteros, O., Vorobyova, E., Parro, V., Rodriguez Manfredi, J. A., Gomez, F. Strategies for detection of putative life on Europa. Adv Space Res. 48, 678-688 (2011).
- Gleeson, D. F., et al. Biosignature Detection at an Arctic Analog to Europa. Astrobiology. 12, 135-150 (2012).
- Carr, C. E., Zuber, M. T., Ruvkun, G., Ieee, . 2013 Ieee Aerospace Conference IEEE Aerospace Conference Proceedings. , (2013).
- Konstantinidis, K., et al. A lander mission to probe subglacial water on Saturn’s moon Enceladus for life. Acta Astronautica. 106, 63-89 (2015).
- McKay, C. P., Anbar, A. D., Porco, C., Tsou, P. Follow the Plume: The Habitability of Enceladus. Astrobiology. 14, 352-355 (2014).
- Hoover, R. B., Hoover, R. B., Levin, G. V., Rozanov, A. Y., Wickramasinghe, N. C. . Instruments, Methods, and Missions for Astrobiology Xvii. , (2015).
- Handelsman, J. Metagenomics: application of genomics to uncultured microorganisms. Microbiol Mol Biol Rev: MMBR. 68, 669-685 (2004).
- Lebaron, P., Parthuisot, N., Catala, P. Comparison of blue nucleic acid dyes for flow cytometric enumeration of bacteria in aquatic systems. Appl Environ Microbiol. 64, 1725-1730 (1998).
- Marie, D., Partensky, F., Jacquet, S., Vaulot, D. Enumeration and cell cycle analysis of natural populations of marine picoplankton by flow cytometry using the nucleic acid stain SYBR Green I. Appl Environ Microbiol. 63, 186-193 (1997).
- Noble, R. T., Fuhrman, J. A. Use of SYBR Green I for rapid epifluorescence counts of marine viruses and bacteria. Aquat Microb Ecol. 14, 113-118 (1998).
- Forster, S., Snape, J. R., Lappin-Scott, H. M., Porter, J. Simultaneous fluorescent gram staining and activity assessment of activated sludge bacteria. Appl Environ Microbiol. 68, 4772-4779 (2002).
- Lauer, B. A., Reller, L. B., Mirrett, S. Comparison Of Acridine-Orange And Gram Stains For Detection Of Microorganisms In Cerebrospinal-Fluid And Other Clinical Specimens. J Clin Microbiol. 14, 201-205 (1981).
- Mason, D. J., Shanmuganathan, S., Mortimer, F. C., Gant, V. A. A fluorescent gram stain for flow cytometry and epifluorescence microscopy. Appl Environ Microbiol. 64, 2681-2685 (1998).
- Saida, H., Ytow, N., Seki, H. Photometric application of the Gram stain method to characterize natural bacterial populations in aquatic environments. Appl Environ Microbiol. 64, 742-747 (1998).
- Sizemore, R. K., Caldwell, J. J., Kendrick, A. S. Alternate Gram Staining Technique Using A Fluorescent Lectin. Appl Environ Microbiol. 56, 2245-2247 (1990).
- Bitton, G., Dutton, R. J., Foran, J. A. New Rapid Technique For Counting Microorganisms Directly On Membrane Filters. Stain Technology. 58, 343-346 (1983).
- Broadaway, S. C., Barton, S. A., Pyle, B. H. Rapid staining and enumeration of small numbers of total bacteria in water by solid-phase laser cytometry. Appl Environ Microbiol. 69, 4272-4273 (2003).
- Yagupsky, P., Nolte, F. S. Quantitative aspects of septicemia. Clin Microbiol Rev. 3, 269-279 (1990).
- Kang, D. K., et al. Rapid detection of single bacteria in unprocessed blood using Integrated Comprehensive Droplet Digital Detection. Nat Commun. 5, 5427 (2014).
- Hand, K. P. . Report of the Europa Lander Science Definition Team. , (2017).
- Popescu, G., et al. Optical imaging of cell mass and growth dynamics. Am J Physiol-Cell Physiol. 295, C538-C544 (2008).
- Mir, M., et al. Optical measurement of cycle-dependent cell growth. Proc Natl Acad Sci U S A. 108, 13124-13129 (2011).
- Rappaz, B., et al. Noninvasive characterization of the fission yeast cell cycle by monitoring dry mass with digital holographic microscopy. J.Biomed Opt. 14, 034049 (2009).
- Jo, Y., et al. Label-free identification of individual bacteria using Fourier transform light scattering. Opt. Express. 23, 15792-15805 (2015).
- Wallace, J. K., et al. Robust, compact implementation of an off-axis digital holographic microscope. Opt. Express. 23, 17367-17378 (2015).
- Lindensmith, C. A., et al. A Submersible, Off-Axis Holographic Microscope for Detection of Microbial Motility and Morphology in Aqueous and Icy Environments. Plos One. 11, e0147700 (2016).
- Dumas, E. M., Ozenne, V., Mielke, R. E., Nadeau, J. L. Toxicity of CdTe Quantum Dots in Bacterial Strains. IEEE Trans. NanoBiosci. 8, 58-64 (2009).
- Frisk, A., Jyot, J., Arora, S. K., Ramphal, R. Identification and functional characterization of flgM, a gene encoding the anti-sigma 28 factor in Pseudomonas aeruginosa. J Bacteriol. 184, 1514-1521 (2002).
- Adler, J., Templeton, B. The effect of environmental conditions on the motility of Escherichia coli. Journal Gen Microbiol. 46, 175-184 (1967).
- Piedrahita-Quintero, P., Castaneda, R., Garcia-Sucerquia, J. Numerical wave propagation in Image J. Appl Opt. 54, 6410-6415 (2015).
- Schnars, U., Jüptner, W. P. Digital recording and numerical reconstruction of holograms. Meas. Sci. Technol. 13, R85 (2002).

