大动物淋巴结的采集与处理 RNA 分析: 大动物 Transcriptomic 淋巴结的制备研究
Summary
本议定书概述了从大型动物的淋巴结组织 transcriptomic 分析中分离 RNA 的程序, 包括从家畜和野生动物中鉴别和切除淋巴结的步骤, 取样方法为了在多个动物之间提供一致性, 并考虑到收集后保存和处理 RNA 分析的代表性结果。
Abstract
大型动物 (牲畜和野生动物) 是人畜共患病病原体的重要储集物, 包括布鲁氏杆菌、牛分枝菌、沙门氏菌和大肠杆菌, 并有助于研究发病机制和/或细菌在天然寄主中的传播。随着淋巴结在宿主免疫应答中的关键作用, 淋巴结组织作为潜在的 RNA 来源, 为下游 transcriptomic 分析, 以评估在细胞感染过程中基因表达的时间变化。本文概述了在家畜的淋巴结收集, 组织取样和下游 RNA 处理的过程, 使用牛 (金牛座) 作为模型, 并提供了更多的例子, 美国野牛 (野牛野牛).该协议包括有关人体内多个关键部位淋巴结的位置、识别和清除的信息。此外, 还提出了一种活检抽样方法, 允许在多个动物之间进行抽样的一致性。讨论了样品保存的几个考虑因素, 包括 rna 序列和 rt-pcr 等下游方法的生成。由于大型动物vs小鼠时间课程研究中固有的长时间延迟, 提出了野牛和牛淋巴结组织的代表性结果来描述这种组织类型中退化的时间过程, 在审查其他组织中 RNA 降解的以前的方法学研究。总的来说, 这项议定书将是有益的两个兽医研究员开始转录项目的大型动物样本和对学习技术的分子生物学家感兴趣的体内组织取样和体外处理。
Introduction
RNA 序列分析的淋巴结转录提供了机会, 以描述动物的免疫反应的各种病原体。虽然这种方法已广泛应用于小鼠, 但最近的分析已经扩展到较大的哺乳动物1,2。牲畜/大型动物淋巴结可用于鉴定宿主对感染的反应, 不仅用于疫苗或遗传易感性研究以及确定药物发展目标, 而且还作为人类研究的模范系统。关于人畜共患病。例如, 在布鲁氏菌病 (每年影响全世界100万人的人畜共患病细菌疾病) 的情况下, 尽管费用大大增加, 但绵羊或山羊的研究与人类感染和人类疫苗的相关性更大。发展比实验动物模型。老鼠感染模型重述了内皮系统感染, 但没有特点的临床标志3.
在大型动物实验中, 与实验动物研究相比, 组织收获的过程必然涉及安乐死与组织收集之间的较长延迟, 这给保存高质量的 RNA。完整的 RNA 是产生生物相关 transcriptomic 数据的必要条件。从组织样本中生成高质量的 RNA 对于在密闭设施中进行的大型动物病原体研究尤其重要。这种研究在本质上更难执行, 因为它们不仅需要核定的设施和训练有素的人员, 而且还要承担重大的财政费用, 这取决于工作, 可以从数以万计的美元不等。这些类型的研究还涉及跨学科协作和跨学科的知识, 以完成他们的工作, 增加他们的复杂性。因此, 对样本收集和保存的简化系统进行培训、开发和遵守, 为受感染动物组织的下游分子研究提供了重要的好处。
与小鼠淋巴结相似抽样相比, 大淋巴结的收集对组织收集带来了额外的挑战。标本切除的准备需要对淋巴结的解剖, 包括相关的内部结构有基本的理解。淋巴结的结构由淋巴小叶包围, 由鼻窦充满淋巴。这些结构被封闭在一个坚硬的纤维胶囊中。4淋巴小叶是 “淋巴结基本解剖和功能单元”, 由卵泡、深皮质单元、髓质线和鼻窦4 (图 1A) 组成。B 和 T 淋巴细胞分别是卵泡和深皮质单位的家。这些结构提供一个3D 支架和促进淋巴细胞和抗原或抗原呈现细胞之间的相互作用。
严重的, 卵泡和深皮质单位可以确定在切割表面, 因为他们包含一个密集的网状网, 并显示较深的鼻窦, 这是由一个更微妙的网状网和显得更轻 (图 1B)。根据惯例, 病理学家指淋巴结的区域为浅皮层 (卵泡)、paracortex (深皮质单位) 和髓质 (髓质线和鼻窦)。对所有三个区域进行的适当检查已被认为是淋巴结常规病理检查指南的最佳实践5。请注意, 淋巴结的一致性、大小和颜色有相当大的变化, 即使在单个动物中也是如此。随着动物年龄的增长, 他们的淋巴结将趋于减少, 并变得比年轻动物更牢固, 通常由于其结缔组织的增加和正常淋巴结构的减少6,7。
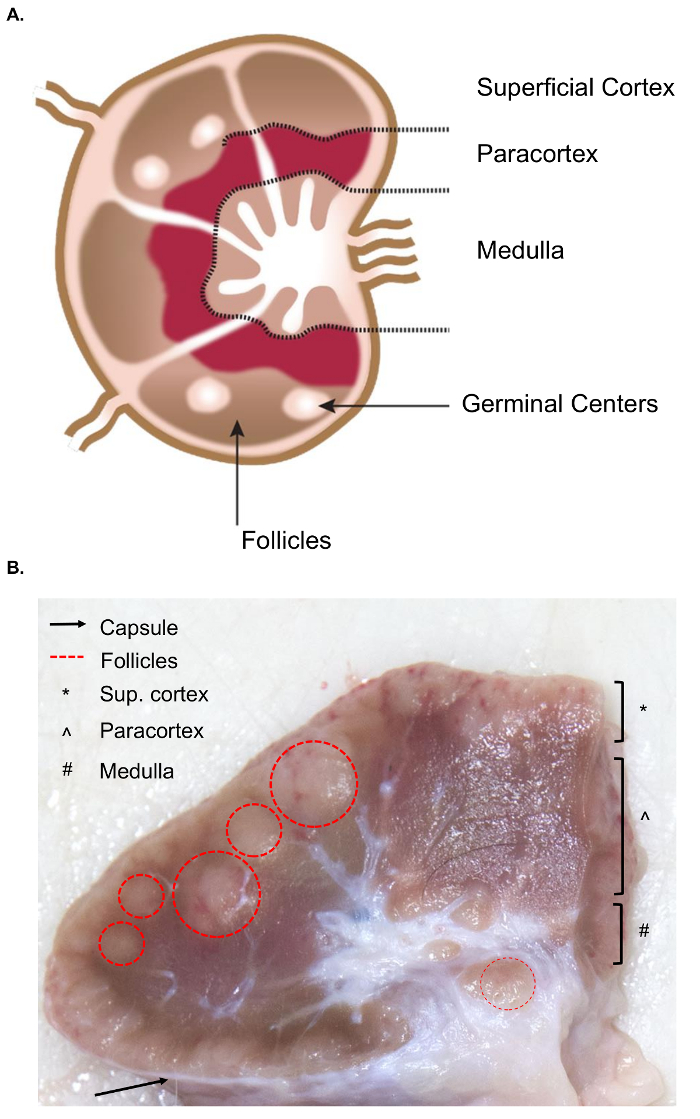
图 1.淋巴结解剖。(A) 此卡通图像显示了淋巴结的解剖, 描述了关键结构。(B) 此静止图像显示一个牛淋巴结切开横断面。对肉眼可见的相关结构/层进行了突出显示。请单击此处查看此图的较大版本.
根据实验性的问题, 不同淋巴结的收集和分析有一定的兴趣。外周淋巴结位于皮下组织深处。在牛, 周围或浅淋巴结经常用于临床和实验的做法包括腮腺, 颌下腺, 咽后, prescapular, prefemoral (precrural) 和浅腹股沟 (supramammary 在女性, 阴囊在男性) (图 2)。在表 1中, 概述了牛系统8中描述的主要浅淋巴结的属性。下面, 提出了一些潜在的牛传染性细菌性疾病淋巴结采集计划, 作为调查的出发点。
布鲁氏菌流产/布鲁氏菌 melitensis: 标准验尸为b. 流产受感染的牛和b. melitensis感染的山羊在国家动物疾病中心恢复 supramammary, prescapular 和腮腺淋巴结组织, 无论是为细菌计数的研磨和为 rna 的准备为宿主 rna 表达分析。在实验感染的牛9中, 每个淋巴结都可以定期恢复B. 流产.在B. melitensis-感染的山羊中, 在感染后至少九月内, 使用我们研究中的基于 RNA 的方法 (Boggiatto et., 未发表), 可以在这些淋巴结类型中检测到细菌的存在。沙门氏菌sp: prescapular、subiliac (prefemoral) 和肠系膜淋巴结在对沙门氏菌患病率10、11、12和对 transcriptomic 研究有潜在的兴趣。大肠杆菌 O157:H7: 肠系膜淋巴结 (在中间小肠和远端小肠位置) 可以是偶尔恢复的细菌在受感染的小牛 (但不是在受感染的成年牛)13。钩端螺旋体病 (钩端螺旋体 sp.): 在引流乳腺14的淋巴结中观察到细菌的长期持续存在。牛分枝杆菌: 在牛中, 这些细菌已经从小牛的纵隔和气管支气管淋巴结的实验性感染中恢复了15。此外, 淋巴结 RNA 被用来检查大型动物宿主对病毒的反应, 如猪繁殖和呼吸道综合征病毒2。图 2描述了牛体内这些主要淋巴结的子集位置。
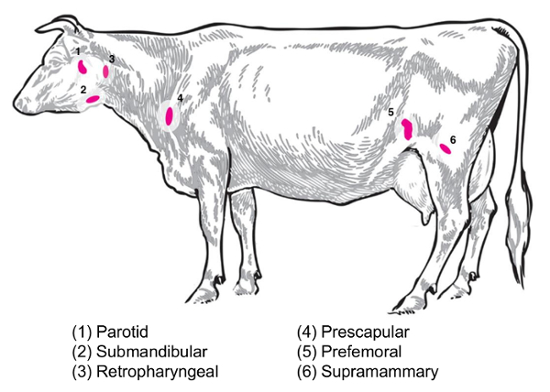
图 2:动画描述选定的淋巴结位置金牛座. 编号的淋巴结被标注。请单击此处查看此图的较大版本.
在本文和相关视频中, 我们提出了一种用于 RNA 研究的大型动物淋巴结分离的协议, 旨在为参与大型动物感染 transcriptomic 研究的分子生物学家提供信息。首先, 我们以牛和野牛组织的抽样为例, 概述了淋巴结的分离过程。与这个演示, 如显示在视频中, 是一个工作流为一个可重现的组织取样, RNA 隔离。接下来, 我们描述了处理感染性淋巴结的重要因素, 重点是安全性、一致性和 RNA 质量。
用酸化苯酚-胍基异硫氰酸酯试剂制备组织中的 RNA, 是基于 Chomczynski 和萨基16、17的原始方法, 在存在的情况下, 对基于二氧化硅的自旋柱进行纯化。基于 Vogelstein 和 chaotropic18的原始工作的代理。我们还研究了从替代方法保存的牛淋巴结中恢复转录组学 RNA 的可能性。最后, 我们探讨了时间变量对大动物验尸 rna 质量的影响, 包括一个代表性的实验, 描述了在安乐死与从野牛中恢复的 rna 剖面上的采样时间的增加的影响, 并牛淋巴结。这篇文章将不仅对分子生物学家, 而且对兽医研究人员开始 transcriptomic 研究是有用的。
Protocol
Representative Results
Discussion
大多数的 transcriptomic 研究和相关的协议集中在老鼠, 老鼠, 或验尸后的人类样本。然而, 对牲畜和野生动物的调查为对疾病的免疫反应的定性提供了广泛的机会, 无论是适用于兽医医学, 还是关于人畜共患病, 人类公共卫生。该协议提供了一个概要的关键考虑的高完整性 RNA 提取从组织从大型动物, 如牛, 野牛, 山羊和绵羊。动物的大小, 以及样本收集的条件, 使恢复更广泛的过程, 更长的验尸后处理时?…
Disclosures
The authors have nothing to disclose.
Acknowledgements
作者感谢詹姆斯福斯在所有录像和视频处理方面的出色工作;迈克尔. 马蒂为他的优秀作品在一代数字化牛形象;莉莉娅瓦尔特为她的帮助与 RNA 提取和 Bioanalyzer 奔跑;米奇. 帕默和卡莉 Kanipe 对淋巴结图像进行了有益的回顾和反馈;和动物护理和兽医人员在国家动物疾病中心为他们的辛勤工作和协助畜牧业和验尸的准备。
Materials
| RNA preservation solution (we used RNALater for all experiments) | ThermoFisher | AM7020 | |
| 1.5 ml or 2 ml polypropylene microcentrifuge tubes | Fisher Scientific | 05-408-129 | |
| Disposable scalpels | Daigger Scientific | EF7281 | |
| Tissue forceps, rat tooth | Fisher Scientific | 12-460-117 | Other tissue forceps available including curved tip, tapered edge, etc. , depends on user preference |
| 3 mm punch biopsy needles | Fisher Scientific | NC9949469 | |
| Sharps container (small and transportable for necropsy) | Stericycle | 8900SA | 1 qt. size shown here |
| Cutting boards or disposable trays | Fisher Scientific | 09-002-24A | Available in a variety of sizes, depends on user preference |
| Personal protective equipment | Varies with pathogen (gloves, respirator masks, goggles, etc.) | ||
| Phenol-based RNA extraction reagent (we used TRIzol Reagent for all experiments) | ThermoFisher | 15596026 | |
| Silica column-based RNA extraction kit (we used the PureLink RNA Mini kit for all experiments) | ThermoFisher | 12183018A | Designed for up to 100 mg tissue |
| 100% Ethanol (200 proof for molecular biology) | Sigma-Aldrich | E7023 | |
| Tissue homogenizer with enclosed homogenization tubes (we used the gentleMACS dissociator for all experiments) | Miltenyi Biotec | 130-093-235 | |
| Agarose (General, for gel electrophoresis) | Sigma-Aldrich | A9539 | |
| 1X TBE | Fisher Scientific | BP24301 | Can also make from scratch in the laboratory |
| Deionized formamide | EMD Millipore | S4117 | |
| Sodium dodecyl sulfate | Sigma-Aldrich | L3771 | |
| Bromophenol blue | Sigma-Aldrich | 114391 | |
| Xylene cyanol | Sigma-Aldrich | X4126 | |
| EDTA (Ethylenediaminetetraacetic acid) | Sigma-Aldrich | EDS | |
| UV-Vis Spectrophotometer (we used the NanoDrop Spectrophotometer) | ThermoFisher | ND-2000 | |
| Device for quantitative RNA assessment (we used the Bioanalyzer, with associated components and protocols) | Agilent | G2939BA | |
| FFPE RNA extraction kit (we used the RecoverAll Total Nucleic Acid Isolation Kit for Formalin Fixed, Paraffin Embedded Tissue) | ThermoFisher | AM1975 | |
| Plastic spreader (L-shaped spreader) | Fisher Scientific | 14-665-231 | Only needed for sterility testing for samples from infected animals |
| Necropsy knives | Livestock Concepts | WI-0009209 |
References
- Tizioto, P. C., et al. Immunological response to single pathogen challenge with agents of the bovine respiratory disease complex: an RNA-sequence analysis of the bronchial lymph node transcriptome. PLoS One. 10 (6), e0131459 (2015).
- Miller, L. C., et al. Analysis of the swine tracheobronchial lymph node transcriptomic response to infection with a Chinese highly pathogenic strain of porcine reproductive and respiratory syndrome virus. BMC Veterinary Research. 8, 208 (2012).
- Silva, T. M. A., Costa, E. A., Paixao, T. A., Tsolis, R. M., Santos, R. L. Laboratory animal models for brucellosis research. Journal of Biomedicine and Biotechnology. 2011, 518323 (2011).
- Willard-Mack, C. L. Normal structure, function, and histology of the lymph nodes. Toxicologic Pathology. 34, 409-424 (2006).
- Elmore, S. A. Histopathology of the lymph nodes. Toxicologic Pathology. 34 (5), 425-454 (2006).
- Luscieti, P., Hubschmid, T. h., Cottier, H., Hess, M. W., Sobin, L. H. Human lymph node morphology as a function of age and site. Journal of Clinical Pathology. 33 (5), 454-461 (1980).
- Hadamitsky, C., et al. Age-dependent histoarchitectural changes in human lymph nodes: an underestimated process with clinical relevance?. Journal of Anatomy. 216 (5), 556-562 (2010).
- Sisson, S., Grossman, J. D., Getty, R. . Sisson and Grossman’s The Anatomy of Domestic Animals, Volume 1. , (1975).
- Olsen, S. C., Johnson, C. Comparison of abortion and infection after experimental challenge of pregnant bison and cattle with Brucella abortus strain 2308. Clinical and Vaccine Immunology. 18 (12), 2075-2078 (2011).
- Brichta-Harhay, D. M., et al. Microbiological analysis of bovine lymph nodes for the detection of Salmonella enterica. Journal of Food Protection. 75 (5), 854-858 (2012).
- Samuel, J. L., O’Boyle, D. A., Mathers, W. J., Frost, A. J. Isolation of Salmonella from mesenteric lymph nodes of healthy cattle at slaughter. Research in Veterinary Science. 28 (2), 238-241 (1980).
- Arthur, T. M., et al. Prevalence and characterization of Salmonella in bovine lymph nodes potentially destined for use in ground beef. Journal of Food Protection. 71 (8), 1685-1688 (2008).
- Cray, W. C., Moon, H. W. Experimental infection of calves and adult cattle with Escherichia coli O157:H7. Applied and Environmental Microbiology. 61 (4), 1586-1590 (1995).
- Thiermann, A. B. Experimental leptospiral infections in pregnant cattle with organisms of the Hebdomadis serogroup. American Journal of Veterinary Research. 43 (5), 780-784 (1982).
- Palmer, M. V., Waters, W. R., Whipple, D. L. Investigation of the transmission of Mycobacterium bovis from deer to cattle through indirect contact. American Journal of Veterinary Research. 65 (11), 1483-1489 (2004).
- Chomczynski, P. A reagent for the single-step simultaneous isolation of RNA, DNA, and proteins from cell and tissue samples. BioTechniques. 15 (3), 532-537 (1993).
- Chomczynski, P., Sacchi, N. Single step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction. Analytical Biochemistry. 162 (1), 156-159 (1987).
- Vogelstein, B., Gillespie, D. Preparative and analytical purification of DNA from agarose. Proceedings of the National Academy of Sciences of the United States of America. 76 (2), 615-619 (1979).
- . . AVMA Guidelines for the Euthanasia of Animals. , (2013).
- . . TRIzol Reagent Manual. , (2016).
- . . Disruption and Homogenization of Tissue for the Extraction of RNA. , (2014).
- . . PureLink RNA Mini Kit Protocol . , (2012).
- . . RNA Gel-loading Buffer. , (2006).
- Rio, D. C., Ares, M., Hannon, G. J., Nilsen, T. W. Nondenaturing Agarose Gel Electrophoresis of RNA. Cold Spring Harbor Protocols. , (2010).
- Mueller, O., et al. A microfluidic system for high-speed reproducible DNA sizing and quantitation. Electrophoresis. 21 (1), 128-134 (2000).
- Schroeder, A., et al. The RIN: an RNA integrity number for assigning integrity values to RNA measurements. BMC Molecular Biology. 7 (3), (2006).
- Suvarna, K. S., Layton, C., Bancroft, J. D. . Bancroft’s Theory and Practice of Histological Techniques. , (2012).
- Medawar, P. B. The rate of penetration of fixatives. Journal of Microscopy. 61 (1-2), 46-57 (1941).
- Canene-Adams, K. Preparation of formalin-fixed paraffin-embedded tissue for immunohistochemistry. Methods in Enzymology. 533, 225-233 (2013).
- Fleige, S., Pfaffl, M. W. RNA integrity and the effect on the real-time qRT-PCR performance. Molecular Aspects of Medicine. 27, 126-139 (2006).
- Fajardy, I., et al. Time course analysis of RNA stability in human placenta. BMC Molecular Biology. 10 (21), (2009).
- Bahar, B., et al. Long-term stability of RNA in post-mortem bovine skeletal muscle, liver and subcutaneous adipose tissues. BMC Molecular Biology. 8, 108 (2007).
- Yamagishi, A., et al. Gene profiling and bioinformatics analyses reveal time course differential gene expression in surgically resected colorectal tissues. Oncology Reports. 31 (4), 1532-1538 (2014).
- Choi, S., Ray, H. E., Lai, S. H., Alwood, J. S., Globus, R. K. Preservation of multiple mammalian tissues to maximize science return from ground based and spaceflight experiments. PLoS One. 11 (12), e0167391 (2016).
- Almeida, A., Thiery, J. P., Magdelenat, H., Radvanyi, F. Gene expression analysis by real-time reverse transcription polymerase chain reaction: influence of tissue handling. Analytical Biochemistry. 328 (2), 101-108 (2004).
- Lee, S. M. L., Schelcher, C., Thasler, R., Schiergens, T. S., Thasler, W. E. Pre-analytical determination of the effect of extended warm or cold ischaemia on RNA stability in the human ileum mucosa. PLoS One. 10 (9), e0138214 (2015).
- Kap, M., et al. The influence of tissue procurement procedures on RNA integrity, gene expression, and morphology in porcine and human liver tissue. Biopreservation and Biobanking. 13 (3), 200-206 (2015).
- Hong, S. H., et al. Effects of delay in the snap freezing of colorectal cancer tissues on the quality of DNA and RNA. Journal of the Korean Society of Coloproctology. 26 (5), 316-325 (2010).
- Lee, S. M., et al. RNA stability in human liver: comparison of different processing times, temperatures and methods. Molecular Biotechnology. 53 (1), 1-8 (2013).
- Morrison, P. K., et al. Post-mortem stability of RNA in skeletal muscle and adipose tissue and the tissue-specific expression of myostatin, perilipin and associated factors in the horse. PLoS One. 9 (6), e100810 (2014).
- Seear, P. J., Sweeney, G. E. Stability of RNA isolated from post-mortem tissues of Atlantic salmon (Salmo salar L.). Fish Physiology and Biochemistry. 34 (1), 19-24 (2008).
- Marchuk, L., Sciore, P., Reno, C., Frank, C. B., Hart, D. A. Postmortem stability of total RNA isolated from rabbit ligament, tendon, and cartilage. Biochimica et Biophysica Acta. 1379 (2), 171-177 (1998).
- Inoue, H., Kimura, A., Tuji, T. Degradation profile of mRNA in a dead rat body: basic semi-quantification study. Forensic Science International. 130 (2-3), 127-132 (2002).
- Micke, P., et al. Biobanking of fresh frozen tissue: RNA is stable in nonfixed surgical specimens. Laboratory Investigation. 86 (2), 202-211 (2006).
- Srinivasan, M., Sedmak, D., Jewell, S. Effect of fixatives and tissue processing on the content and integrity of nucleic acids. The American Journal of Pathology. 161 (6), 1961-1971 (2002).
- Ahmed, A. M., et al. Variation in the ovine abomasal lymph node transcriptome between breeds known to differ in resistance to the gastrointestinal nematode. PLoS One. 10 (5), e0124823 (2015).
- Russell, G. C., et al. Host gene expression changes in cattle infected with Alcelaphine herpesvirus 1. Virus Research. 169 (1), 246-254 (2012).
- Avraham, R., et al. A highly multiplexed and sensitive RNA-seq protocol for simultaneous analysis of host and pathogen transcriptomes. Nature Protocols. 11, 1477-1491 (2016).

