Collecte et traitement des ganglions lymphatiques des animaux de grande taille pour l’analyse de l’ARN : préparation pour l’étude de transcriptomique ganglion des grandes espèces animales
Summary
Ce protocole donne un aperçu des procédures d’isolement d’ARN pour le profilage de transcriptomique des tissus ganglionnaires de grands animaux, y compris les étapes dans l’identification et l’excision des ganglions lymphatiques du bétail et la faune, échantillonnage des approches pour assurer la cohérence entre plusieurs animaux et des considérations plus des résultats représentatifs pour la préservation de l’après la collecte et le traitement aux fins d’analyse de RNA.
Abstract
Les grands animaux (bétail et la faune) servent d’importants réservoirs d’agents pathogènes zoonotiques, y compris les Brucella, Mycobacterium bovis, Salmonellaet e. coliet sont utiles pour l’étude de la pathogenèse et/ou propagation de la bactérie dans ses hôtes naturels. Avec la touche de fonction des ganglions lymphatiques dans la réponse immunitaire de l’hôte, ganglion lymphatique tissus servent comme une source potentielle de l’ARN pour les analyses transcriptomiques en aval, afin d’évaluer les changements temporels dans l’expression des gènes dans les cellules au cours d’une infection. Cet article présente une vue d’ensemble du processus de ganglion lymphatique collection et prélèvement d’ARN en aval traitement du bétail, utilisant des bovins (Bos taurus) comme un modèle, avec des exemples supplémentaires fournis par le bison d’Amérique (Bison bison ). Le protocole contient des informations sur la localisation, l’identification et enlèvement des ganglions lymphatiques de plusieurs sites clés dans le corps. Par ailleurs, une méthode d’échantillonnage de biopsie est présentée qui permet une cohérence de l’échantillonnage à travers plusieurs animaux. Plusieurs considérations pour la conservation de l’échantillon sont examinées, y compris la production d’ARN adapté aux méthodologies en aval comme RNA-sequencing et RT-PCR. Par les longs délais inhérents aux gros animaux vs des études souris temps, des résultats représentatifs de bison et de tissus bovins ganglion sont présentés pour décrire l’évolution temporelle de la dégradation dans ce type de tissu, dans le cadre d’un examen de travaux méthodologique antérieurs sur la dégradation de l’ARN dans d’autres tissus. Dans l’ensemble, ce protocole sera utile pour les deux chercheurs vétérinaires en début du transcriptome projets sur grands échantillons d’animaux et de biologistes moléculaires intéressés par l’apprentissage de techniques pour échantillons in vivo et in vitro de traitement.
Introduction
RNA-sequencing analyse du transcriptome de ganglions lymphatiques offre la possibilité de caractériser la réponse immunitaire des animaux à une variété de pathogènes. Tandis que cette méthodologie a été utilisée intensivement chez les souris, les analyses ont récemment étendu dans les plus grands mammifères1,2. Bétail/gros ganglions animales peut être utilisées pour caractériser les réactions de l’hôte à une infection, non seulement pour leur utilisation dans les vaccins ou les études de susceptibilité génétique et l’identification de cibles pour le développement de médicaments, mais aussi comme systèmes modèles pour les études chez l’homme sur les maladies zoonotiques. Par exemple, dans le cas de brucellose (une maladie zoonotique bactérienne qu’effets un demi-million de personnes dans le monde entier chaque année), malgré a considérablement augmenté des coûts, des études chez les moutons ou les chèvres sont plus pertinents pour l’infection humaine et le vaccin pour les humains développement de modèles animaux de laboratoire. Modèles de souris infection récapitulent l’infection du système réticulo-endothélial mais pas les signes cliniques caractéristiques3.
Lors des expérimentations animales importantes par rapport aux études animales de laboratoire, le processus de récolte nécessairement le tissu implique un délai plus long entre l’euthanasie et la collection de tissus, qui constitue un défi potentiel pour la conservation des RNA de haute qualité. L’ARN intact est essentiel pour la génération de données transcriptomiques biologiquement pertinente. Est de la génération de l’ARN de qualité des échantillons de tissus particulièrement critique pour les études de pathogène animal grand menée dans des installations de confinement. Ces études sont par nature plus difficiles à effectuer car non seulement ils exigent agréées et un personnel hautement qualifié mais aussi portent des frais importants, qui, selon les travaux, peuvent varier de quelques dizaines à des centaines de milliers de dollars. Ces types d’études impliquent aussi une collaboration interdisciplinaire et connaissances interdisciplinaires pour leur achèvement, ajoutant à leur complexité. Par conséquent, formation, développement d’et l’adhésion à un système simplifié de prélèvement et de conservation procure des avantages importants pour les études moléculaires en aval des tissus provenant d’animaux infectés.
La collection des plus gros ganglions lymphatiques présente des difficultés supplémentaires pour la collection de tissus par rapport à l’échantillonnage semblable des ganglions murines. La préparation pour l’excision de l’échantillon nécessite une compréhension de base de l’anatomie du ganglion, y compris les structures internes pertinentes. La structure d’un ganglion lymphatique est constituée de lobules lymphoïdes entourées de sinus remplis de ganglions. Ces structures sont enfermés dans une capsule dure, fibreuse. 4 un lobule lymphoïde est le « unité anatomique et fonctionnelle fondamentale du ganglion » et se compose de follicules, une unité profonde corticale et cordons médullaires et sinus4 (Figure 1 a). Les lymphocytes B et T sont abrite les follicules et profondément corticales unités, respectivement. Ces structures offrent un échafaudage 3D et facilitent l’interaction entre le lymphocytes ou cellules présentatrices d’antigène.
Grossièrement, follicules et unités profond corticales peuvent être identifiées sur la surface coupée car ils contiennent un maillage plus dense réticulaire et apparaissent plus sombres que les sinus, qui sont constituées d’un maillage réticulaire plus délicat et apparaissent plus léger (Figure 1 b). Par convention, les pathologistes se référer aux régions des ganglions lymphatiques comme le cortex superficiel (follicules), le paracortex (unités corticales profonde) et le bulbe rachidien (cordons médullaires et des sinus). Un examen sérieux de ces trois régions a été jugé comme le meilleur pratique dans les lignes directrices de routine d’examen pathologique des ganglions lymphatiques,5. Notez qu’il existe une variation considérable de la cohérence, la taille et couleur des ganglions lymphatiques, même au sein d’un seul animal. Comme l’âge des animaux, leurs ganglions lymphatiques aura tendance à diminuer en taille et devenir plus ferme que celles des animaux plus jeunes, généralement due à une augmentation dans leur tissu conjonctif et une réduction de la normale lymphoïde structure6,7.
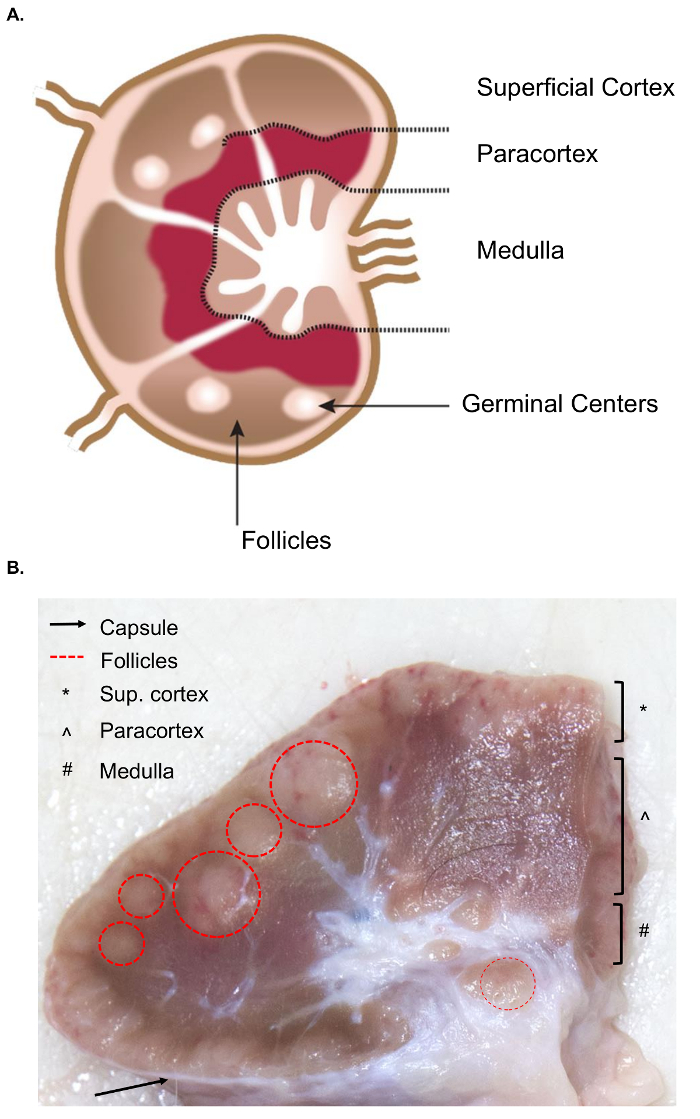
Figure 1. Anatomie du ganglion. (A) cette image de dessin animé montre l’anatomie du ganglion, représentant les principales structures. (B) toujours cette image montre un ganglion bovin couper en coupe transversale. Les structures/couches pertinentes qui sont visibles à le œil nu sont mises en évidence. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Selon la question expérimentale, ganglions différentes seront d’intérêt pour la collecte et l’analyse. Ganglions lymphatiques périphériques sont celles situées profondément dans le tissu sous-cutané. Chez les bovins, les ganglions lymphatiques périphériques ou superficielles souvent utilisés en pratique clinique et expérimentale incluent parotides, sous-maxillaires, rétropharyngiens, préscapulaires préfémoraux (PRECRURAUX) et inguinal superficiel (supramammaires chez les femelles, du scrotum chez les mâles) () La figure 2). Dans le tableau 1, les propriétés des principaux ganglions superficielles, comme décrit dans le système de bovins8, sont mentionnées. Ci-dessous, quelques plans de collection ganglion potentiels pour les maladies infectieuses bactériennes des bovins sont présentés comme point de départ pour l’enquête.
Brucella abortus/Brucella melitensis: norme necropsies pour b. abortus-infectés par les bovins et br. melitensis-chèvres infectés au Centre National des maladies animales récupérer supramammaires, prescapular et le tissu ganglionnaire parotide , tous deux pour la moudre pour l’énumération bactérienne et pour la préparation d’ARN pour le profilage de l’expression hôte RNA. B. abortus peuvent être régulièrement récupérés dans chacun de ces ganglions lymphatiques chez les bovins infectés expérimentalement9. La présence de bactéries dans chacun de ces types de ganglion lymphatique peut être détectée dans br. melitensis-infecté chèvres jusqu’après l’infection au moins neuf mois selon les méthodes de base d’ARN de nos études (Boggiatto et al., inédit). Salmonella SP: le prescapular, subiliaque (préfémoral), et les ganglions mésentériques ont été utiles lors de la détermination des caractéristiques des carcasses de gros bovins pour une prévalence de Salmonella 10,11,12 et serait d’intérêt potentiel pour les études de transcriptomique. E. coli O157 : H7: les ganglions mésentériques (à l’intestin grêle moyen et les emplacements de l’intestin grêle distal) peut être les sites d’une reprise occasionnelle des bactéries chez les veaux infectés (mais pas chez les bovins adultes infectés)13. La leptospirose (Leptospira SP.): une persistance chronique des bactéries a été observée dans les ganglions lymphatiques de drainage de la glande mammaire14. Mycobacterium bovis : Chez les bovins, les bactéries ont été récupérée infection post expérimentale par les ganglions lymphatiques médiastinaux et trachéo-bronchique de veaux15. En outre, ganglion RNA a été utilisée pour examiner les réactions de l’hôte animal grand à des virus, tels que le syndrome dysgénésique et respiratoire porcin virus2. La figure 2 montre l’emplacement d’un sous-ensemble de ces principaux ganglions lymphatiques dans le corps de bovins.
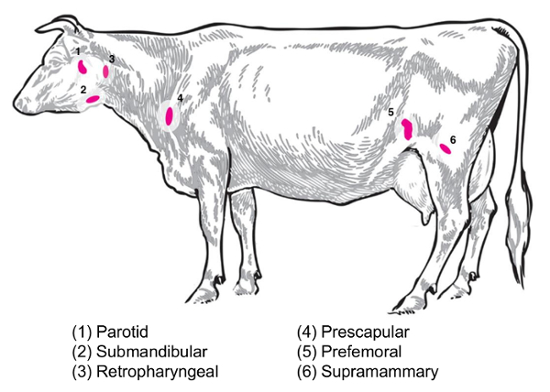
Figure 2 : Caricature illustrant certains ganglions lymphatiques emplacements dans Bos taurus . Les ganglions lymphatiques numérotées sont annotées. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Dans cet article et la vidéo associée, nous présentons un protocole pour l’isolement des gros animaux ganglions lymphatiques pour des études de RNA, conçues pour être instructif pour les biologistes moléculaires participant aux études de transcriptomique des grandes infections animales. Tout d’abord, nous donnent un aperçu de la procédure d’isolement pour les ganglions lymphatiques, utilisant l’échantillonnage de tissus bovins et bisons comme exemples. Associé à cette manifestation, comme indiqué dans la vidéo, est un “workflow” pour un échantillon de tissu reproductible pour ARN. Ensuite, nous décrivons des considérations importantes pour le traitement d’un ganglion infecté, en mettant l’accent sur la sécurité, la cohérence et la qualité de la RNA.
La préparation de l’ARN du tissu avec un réactif d’isothiocyanate de phénol-guanidine acidifié est basée sur la méthode originale de Chomczynski et Sacchi16,17, avec une purification sur colonnes à centrifuger à base de silice en présence de agents chaotropiques basés sur le œuvre originale de Vogelstein et Gillespie18. Nous examinons également le potentiel pour la reprise de l’ARN pour transcriptomique de bovins ganglions lymphatiques conservés par des méthodes alternatives. Enfin, nous examinons l’impact de la variable de temps sur la qualité de RNA dans grands autopsies animales, y compris une expérience représentative montrant l’effet de l’augmentation dans le temps entre l’euthanasie et l’échantillonnage sur le profil de RNA récupéré de bison et bovines ganglions lymphatiques. Cet article sera utile non seulement pour les biologistes moléculaires, mais aussi de vétérinaires chercheurs commençant des études de transcriptomique.
Protocol
Representative Results
Discussion
La majorité des études transcriptomique et les protocoles associés se consacrée sur la souris, le rat ou post-mortem des échantillons humains. Cependant, enquêtes du bétail et la faune offrent un large éventail de possibilités pour la caractérisation de la réponse immunitaire à la maladie, tant qu’il y a lieu, à la médecine vétérinaire et, en ce qui concerne les maladies zoonotiques, pour la santé publique humaine. Ce protocole a donné un aperçu des considérations clés pour l’extraction de l’AR…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Les auteurs tiennent à remercier James Fosse pour son excellent travail sur tous les vidéographie et traitement vidéo ; Michael Marti pour son excellent travail dans la génération des images numérisées de bétail ; Lilia Walther pour son aide avec l’extraction de l’ARN et Bioanalyzer s’exécute ; Mitch Palmer et Carly Kanipe pour leur examen utile et commentaires sur les images de ganglion lymphatique ; et le soin des animaux et du personnel vétérinaire à l’Animal National Disease Center pour l’ensemble de leur travail acharné et l’aide à l’élevage et la préparation des autopsies.
Materials
| RNA preservation solution (we used RNALater for all experiments) | ThermoFisher | AM7020 | |
| 1.5 ml or 2 ml polypropylene microcentrifuge tubes | Fisher Scientific | 05-408-129 | |
| Disposable scalpels | Daigger Scientific | EF7281 | |
| Tissue forceps, rat tooth | Fisher Scientific | 12-460-117 | Other tissue forceps available including curved tip, tapered edge, etc. , depends on user preference |
| 3 mm punch biopsy needles | Fisher Scientific | NC9949469 | |
| Sharps container (small and transportable for necropsy) | Stericycle | 8900SA | 1 qt. size shown here |
| Cutting boards or disposable trays | Fisher Scientific | 09-002-24A | Available in a variety of sizes, depends on user preference |
| Personal protective equipment | Varies with pathogen (gloves, respirator masks, goggles, etc.) | ||
| Phenol-based RNA extraction reagent (we used TRIzol Reagent for all experiments) | ThermoFisher | 15596026 | |
| Silica column-based RNA extraction kit (we used the PureLink RNA Mini kit for all experiments) | ThermoFisher | 12183018A | Designed for up to 100 mg tissue |
| 100% Ethanol (200 proof for molecular biology) | Sigma-Aldrich | E7023 | |
| Tissue homogenizer with enclosed homogenization tubes (we used the gentleMACS dissociator for all experiments) | Miltenyi Biotec | 130-093-235 | |
| Agarose (General, for gel electrophoresis) | Sigma-Aldrich | A9539 | |
| 1X TBE | Fisher Scientific | BP24301 | Can also make from scratch in the laboratory |
| Deionized formamide | EMD Millipore | S4117 | |
| Sodium dodecyl sulfate | Sigma-Aldrich | L3771 | |
| Bromophenol blue | Sigma-Aldrich | 114391 | |
| Xylene cyanol | Sigma-Aldrich | X4126 | |
| EDTA (Ethylenediaminetetraacetic acid) | Sigma-Aldrich | EDS | |
| UV-Vis Spectrophotometer (we used the NanoDrop Spectrophotometer) | ThermoFisher | ND-2000 | |
| Device for quantitative RNA assessment (we used the Bioanalyzer, with associated components and protocols) | Agilent | G2939BA | |
| FFPE RNA extraction kit (we used the RecoverAll Total Nucleic Acid Isolation Kit for Formalin Fixed, Paraffin Embedded Tissue) | ThermoFisher | AM1975 | |
| Plastic spreader (L-shaped spreader) | Fisher Scientific | 14-665-231 | Only needed for sterility testing for samples from infected animals |
| Necropsy knives | Livestock Concepts | WI-0009209 |
References
- Tizioto, P. C., et al. Immunological response to single pathogen challenge with agents of the bovine respiratory disease complex: an RNA-sequence analysis of the bronchial lymph node transcriptome. PLoS One. 10 (6), e0131459 (2015).
- Miller, L. C., et al. Analysis of the swine tracheobronchial lymph node transcriptomic response to infection with a Chinese highly pathogenic strain of porcine reproductive and respiratory syndrome virus. BMC Veterinary Research. 8, 208 (2012).
- Silva, T. M. A., Costa, E. A., Paixao, T. A., Tsolis, R. M., Santos, R. L. Laboratory animal models for brucellosis research. Journal of Biomedicine and Biotechnology. 2011, 518323 (2011).
- Willard-Mack, C. L. Normal structure, function, and histology of the lymph nodes. Toxicologic Pathology. 34, 409-424 (2006).
- Elmore, S. A. Histopathology of the lymph nodes. Toxicologic Pathology. 34 (5), 425-454 (2006).
- Luscieti, P., Hubschmid, T. h., Cottier, H., Hess, M. W., Sobin, L. H. Human lymph node morphology as a function of age and site. Journal of Clinical Pathology. 33 (5), 454-461 (1980).
- Hadamitsky, C., et al. Age-dependent histoarchitectural changes in human lymph nodes: an underestimated process with clinical relevance?. Journal of Anatomy. 216 (5), 556-562 (2010).
- Sisson, S., Grossman, J. D., Getty, R. . Sisson and Grossman’s The Anatomy of Domestic Animals, Volume 1. , (1975).
- Olsen, S. C., Johnson, C. Comparison of abortion and infection after experimental challenge of pregnant bison and cattle with Brucella abortus strain 2308. Clinical and Vaccine Immunology. 18 (12), 2075-2078 (2011).
- Brichta-Harhay, D. M., et al. Microbiological analysis of bovine lymph nodes for the detection of Salmonella enterica. Journal of Food Protection. 75 (5), 854-858 (2012).
- Samuel, J. L., O’Boyle, D. A., Mathers, W. J., Frost, A. J. Isolation of Salmonella from mesenteric lymph nodes of healthy cattle at slaughter. Research in Veterinary Science. 28 (2), 238-241 (1980).
- Arthur, T. M., et al. Prevalence and characterization of Salmonella in bovine lymph nodes potentially destined for use in ground beef. Journal of Food Protection. 71 (8), 1685-1688 (2008).
- Cray, W. C., Moon, H. W. Experimental infection of calves and adult cattle with Escherichia coli O157:H7. Applied and Environmental Microbiology. 61 (4), 1586-1590 (1995).
- Thiermann, A. B. Experimental leptospiral infections in pregnant cattle with organisms of the Hebdomadis serogroup. American Journal of Veterinary Research. 43 (5), 780-784 (1982).
- Palmer, M. V., Waters, W. R., Whipple, D. L. Investigation of the transmission of Mycobacterium bovis from deer to cattle through indirect contact. American Journal of Veterinary Research. 65 (11), 1483-1489 (2004).
- Chomczynski, P. A reagent for the single-step simultaneous isolation of RNA, DNA, and proteins from cell and tissue samples. BioTechniques. 15 (3), 532-537 (1993).
- Chomczynski, P., Sacchi, N. Single step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction. Analytical Biochemistry. 162 (1), 156-159 (1987).
- Vogelstein, B., Gillespie, D. Preparative and analytical purification of DNA from agarose. Proceedings of the National Academy of Sciences of the United States of America. 76 (2), 615-619 (1979).
- . . AVMA Guidelines for the Euthanasia of Animals. , (2013).
- . . TRIzol Reagent Manual. , (2016).
- . . Disruption and Homogenization of Tissue for the Extraction of RNA. , (2014).
- . . PureLink RNA Mini Kit Protocol . , (2012).
- . . RNA Gel-loading Buffer. , (2006).
- Rio, D. C., Ares, M., Hannon, G. J., Nilsen, T. W. Nondenaturing Agarose Gel Electrophoresis of RNA. Cold Spring Harbor Protocols. , (2010).
- Mueller, O., et al. A microfluidic system for high-speed reproducible DNA sizing and quantitation. Electrophoresis. 21 (1), 128-134 (2000).
- Schroeder, A., et al. The RIN: an RNA integrity number for assigning integrity values to RNA measurements. BMC Molecular Biology. 7 (3), (2006).
- Suvarna, K. S., Layton, C., Bancroft, J. D. . Bancroft’s Theory and Practice of Histological Techniques. , (2012).
- Medawar, P. B. The rate of penetration of fixatives. Journal of Microscopy. 61 (1-2), 46-57 (1941).
- Canene-Adams, K. Preparation of formalin-fixed paraffin-embedded tissue for immunohistochemistry. Methods in Enzymology. 533, 225-233 (2013).
- Fleige, S., Pfaffl, M. W. RNA integrity and the effect on the real-time qRT-PCR performance. Molecular Aspects of Medicine. 27, 126-139 (2006).
- Fajardy, I., et al. Time course analysis of RNA stability in human placenta. BMC Molecular Biology. 10 (21), (2009).
- Bahar, B., et al. Long-term stability of RNA in post-mortem bovine skeletal muscle, liver and subcutaneous adipose tissues. BMC Molecular Biology. 8, 108 (2007).
- Yamagishi, A., et al. Gene profiling and bioinformatics analyses reveal time course differential gene expression in surgically resected colorectal tissues. Oncology Reports. 31 (4), 1532-1538 (2014).
- Choi, S., Ray, H. E., Lai, S. H., Alwood, J. S., Globus, R. K. Preservation of multiple mammalian tissues to maximize science return from ground based and spaceflight experiments. PLoS One. 11 (12), e0167391 (2016).
- Almeida, A., Thiery, J. P., Magdelenat, H., Radvanyi, F. Gene expression analysis by real-time reverse transcription polymerase chain reaction: influence of tissue handling. Analytical Biochemistry. 328 (2), 101-108 (2004).
- Lee, S. M. L., Schelcher, C., Thasler, R., Schiergens, T. S., Thasler, W. E. Pre-analytical determination of the effect of extended warm or cold ischaemia on RNA stability in the human ileum mucosa. PLoS One. 10 (9), e0138214 (2015).
- Kap, M., et al. The influence of tissue procurement procedures on RNA integrity, gene expression, and morphology in porcine and human liver tissue. Biopreservation and Biobanking. 13 (3), 200-206 (2015).
- Hong, S. H., et al. Effects of delay in the snap freezing of colorectal cancer tissues on the quality of DNA and RNA. Journal of the Korean Society of Coloproctology. 26 (5), 316-325 (2010).
- Lee, S. M., et al. RNA stability in human liver: comparison of different processing times, temperatures and methods. Molecular Biotechnology. 53 (1), 1-8 (2013).
- Morrison, P. K., et al. Post-mortem stability of RNA in skeletal muscle and adipose tissue and the tissue-specific expression of myostatin, perilipin and associated factors in the horse. PLoS One. 9 (6), e100810 (2014).
- Seear, P. J., Sweeney, G. E. Stability of RNA isolated from post-mortem tissues of Atlantic salmon (Salmo salar L.). Fish Physiology and Biochemistry. 34 (1), 19-24 (2008).
- Marchuk, L., Sciore, P., Reno, C., Frank, C. B., Hart, D. A. Postmortem stability of total RNA isolated from rabbit ligament, tendon, and cartilage. Biochimica et Biophysica Acta. 1379 (2), 171-177 (1998).
- Inoue, H., Kimura, A., Tuji, T. Degradation profile of mRNA in a dead rat body: basic semi-quantification study. Forensic Science International. 130 (2-3), 127-132 (2002).
- Micke, P., et al. Biobanking of fresh frozen tissue: RNA is stable in nonfixed surgical specimens. Laboratory Investigation. 86 (2), 202-211 (2006).
- Srinivasan, M., Sedmak, D., Jewell, S. Effect of fixatives and tissue processing on the content and integrity of nucleic acids. The American Journal of Pathology. 161 (6), 1961-1971 (2002).
- Ahmed, A. M., et al. Variation in the ovine abomasal lymph node transcriptome between breeds known to differ in resistance to the gastrointestinal nematode. PLoS One. 10 (5), e0124823 (2015).
- Russell, G. C., et al. Host gene expression changes in cattle infected with Alcelaphine herpesvirus 1. Virus Research. 169 (1), 246-254 (2012).
- Avraham, R., et al. A highly multiplexed and sensitive RNA-seq protocol for simultaneous analysis of host and pathogen transcriptomes. Nature Protocols. 11, 1477-1491 (2016).

