Raccolta e trattamento dei linfonodi da grandi animali per l'analisi del RNA: preparazione per gli studi di trascrittomica di linfonodo di grandi specie animali
Summary
Questo protocollo fornisce una panoramica delle procedure per l’isolamento di RNA per la profilazione di trascrittomica di linfonodo tessuti da grandi animali, compresi i passaggi nell’identificazione e l’asportazione dei linfonodi da bestiame e animali selvatici, approcci di campionamento per garantire la consistenza attraverso più animali e considerazioni più risultati rappresentativi per la conservazione post-raccolta e l’elaborazione per l’analisi del RNA.
Abstract
Grandi animali (bestiame e animali selvatici) servono come importanti serbatoi di agenti patogeni zoonotici, compreso Brucella, Mycobacterium bovis, Salmonellaed e. colie sono utili per lo studio della patogenesi e/o diffusione dei batteri negli ospiti naturali. Con la funzione chiave di linfonodi nella risposta immunitaria ospite, linfonodo tessuti servire come una potenziale fonte di RNA per analisi trascrittomica a valle, al fine di valutare i cambiamenti temporali nell’espressione genica in cellule nel corso di un’infezione. Questo articolo presenta una panoramica del processo di raccolta di linfonodo, campioni tissutali e a valle RNA processing in bestiame, usando bovini (Bos taurus) come un modello, con ulteriori esempi forniti dal bisonte americano (bisonte del bisonte di ). Il protocollo comprende le informazioni relative all’ubicazione, identificazione e rimozione dei linfonodi da più siti chiave nel corpo. Inoltre, è presentata una metodologia di prelievo bioptico che consente per una consistenza di campionamento attraverso più animali. Diverse considerazioni per la conservazione del campione sono discusse, compreso la generazione di RNA adatto a valle metodologie come RNA-sequenziamento e RT-PCR. A causa dei lunghi ritardi insiti nella grande animale vs mouse tempo corso di studi, risultati rappresentativi da bison e tessuti bovini linfonodo sono presentati per descrivere il corso di tempo del degrado in questo tipo di tessuto, nel contesto di una revisione del precedenti lavori metodologici sulla degradazione del RNA in altri tessuti. Nel complesso, questo protocollo sarà utile per entrambi ricercatori veterinari inizio transcriptome progetti su grandi campioni di animali e per i biologi molecolari sono interessati ad apprendere tecniche per in vivo campioni tissutali e trattamento in vitro .
Introduction
RNA-ordinando l’analisi del trascrittoma di linfonodi offre l’opportunità di caratterizzare la risposta immunitaria degli animali ad una varietà di agenti patogeni. Mentre questa metodologia è stata utilizzata estesamente in topi, analisi recentemente sono state espandendo in più grandi mammiferi1,2. Bestiame/grande animale dei linfonodi può essere utilizzati per caratterizzare la risposta dell’ospite all’infezione, non solo per il loro uso nel vaccino o gli studi di predisposizione genetica e per l’identificazione di bersagli per lo sviluppo di farmaci, ma anche come sistemi modello per studi umani su malattie zoonotiche. Ad esempio, nel caso di brucellosi (una malattia zoonotica batterica che impatti mezzo milione di persone nel mondo ogni anno), nonostante ha aumentato significativamente i costi, gli studi in pecore o capre sono più rilevanti per l’infezione umana e vaccino umano sviluppo di modelli animali di laboratorio. Modelli murini di infezione ricapitolano l’infezione del sistema reticoloendoteliale ma non i segni clinici caratteristici3.
Negli esperimenti sugli animali grandi rispetto agli studi sugli animali di laboratorio, il processo di tessuto raccolta necessariamente comporta un ritardo maggiore tra l’eutanasia e l’insieme del tessuto, che presenta una sfida per la conservazione della RNA di alta qualità. Il RNA intatto è essenziale per la generazione di dati di trascrittomica biologicamente rilevanti. La generazione di RNA di alta qualità da campioni di tessuto è particolarmente critica per gli studi di grande agente patogeno animale condotto in strutture di contenimento. Tali studi sono intrinsecamente più difficili da eseguire come non solo richiedono impianti autorizzati e personale altamente qualificato, ma anche portare significativi costi finanziari, che, a seconda del lavoro, possono variare da decine a centinaia di migliaia di dollari. Questi tipi di studi implicano anche una collaborazione interdisciplinare e conoscenza interdisciplinare per il loro completamento, aggiungendo alla loro complessità. Di conseguenza, formazione, sviluppo di e il rispetto di un sistema semplificato per la raccolta dei campioni e conservazione offre notevoli vantaggi per gli studi molecolari a valle dei tessuti da animali infetti.
La collezione dei linfonodi più grande presenta ulteriori sfide per la raccolta di tessuto rispetto al simile campionamento dei linfonodi murini. La preparazione per l’asportazione di campione richiede una conoscenza di base dell’anatomia del linfonodo, comprese le pertinenti strutture interne. La struttura di un linfonodo è costituita da lobuli linfoidi circondati da seni pieni di linfa. Queste strutture sono racchiuse da una capsula fibrosa, dura. 4 un lobulo linfoide è il “anatomica e funzionale unità base del linfonodo” ed è composto di follicoli, un’unità profonda corticale e cordoni midollari e seni4 (Figura 1A). I linfociti B e T sono sede del follicoli e corticale profonda unità, rispettivamente. Queste strutture forniscono un ponteggio 3D e facilitano l’interazione tra linfociti e cellule presentanti l’antigene o antigene.
Grossolanamente, follicoli e unità profonda corticale può essere identificato sulla superficie di taglio, dato che contengono un denso reticolo reticolare e appaiono più scuri rispetto i seni, che sono costituiti da un reticolo reticolare più delicato e appaiono più chiare (Figura 1B). Per convenzione, i patologi si riferiscono alle regioni dei linfonodi come la corteccia superficiale (follicoli), la paracorteccia (profondità corticale unità) e il midollo (cordoni midollari e seni paranasali). Un esame adeguato di tutte le tre regioni è stato considerato come migliore pratica negli orientamenti per l’esame patologico sistematico per linfonodi5. Nota che c’è una considerevole variazione nella consistenza, dimensione e il colore dei linfonodi, anche all’interno di un singolo animale. Come età animali, loro linfonodi tenderà a diminuire di dimensioni e diventare più forte di quelli degli animali più giovani, in genere a causa di un aumento nel loro tessuto connettivo e una riduzione del normale linfoide struttura6,7.
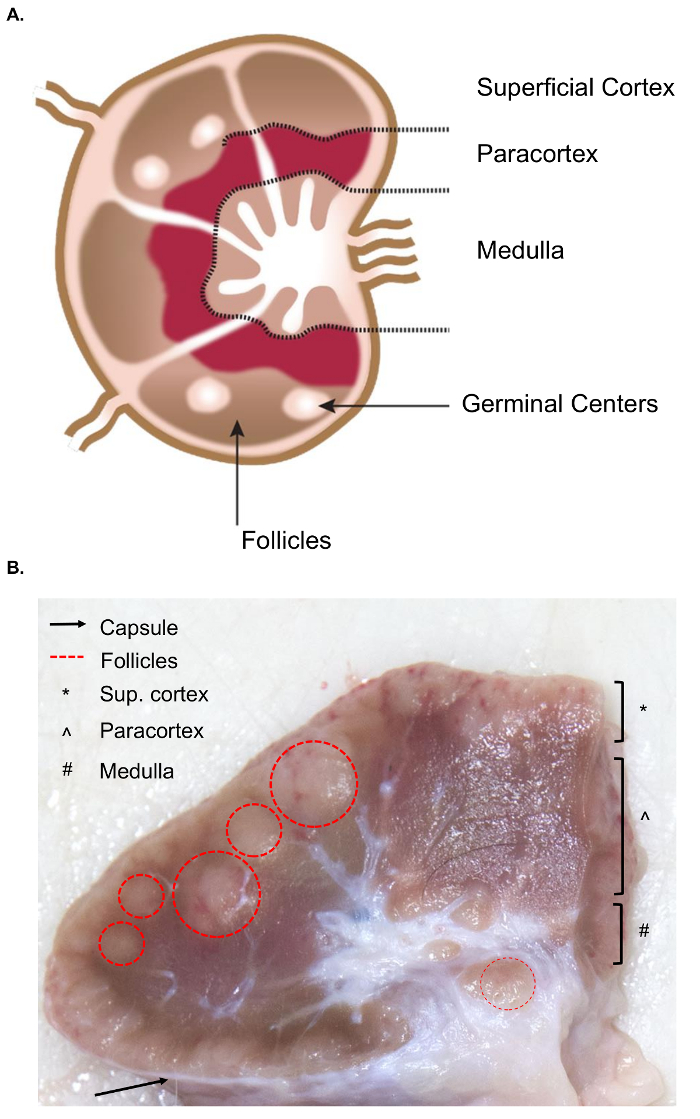
Figura 1. Anatomia del linfonodo. (A) questa immagine del fumetto Mostra l’anatomia del linfonodo, raffiguranti strutture chiave. (B) questo fermo immagine mostra un linfonodo bovino tagliato in sezione trasversale. Sono evidenziati le pertinenti strutture/strati che sono visibili ad occhio nudo. Clicca qui per visualizzare una versione più grande di questa figura.
A seconda della domanda sperimentale, vari linfonodi sarà di interesse per la raccolta e l’analisi. Linfonodi periferici sono quelli situati nel tessuto sottocutaneo profondo. Nei bovini, linfonodi periferici o superficiale, spesso utilizzati nella pratica clinica e sperimentale includono parotide, sottomandibolare, retropharyngeal, -prescapolari, prefemorali (precrural) e superficiale inguinale (soprammammari in femmine, scrotale nei maschi) ( Figura 2). In tabella 1, sono descritte le proprietà della chiave linfonodi superficiali, come descritto nel bestiame sistema8. Sotto, alcuni potenziali piani di raccolta di linfonodo per malattie infettive batteriche del bestiame sono presentati come punto di partenza per l’indagine.
Brucella abortus/Brucella melitensis: Standard necropsies per b. abortus-infetti bestiame e b. melitensis-capre infette al National Animal Disease Center recupero tessuto linfonodo parotideo, prescapular e soprammammari , sia per la macinazione per l’enumerazione batterica e per la preparazione di RNA per il delineamento di espressione host RNA. B. abortus possono essere recuperati regolarmente in ciascuno di questi linfonodi in bestiami infettati sperimentalmente9. La presenza di batteri in ognuno di questi tipi di linfonodo può essere rilevata in b. melitensis-infettati capre fino a post-infezione almeno nove mesi, usando le metodologie basate su RNA dai nostri studi (Boggiatto et al., non pubblicato). Salmonella SP.: prescapular, subiliac (prefemorali), e i linfonodi mesenterici sono stati utili durante la profilatura delle carcasse di bovini per una prevalenza di Salmonella 10,11,12 e sarebbe di potenziale interesse per gli studi di trascrittomica. Escherichia coli O157: H7: linfonodi mesenterici (a medio piccolo intestino e intestino tenue distale posizioni) possono essere i siti di un recupero occasionale dei batteri in vitelli infetti (ma non in bovini adulti infetti)13. Leptospirosi (Leptospira SP.): una persistenza cronica dei batteri è stata osservata nei linfonodi drenanti la ghiandola mammaria14. Mycobacterium bovis : Nei bovini, i batteri sono stati recuperati di infezione post-sperimentale dai linfonodi mediastinici e tracheobronchial vitelli15. Inoltre, RNA di linfonodo è stato utilizzato per esaminare le risposte di grande animale ospite ai virus, come la sindrome riproduttiva e respiratoria del suino virus2. Figura 2 descrive la posizione di un sottoinsieme di questi linfonodi principali nel corpo del bestiame.
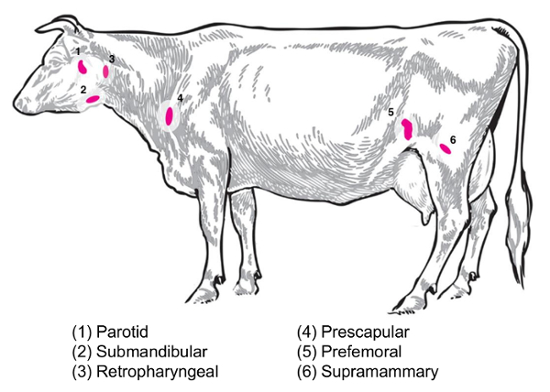
Figura 2: Cartone animato raffigurante località selezionata di linfonodo in Bos taurus . I linfonodi numerati vengono annotati. Clicca qui per visualizzare una versione più grande di questa figura.
In questo articolo e il video associato, presentiamo un protocollo per l’isolamento dei linfonodi grandi animali per studi di RNA, progettati per essere informativo per i biologi molecolari coinvolti negli studi di trascrittomica di grandi infezioni degli animali. In primo luogo, forniamo una panoramica della procedura di isolamento per i linfonodi, mediante campionamento dai tessuti bovini e bisonte come esempi. Accoppiato con questa dimostrazione, come mostrato nel video, è un flusso di lavoro per un campione del tessuto riproducibile per isolamento del RNA. Descriveremo inoltre considerazioni importanti per l’elaborazione di un linfonodo infetto, con un focus sulla sicurezza, la coerenza e la qualità di RNA.
La preparazione di RNA dal tessuto con un reagente di isotiocianato di fenolo-guanidina acidificato è basata sul metodo originale di Chomczynski e Sacchi16,17, con una purificazione su colonne di spin a base di silice in presenza di agenti caotropici basati sul lavoro originale di Vogelstein e Gillespie18. Esaminiamo anche il potenziale per il recupero di RNA per trascrittomica dai linfonodi bestiame conservati con metodi alternativi. Infine, abbiamo esplorare l’impatto della variabile tempo sulla qualità di RNA in grande autopsie animali, tra cui un esperimento rappresentanza raffigurante l’effetto di un aumento nel tempo tra l’eutanasia e il campionamento sul profilo del RNA recuperato da bison e bovina nei linfonodi. Questo articolo sarà utile non solo per i biologi molecolari, ma anche a veterinari ricercatori che inizia gli studi di trascrittomica.
Protocol
Representative Results
Discussion
La maggior parte dei studi di trascrittomica e dei relativi protocolli concentrarsi su campioni umani, mouse, ratto o post mortem. Tuttavia, le indagini in bestiame e animali selvatici forniscono una vasta gamma di opportunità per la caratterizzazione della risposta immune alla malattia, sia come applicabile alla medicina veterinaria e, per quanto riguarda le malattie zoonotiche, per la salute umana. Questo protocollo prevedeva un contorno di considerazioni chiave per l’estrazione di RNA ad alta integrità dai tessuti d…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Gli autori vorrei ringraziare James Fosse per il suo eccellente lavoro su tutti i Videografia ed elaborazione video; Michael Marti per il suo eccellente lavoro nella generazione di immagini digitalizzate bestiame; Lilia Walther per il suo aiuto con estrazione del RNA e Bioanalyzer viene eseguito; Mitch Palmer e Carly Kanipe per la loro recensione utile e feedback sulle immagini di linfonodo; e la cura degli animali e il personale veterinario presso il National Animal Disease Center per tutti i loro duro lavoro e assistenza con la zootecnia e la preparazione per autopsie.
Materials
| RNA preservation solution (we used RNALater for all experiments) | ThermoFisher | AM7020 | |
| 1.5 ml or 2 ml polypropylene microcentrifuge tubes | Fisher Scientific | 05-408-129 | |
| Disposable scalpels | Daigger Scientific | EF7281 | |
| Tissue forceps, rat tooth | Fisher Scientific | 12-460-117 | Other tissue forceps available including curved tip, tapered edge, etc. , depends on user preference |
| 3 mm punch biopsy needles | Fisher Scientific | NC9949469 | |
| Sharps container (small and transportable for necropsy) | Stericycle | 8900SA | 1 qt. size shown here |
| Cutting boards or disposable trays | Fisher Scientific | 09-002-24A | Available in a variety of sizes, depends on user preference |
| Personal protective equipment | Varies with pathogen (gloves, respirator masks, goggles, etc.) | ||
| Phenol-based RNA extraction reagent (we used TRIzol Reagent for all experiments) | ThermoFisher | 15596026 | |
| Silica column-based RNA extraction kit (we used the PureLink RNA Mini kit for all experiments) | ThermoFisher | 12183018A | Designed for up to 100 mg tissue |
| 100% Ethanol (200 proof for molecular biology) | Sigma-Aldrich | E7023 | |
| Tissue homogenizer with enclosed homogenization tubes (we used the gentleMACS dissociator for all experiments) | Miltenyi Biotec | 130-093-235 | |
| Agarose (General, for gel electrophoresis) | Sigma-Aldrich | A9539 | |
| 1X TBE | Fisher Scientific | BP24301 | Can also make from scratch in the laboratory |
| Deionized formamide | EMD Millipore | S4117 | |
| Sodium dodecyl sulfate | Sigma-Aldrich | L3771 | |
| Bromophenol blue | Sigma-Aldrich | 114391 | |
| Xylene cyanol | Sigma-Aldrich | X4126 | |
| EDTA (Ethylenediaminetetraacetic acid) | Sigma-Aldrich | EDS | |
| UV-Vis Spectrophotometer (we used the NanoDrop Spectrophotometer) | ThermoFisher | ND-2000 | |
| Device for quantitative RNA assessment (we used the Bioanalyzer, with associated components and protocols) | Agilent | G2939BA | |
| FFPE RNA extraction kit (we used the RecoverAll Total Nucleic Acid Isolation Kit for Formalin Fixed, Paraffin Embedded Tissue) | ThermoFisher | AM1975 | |
| Plastic spreader (L-shaped spreader) | Fisher Scientific | 14-665-231 | Only needed for sterility testing for samples from infected animals |
| Necropsy knives | Livestock Concepts | WI-0009209 |
References
- Tizioto, P. C., et al. Immunological response to single pathogen challenge with agents of the bovine respiratory disease complex: an RNA-sequence analysis of the bronchial lymph node transcriptome. PLoS One. 10 (6), e0131459 (2015).
- Miller, L. C., et al. Analysis of the swine tracheobronchial lymph node transcriptomic response to infection with a Chinese highly pathogenic strain of porcine reproductive and respiratory syndrome virus. BMC Veterinary Research. 8, 208 (2012).
- Silva, T. M. A., Costa, E. A., Paixao, T. A., Tsolis, R. M., Santos, R. L. Laboratory animal models for brucellosis research. Journal of Biomedicine and Biotechnology. 2011, 518323 (2011).
- Willard-Mack, C. L. Normal structure, function, and histology of the lymph nodes. Toxicologic Pathology. 34, 409-424 (2006).
- Elmore, S. A. Histopathology of the lymph nodes. Toxicologic Pathology. 34 (5), 425-454 (2006).
- Luscieti, P., Hubschmid, T. h., Cottier, H., Hess, M. W., Sobin, L. H. Human lymph node morphology as a function of age and site. Journal of Clinical Pathology. 33 (5), 454-461 (1980).
- Hadamitsky, C., et al. Age-dependent histoarchitectural changes in human lymph nodes: an underestimated process with clinical relevance?. Journal of Anatomy. 216 (5), 556-562 (2010).
- Sisson, S., Grossman, J. D., Getty, R. . Sisson and Grossman’s The Anatomy of Domestic Animals, Volume 1. , (1975).
- Olsen, S. C., Johnson, C. Comparison of abortion and infection after experimental challenge of pregnant bison and cattle with Brucella abortus strain 2308. Clinical and Vaccine Immunology. 18 (12), 2075-2078 (2011).
- Brichta-Harhay, D. M., et al. Microbiological analysis of bovine lymph nodes for the detection of Salmonella enterica. Journal of Food Protection. 75 (5), 854-858 (2012).
- Samuel, J. L., O’Boyle, D. A., Mathers, W. J., Frost, A. J. Isolation of Salmonella from mesenteric lymph nodes of healthy cattle at slaughter. Research in Veterinary Science. 28 (2), 238-241 (1980).
- Arthur, T. M., et al. Prevalence and characterization of Salmonella in bovine lymph nodes potentially destined for use in ground beef. Journal of Food Protection. 71 (8), 1685-1688 (2008).
- Cray, W. C., Moon, H. W. Experimental infection of calves and adult cattle with Escherichia coli O157:H7. Applied and Environmental Microbiology. 61 (4), 1586-1590 (1995).
- Thiermann, A. B. Experimental leptospiral infections in pregnant cattle with organisms of the Hebdomadis serogroup. American Journal of Veterinary Research. 43 (5), 780-784 (1982).
- Palmer, M. V., Waters, W. R., Whipple, D. L. Investigation of the transmission of Mycobacterium bovis from deer to cattle through indirect contact. American Journal of Veterinary Research. 65 (11), 1483-1489 (2004).
- Chomczynski, P. A reagent for the single-step simultaneous isolation of RNA, DNA, and proteins from cell and tissue samples. BioTechniques. 15 (3), 532-537 (1993).
- Chomczynski, P., Sacchi, N. Single step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction. Analytical Biochemistry. 162 (1), 156-159 (1987).
- Vogelstein, B., Gillespie, D. Preparative and analytical purification of DNA from agarose. Proceedings of the National Academy of Sciences of the United States of America. 76 (2), 615-619 (1979).
- . . AVMA Guidelines for the Euthanasia of Animals. , (2013).
- . . TRIzol Reagent Manual. , (2016).
- . . Disruption and Homogenization of Tissue for the Extraction of RNA. , (2014).
- . . PureLink RNA Mini Kit Protocol . , (2012).
- . . RNA Gel-loading Buffer. , (2006).
- Rio, D. C., Ares, M., Hannon, G. J., Nilsen, T. W. Nondenaturing Agarose Gel Electrophoresis of RNA. Cold Spring Harbor Protocols. , (2010).
- Mueller, O., et al. A microfluidic system for high-speed reproducible DNA sizing and quantitation. Electrophoresis. 21 (1), 128-134 (2000).
- Schroeder, A., et al. The RIN: an RNA integrity number for assigning integrity values to RNA measurements. BMC Molecular Biology. 7 (3), (2006).
- Suvarna, K. S., Layton, C., Bancroft, J. D. . Bancroft’s Theory and Practice of Histological Techniques. , (2012).
- Medawar, P. B. The rate of penetration of fixatives. Journal of Microscopy. 61 (1-2), 46-57 (1941).
- Canene-Adams, K. Preparation of formalin-fixed paraffin-embedded tissue for immunohistochemistry. Methods in Enzymology. 533, 225-233 (2013).
- Fleige, S., Pfaffl, M. W. RNA integrity and the effect on the real-time qRT-PCR performance. Molecular Aspects of Medicine. 27, 126-139 (2006).
- Fajardy, I., et al. Time course analysis of RNA stability in human placenta. BMC Molecular Biology. 10 (21), (2009).
- Bahar, B., et al. Long-term stability of RNA in post-mortem bovine skeletal muscle, liver and subcutaneous adipose tissues. BMC Molecular Biology. 8, 108 (2007).
- Yamagishi, A., et al. Gene profiling and bioinformatics analyses reveal time course differential gene expression in surgically resected colorectal tissues. Oncology Reports. 31 (4), 1532-1538 (2014).
- Choi, S., Ray, H. E., Lai, S. H., Alwood, J. S., Globus, R. K. Preservation of multiple mammalian tissues to maximize science return from ground based and spaceflight experiments. PLoS One. 11 (12), e0167391 (2016).
- Almeida, A., Thiery, J. P., Magdelenat, H., Radvanyi, F. Gene expression analysis by real-time reverse transcription polymerase chain reaction: influence of tissue handling. Analytical Biochemistry. 328 (2), 101-108 (2004).
- Lee, S. M. L., Schelcher, C., Thasler, R., Schiergens, T. S., Thasler, W. E. Pre-analytical determination of the effect of extended warm or cold ischaemia on RNA stability in the human ileum mucosa. PLoS One. 10 (9), e0138214 (2015).
- Kap, M., et al. The influence of tissue procurement procedures on RNA integrity, gene expression, and morphology in porcine and human liver tissue. Biopreservation and Biobanking. 13 (3), 200-206 (2015).
- Hong, S. H., et al. Effects of delay in the snap freezing of colorectal cancer tissues on the quality of DNA and RNA. Journal of the Korean Society of Coloproctology. 26 (5), 316-325 (2010).
- Lee, S. M., et al. RNA stability in human liver: comparison of different processing times, temperatures and methods. Molecular Biotechnology. 53 (1), 1-8 (2013).
- Morrison, P. K., et al. Post-mortem stability of RNA in skeletal muscle and adipose tissue and the tissue-specific expression of myostatin, perilipin and associated factors in the horse. PLoS One. 9 (6), e100810 (2014).
- Seear, P. J., Sweeney, G. E. Stability of RNA isolated from post-mortem tissues of Atlantic salmon (Salmo salar L.). Fish Physiology and Biochemistry. 34 (1), 19-24 (2008).
- Marchuk, L., Sciore, P., Reno, C., Frank, C. B., Hart, D. A. Postmortem stability of total RNA isolated from rabbit ligament, tendon, and cartilage. Biochimica et Biophysica Acta. 1379 (2), 171-177 (1998).
- Inoue, H., Kimura, A., Tuji, T. Degradation profile of mRNA in a dead rat body: basic semi-quantification study. Forensic Science International. 130 (2-3), 127-132 (2002).
- Micke, P., et al. Biobanking of fresh frozen tissue: RNA is stable in nonfixed surgical specimens. Laboratory Investigation. 86 (2), 202-211 (2006).
- Srinivasan, M., Sedmak, D., Jewell, S. Effect of fixatives and tissue processing on the content and integrity of nucleic acids. The American Journal of Pathology. 161 (6), 1961-1971 (2002).
- Ahmed, A. M., et al. Variation in the ovine abomasal lymph node transcriptome between breeds known to differ in resistance to the gastrointestinal nematode. PLoS One. 10 (5), e0124823 (2015).
- Russell, G. C., et al. Host gene expression changes in cattle infected with Alcelaphine herpesvirus 1. Virus Research. 169 (1), 246-254 (2012).
- Avraham, R., et al. A highly multiplexed and sensitive RNA-seq protocol for simultaneous analysis of host and pathogen transcriptomes. Nature Protocols. 11, 1477-1491 (2016).

