생체 외에서 교정 무료 단백질 호모-oligomerization 상업 계측 및 무료, 오픈 소스 밝기 분석 소프트웨어를 사용 하 여의 정량화
Summary
이 프로토콜 단백질 호모-oligomerization에서 체 외에 상업 조명 현미경 검사를 사용 하 여 형광 변동 분광학에 따라 측정을 위한 보정 없는 접근을 설명 합니다. 올바른 인수 설정 및 분석 방법을 표시 됩니다.
Abstract
수와 밝기 감지 단백질 호모-oligomerization에 대 한 교정 무료 형광 변동 분광학 (FFS) 기술입니다. 그것은 전통적인 confocal 현미경 디지털 감지기 장비를 사용 하 여 사용할 수 있습니다. 생체 외에서 기술 사용에 대 한 프로토콜 번호와 밝기를 dimerizing 약 AP20187의 추가 전후 mVenus 표시 FKBP12F36V의 oligomeric 상태를 정확 하 게 계량을 볼 수 있는 사용 사례에 의해 표시 됩니다. 정확한 현미경 수집 매개 변수를 사용 하 여 올바른 데이터 전처리 및 분석 방법의 중요성을 설명 합니다. 특히, photobleaching의 선택의 중요성은 강조 했다. 이 저렴 한 방법은 많은 생물학 문맥에서 단백질 단백질 상호 작용 연구에 사용할 수 있습니다.
Introduction
단백질-단백질 상호 작용 나 n 체 외
전통적으로, 결정학 및 cryo 전자 현미경 검사 법 (cryoEM)와 결합 하는 핵 자기 공명 실험은 정확 하 게 단백질의 3 차원 구조를 설명 하 고 그들의 기능을 추정 하는 것을 선택 하는 기술 그들의 높은 해상도 구조 세부 사항을 면밀히 그러나 단백질,, 정적 구조 하 시간과 공간에 다양 한 구조적 변화와 진동 받을 수 있습니다. 이 때문에 구조 결정학에서 정보 또는 CryoEM 데이터 (예: 분자 동역학 시뮬레이션 기술과 단일 분자) 다른 기술로 보완 될 필요가: 그 구조적 관련 단백질의 기능 변경 및 상호 작용, 그리고이 정보는 정적 구조에 존재. 내부 분자 역학 조사, 순서 기술 포스터 공명 에너지 전송 (smFRET) 단일 분자에 따라 매우 효과적인1있습니다. 이러한 방식을 복잡 한 매체에 있는 분자의 다른 부분 모집단을 평가할 수 있습니다. 이것은 매우 중요 하 고, 이러한 변경 신속 하 고 (즉, 두 번째 범위에 나노초) 데이터 수집 중에 발생.
두 가지 주요 방법을 감지 하 고 이러한 변화를 정량화 일반적으로 채택 된다: 솔루션 및 표면 immobilization 단백질. 감지 간의 분자 상호 작용의와 특히, 이합체 화, ligands에 의해 유도 된 과정에 대 한 smFRET은 항상는 최고의 도구가 아니다. 사실, 무서 워 거리 (≈10 nm)에 뿐만 아니라 두 개의 쌍 극 자 (기증자 및 수락자, χ2)의 방향에 따라 그리고 수락자의 흡수 스펙트럼2, 하지만 아마도이 마지막 조건 기증자 방출의 오버랩은 중요 한 하는 experimentalist 수 오른쪽 무서 워 몇 선택. 호모-이합체 화를 프로 빙에 대 한 smFRET의 특정 단점 관심사의 단백질의 라벨에서 온다: hetero smFRET, 이합체 화만 될 수 있습니다 감지 최대 50% (즉, hetero 무서 워만 기증자 수락자를 감지할 수 있을 것입니다 및 기증자 수락자 호모-이합체 하지 기증자 기증자 또는 수락자-수락자, 다른 50%는 이합체의). 형광 상관 분광학 (FCS) 및 파생 상품 (FCCS, 등3) 확인 단백질 보급 상수 및 바인딩 상수에서 생체 외에서 의 사용은 또 다른 대안 이다. 이러한 방식을 완전히 중 호모-이합체 화를 계량 수, 확산 입자의 FCS 한 측정 농도 확산, 반지름 및 확산 계수가 매우 저조한; 분자량에 의존 예를 들어 10 증가 분자량에만 확산 계수4에서 2.15 배 변화를 나타내며. 2 컬러 FCS 또는 FCCS, 호모 이합체의 50%만 위와 같은 이유로 볼 것입니다. 호모-이합체 화에서 생체 외에서 그리고 vivo에서 감지 하는 가장 실용적이 고 양적 접근은 호모 무서 워5 와 수와 명도 (N & B)6. 그 사실을 감안할 때 호모 무서 워 필요 이방성 값 (즉, 광학 요소/분석기 병렬 및 수직 분극 복구)의 특정 계측 복구, N & B 제공 됩니다 여기에 유리한 기술로 단백질 호모-이합체 화 및 집계 검색 합니다. 그것은 될 수 있다 생체 외에서 그리고 vivo에서 상업 설정 고용.
수와 밝기
N & B 최근 검토7되었습니다. 그 리뷰는 라이브 셀에서 기술의 응용 프로그램에 집중 했다. 그것은 가치가 재현이이 방정식으로 수학적 주의 여기 데이터에 적용 될 것입니다 수집 에 체 외. 첫째, 그것은 몇 가지 용어 및 수학 수량을 정의 하는 데 필요한:
- 엔터티가 바인딩되는 함께 분자의 집합입니다.
- 엔터티 밝기 ε (프레임) 당 단위 시간 당 방출 하는 광자의 수입니다.
- n 은 현재 엔터티 수입니다.
- 이미지 시리즈의 과정을 통해 주어진된 픽셀, 의 의미와 σ2 은 그것의 강도에 대 한 분산입니다.
다음,와 광자 계산 탐지기 모바일 엔터티 및 아무 배경 가정
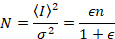
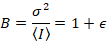
여기서 N 은 명백한 수 와 B 명백한 밝기입니다. 이 인해
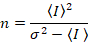
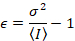
Dalal 외. 8 아날로그 장비와 함께 하나의 3 개의 교정 기간 요구 했다: S 요소, 배경 오프셋및 판독 잡음 σ02. 다음, 다시 모바일 엔터티, 가정
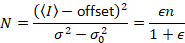
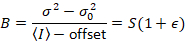
주는
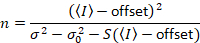
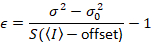
위의 방정식은 다른 Dalal 외 에 주어진 8 그리고 후속 검토입니다. 7 에 Dalal 외. S 분모에 오타 때문에 생략 되었다 그리고이 오류는 검토에서 재현 됐다. 위의 방정식 올바른 것 이다입니다. 지침 S측정, 오프셋 및 σ02 -그들의 의미-에 대 한 설명 Dalal 외 에 의해 주어진 다 8
밝기 ε 이다 oligomeric 확산 엔터티 상태에 비례: ε 그것은 단위체, 세 번 큰 trimers를 위한 trimers 등은 단위체, hexamers에 대 한 큰 두 번은 이합체에 대 한 큰 두 번 있을 것입니다. 이 방법에서는, 밝기 ε를 측정 하나 multimerization의 모든 종류를 계량 수 있습니다.
만약 oligomeric 국가의 혼합물, 수 있으며 밝기는 현재 개별 oligomeric 상태 복구의 수 있습니다. 이 기술의 한계입니다.
알고리즘 및 nandb 소프트웨어 detrend
Photobleaching는을 위한 교정의 중요성은 이전9를강조 했다. Photobleaching은 필연적으로 시간 경과 모드;에 가벼운 현미경 실험 중 발생 둘 다 라이브 세포에 생체 외에서. 많은 접근7표백에 대 한 올바른 문학에서 설명 했습니다. 트렌딩 매개 변수 T 의 자동 선택과 지 수 필터링 기술은 현재 최고입니다. 그것은 무료, 오픈 소스 소프트웨어 nandb9에 통합 된다. 실제로, 그들의 detrending 매개 변수를 수동으로 선택 하는 사용자를 필요로 하는 소프트웨어 임의적이 고 잘못 된 매개 변수 선택이 될 가능성이 있기 때문에 잘못 된 결과가 발생할 수 있습니다. 자동 알고리즘 데이터를 검사 하 고 사용자 개입9에 대 한 필요 없이, 그것에 대 한 적절 한 매개 변수를 결정 합니다. 부드럽게 매개 변수의 최선의 선택으로도 트렌딩는 한계가 하며 잘만 photobleaching 비율 25% 보다 낮은 시뮬레이션9와 같이 작동 합니다. 흥미롭게도, 자동 detrending 루틴을 사용 하 여, 그것의 정확도 하나 낮은 밝기 값을 사용할 수 있습니다 (심지어 B < 1.01), 따라서 낮은 농도, 그리고 아직 충분히 정확 하 게 계량 호모-이합체 화.
Photobleaching는 또한 또 다른 문제를 일으키는: multimer 복잡 한에 photobleached fluorophores의 존재. 이것은 예를 들어, 경우는 삼합체에 3 단위 중 하나 비 형광은 삼합체는 이합체 처럼 나타납니다. 허 고 뮬러10 이 해결 하는 방법 그리고이 수정 또한 후속 검토7에서 강조 되었다. Nandb 소프트웨어는이 보정9포함 되어 있습니다.
FKBP12 F36V 시스템
FKBP12F36V는 자연스럽 게 oligomerize 하지 않습니다 하지만 AP20187 약물 (구어체로 dimerizing ligand BB 라고도 함)11,12의 추가 따라 dimerize 알려져 있는 단백질 이다. 이것은 번호와 밝기에 대 한 이상적인 테스트 케이스: 이라는 FKBP12F36V와 oligomeric 상태의 두 배로 관찰 해야 BB의 추가에.
Protocol
Representative Results
Discussion
N & B multimerization 상업적인 빛 디지털 검출기를 장착 하는 confocal 현미경 검사를 사용 하 여 감지 하는 기술입니다. 이 방법은 단일 지점 FCS, FCCS, 및 smFRET 교정 무료 이며 밝기 계산 하기 때문에 간단 하 고 농도 독립6에 비해 매우 매력적 이다. 그러나 그것은 중요 한 중요성의, 밝기 계산9;를 수행 하기 전에 표백 및 장기 강도 변동에 대 한 수정 표백에 따른 휘도 ?…
Disclosures
The authors have nothing to disclose.
Acknowledgements
이 작품 Wellcome 신망에 의해 지원 되었습니다 105278/Z/14/2 치료에 부여 Wellcome 신뢰 센터 인간 유전학에 의해 Wellcome 신뢰 코어 수상 203852/Z/16/2 자금입니다. 작업 씨에 스 그룹에 암 연구 영국 (C20724/A14414), 그리고 유럽 연합의 지평선 2020 연구 및 혁신 프로그램 그랜트 647278 유럽 연구 위원회에 의해 지원 됩니다.
Materials
| RosettaTM (DE3) pLysS cells | Novagen | 70956-3 | |
| Ampicillin | Sigma Aldrich | PubChem Substance ID 329824407 | |
| Chloramphenicol | Sigma Aldrich | PubChem Substance ID: 24892250 | |
| LB starter culture | QIAGEN | ||
| LB medium | QIAGEN | https://www.sigmaaldrich.com/content/dam/sigma-aldrich/head/search/external-link-icon.gif | |
| IPTG | Sigma Aldrich | PubChem Substance ID 329815691 | |
| IMAC buffer | Medicago | 09-1010-10 | |
| EDTA-free protease inhibitors | Sigma Aldrich | 11873580001 | |
| TALON resin | Clonetech | ||
| Nickel sepharose | GE Healthcare | ||
| S200 16/60 column | GE Healthcare | ||
| Glass bottom 8 well observation dish | Ibidi | 80827 |
References
- Voith von Voithenberg, L., Lamb, D. C. Single pair forster resonance energy transfer: A versatile tool to investigate protein conformational dynamics. BioEssays. 40, (2018).
- Padilla-Parra, S., Auduge, N., Coppey-Moisan, M., Tramier, M. Quantitative FRET analysis by fast acquisition time domain FLIM at high spatial resolution in living cells. Biophysical Journal. 95, 2976-2988 (2008).
- Padilla-Parra, S., Auduge, N., Coppey-Moisan, M., Tramier, M. Dual-color fluorescence lifetime correlation spectroscopy to quantify protein-protein interactions in live cell. Microscopy Research and Technique. 74, 788-793 (2011).
- Muller, J. D., Chen, Y., Gratton, E. Fluorescence correlation spectroscopy. Methods in Enzymology. 361, 69-92 (2003).
- Tramier, M., Coppey-Moisan, M. Fluorescence anisotropy imaging microscopy for homo-FRET in living cells. Methods in Cell Biology. 85, 395-414 (2008).
- Digman, M. A., Dalal, R., Horwitz, A. F., Gratton, E. Mapping the number of molecules and brightness in the laser scanning microscope. Biophysical Journal. 94, 2320-2332 (2008).
- Nolan, R., Iliopoulou, M., Alvarez, L., Padilla-Parra, S. Detecting protein aggregation and interaction in live cells: A guide to number and brightness. Methods. , (2017).
- Dalal, R. B., Digman, M. A., Horwitz, A. F., Vetri, V., Gratton, E. Determination of particle number and brightness using a laser scanning confocal microscope operating in the analog mode. Microscopy Research and Technique. 71, 69-81 (2008).
- Nolan, R., et al. nandb-number and brightness in R with a novel automatic detrending algorithm. Bioinformatics. , (2017).
- Hur, K. H., et al. Quantitative Measurement of Brightness from Living Cells in the Presence of Photodepletion. PLoS One. 9, (2014).
- Amara, J. F., et al. A versatile synthetic dimerizer for the regulation of protein-protein interactions. Proceedings of the National Academy of Sciences of the United States of America. 94, 10618-10623 (1997).
- Clackson, T., et al. Redesigning an FKBP-ligand interface to generate chemical dimerizers with novel specificity. Proceedings of the National Academy of Sciences of the United States of America. 95, 10437-10442 (1998).
- Rollins, C. T., et al. A ligand-reversible dimerization system for controlling protein-protein interactions. Proceedings of the National Academy of Sciences of the United States of America. 97, 7096-7101 (2000).
- Schindelin, J., Rueden, C. T., Hiner, M. C., Eliceiri, K. W. The ImageJ ecosystem: An open platform for biomedical image analysis. Molecular Reproduction and Development. 82, 518-529 (2015).
- . R: A Language and Environment for Statistical Computing. R Core. , (2017).
- . RStudio: Integrated Development Environment for R. R Team. , (2016).
- . nandb R package Available from: https://CRAN.R-project.org/package=nandb (2017)
- Jung, G., Wiehler, J., Zumbusch, A. The photophysics of green fluorescent protein: influence of the key amino acids at positions 65, 203, and 222. Biophysical Journal. 88, 1932-1947 (2005).
- Shaner, N. C., Steinbach, P. A., Tsien, R. Y. A guide to choosing fluorescent proteins. Nature Methods. 2, 905-909 (2005).
- Butkevich, A. N., et al. Hydroxylated fluorescent dyes for live-cell labeling: synthesis, spectra and super-resolution STED. Chemistry: A European Journal. 23, 12114-12119 (2017).

