Calibración-libre In Vitro cuantificación de proteína Homo-Oligomerización utilizando instrumentación comercial y Software de análisis de brillo libre de código abierto
Summary
Este protocolo describe un enfoque libre de calibración para cuantificación de proteína homo-Oligomerización en vitro basado en la espectroscopia de fluorescencia de fluctuación comercial luz microscopia. Se muestran los métodos de análisis y configuración correcta adquisición.
Abstract
Número y el brillo es una técnica de espectroscopia (FFS) de fluctuación libre de calibración de la fluorescencia para la detección de proteína homo-Oligomerización. Pueden emplearse utilizando un microscopio confocal convencional equipado con detectores digitales. Un protocolo para el uso de la técnica de vitro se muestra por medio de un caso de uso donde se observan número y brillo para cuantificar con precisión el estado oligomérico de FKBP12F36V etiqueta de mVenus antes y después de la adición de la droga dimerizing AP20187. Se discuten la importancia de utilizar los parámetros de adquisición microscopio correctos y los métodos de preprocesamiento y análisis de datos correctos. En particular, se destacó la importancia de la opción de corrección de fotoblanqueo. Este método de bajo costo puede ser empleada para estudiar las interacciones de la proteína-proteína en muchos contextos biológicos.
Introduction
Proteínas interacciones n Vitro
Tradicionalmente, la cristalografía y resonancia magnética nuclear experimentos combinados con microscopia del cryo-electrón (cryoEM) son las tecnologías elegidas para describir con precisión la arquitectura tridimensional de las proteínas y deducir su función por escudriñando sus detalles estructurales de alta resolución. Proteínas, sin embargo, no son estructuras estáticas y pueden experimentar una variedad de cambios conformacionales y vibraciones en el tiempo y el espacio. Por esta razón estructural información cristalográfica o CryoEM datos deben complementarse con otras técnicas (por ejemplo, simulaciones de dinámica molecular y técnicas de la sola molécula): la función de una proteína está relacionada con su conformacional cambios y las interacciones y esta información no está presente en una estructura estática. Con el fin de la punta de prueba dinámica intra moleculares, técnicas basadas en la sola molécula Forster transferencia de energía de resonancia (smFRET) son muy eficaz1. Estos enfoques son capaces de evaluar diversas subpoblaciones de moléculas en medios complejos. Esto es muy importante, estos cambios son rápidos y se producen durante la adquisición de los datos (es decir, nanosegundo a segundo rango).
Dos enfoques principales se emplean comúnmente para detectar y cuantificar estos cambios: las proteínas en solución y superficie-inmovilización. Para la detección de interacciones inter moleculares y en particular, el proceso de dimerización inducida por ligandos, smFRET no siempre es la mejor herramienta. De hecho, traste depende no sólo de la distancia (≈10 nm) sino también en la orientación de los dos dipolos (donador y aceptor, χ2) y la superposición de la emisión del donador con espectros de absorción2 del aceptador, pero tal vez esta última condición es menos importante siempre que el experimentador puede eligió el par derecho de traste. Una desventaja particular de smFRET para sondeo homo-dimerización proviene el etiquetado de la proteína de interés: para hetero smFRET, dimerización sólo puede ser detectado hasta un 50% (hetero-traste, es decir, sólo será capaz de detectar aceptador de donantes y donante receptor homo-dímeros no donante donante o de aceptador-aceptor, que es el otro 50% de los dímeros). El uso de la espectroscopia de correlación de fluorescencia (FCS) y derivados (FCCS, etc.3) para determinar proteína constantes de difusión y enlace constantes en vitro es otra alternativa. Estos enfoques no son capaces de cuantificar plenamente homo-dimerización o, como en el coeficiente de concentración y difusión y el radio y la difusión de medidas FCS uno de una partícula difusión dependen muy mal el peso molecular; por ejemplo un aumento de 10 veces el peso molecular sólo implica un cambio de 2.15 veces el coeficiente de difusión4. En el caso de dos colores FCS o FCCS, sólo el 50% de homo dímeros se verá por la misma razón que el anterior. Enfoques más prácticos y cuantitativos para detectar homo-dimerización en vitro e in vivo son homo-traste5 y el número y el brillo (N & B)6. Teniendo en cuenta el hecho de que homo-FRET requiere recuperación de instrumentación específica del valor de anisotropía (es decir, elementos/analizadores ópticos para recuperar la polarización paralela y perpendicular), N & B se presentaron aquí como una técnica favorable para detectar proteínas homo-dimerización y agregación. Puede ser empleado tanto en vitro como en vivo con un establecimiento comercial.
Número y brillo
N & B ha sido recientemente ha7. Esa revisión se centró en la aplicación de la técnica en células vivas. Vale la pena reproducir el formalismo matemático aquí como estas ecuaciones se aplicará a los datos recogidos in vitro. En primer lugar, es necesario definir algunos términos y cantidades matemáticas:
- Una entidad es un conjunto de moléculas que están limitados juntos.
- El ε del brillo de una entidad es el número de fotones emite por unidad de tiempo (por marco).
- n es el número de entidades presentes.
- De un determinado píxel a lo largo de una serie de imágenes, es su media intensidad y σ2 la variación en su intensidad.
Luego, con el conteo de fotones detectores y asumiendo las entidades móviles y sin fondo,
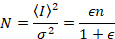
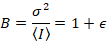
donde N es el número aparente y B es el brillo aparente. Esto se traduce en
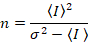
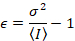
Dalal et al. 8 demostraron que con equipo análogo, uno necesita tres términos de corrección: el factor S, el fondo offsety el ruido de lectura σ02. Luego, otra vez asumiendo las entidades móviles,
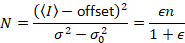
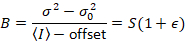
dando
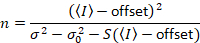
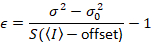
Tenga en cuenta que la ecuación antedicha para es diferente que en Dalal et al. 8 y una revisión posterior. 7 de Dalal et al. S en el denominador fue omitido debido a un error y este error se reprodujo en la revisión. La ecuación anterior es la correcta. Instrucciones para medir S, offset y σ02 – junto con una explicación de su significado – son dadas por Dalal et al. 8
El ε del brillo es proporcional al estado oligomérico de las entidades de difusión: ε será dos veces tan grande para dímeros como para monómeros, tres veces tan grandes para trimers como para monómeros, dos veces tan grandes para hexamers como para trimers y así sucesivamente. De esta manera, medir el ε de brillo, uno puede cuantificar cualquier tipo de multimerization.
Si hay una mezcla de los Estados presentes, número y el brillo no es capaz de recuperar los Estados presentes. Esto es una limitación de la técnica.
Detrend software algoritmo y nandb
La importancia de la corrección de fotoblanqueo ha subrayó previamente9. Fotoblanqueo se produce inevitablemente durante experimentos de microscopía de luz en el modo time-lapse; tanto en células vivas y en vitro. Muchos enfoques se han descrito en la literatura para corregir para el blanqueo de7. La técnica de filtrado exponencial con elección automática de robustos parámetro T es el mejor actual. Está integrado en el software libre de código abierto nandb9. De hecho, software que requiere el usuario seleccionar manualmente los parámetros robustos puede conducir a resultados incorrectos debido a la elección de este parámetro será arbitrario e incorrecto. El algoritmo automático inspecciona los datos y determina el parámetro, sin necesidad de intervención de usuario9. Incluso con la mejor opción del parámetro de suavizado, robustos tienen sus limitaciones y funciona bien sólo con porcentajes de fotoblanqueo inferiores al 25%, como se muestra con simulaciones9. Curiosamente, cuando se utiliza la rutina robustos automática, su exactitud es tal que uno puede trabajar con valores de brillo bajo (incluso B < 1.01) y por lo tanto, de baja intensidad y siendo preciso cuantificar homo-dimerización.
Fotoblanqueo también causa otro problema: la presencia de fluoróforos photobleached en un complejo de multimer. Esto hace que por ejemplo, un trímero aparecen como un dímero cuando una de las tres unidades en el trímero es no fluorescente. Hur y Mueller10 demostraron cómo corregir para esto y esta corrección también se destacó en un informe posterior7. El software nandb incluye corrección9.
La FKBP12 F36V sistema de
FKBP12F36V es una proteína que naturalmente no oligomerize pero se sabe que dimerizan con la adición del fármaco (conocido coloquialmente como el BB dimerizing ligando) AP2018711,12. Esto hace que sea un caso de prueba ideal para número y brillo: con FKBP12F36V, una duplicación de los Estado debe observarse sobre adición de BB.
Protocol
Representative Results
Discussion
N & B es una técnica para detectar multimerization luz comercial exploración confocal microscopio equipado con detectores digitales. Este enfoque es bastante atractivo en comparación con único punto FCS, FCCS y smFRET porque es libre de calibración y el cálculo de brillo es sencillo y concentración independiente6. Es de gran importancia, sin embargo, para corregir las fluctuaciones de intensidad de blanqueo y a largo plazo antes de realizar cálculos de brillo9; un l…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Este trabajo ha sido apoyado por Wellcome Trust conceder 105278/Z/14/2 al R.N. El Wellcome Trust Centre para genética humana está financiado por Wellcome Trust base Premio 203852/Z/16/2. Trabajo en el grupo C.S. es financiado por Cancer Research UK (C20724/A14414) y el Consejo Europeo de investigación horizonte 2020 investigación y innovación programa de beca 647278 de la Unión Europea.
Materials
| RosettaTM (DE3) pLysS cells | Novagen | 70956-3 | |
| Ampicillin | Sigma Aldrich | PubChem Substance ID 329824407 | |
| Chloramphenicol | Sigma Aldrich | PubChem Substance ID: 24892250 | |
| LB starter culture | QIAGEN | ||
| LB medium | QIAGEN | https://www.sigmaaldrich.com/content/dam/sigma-aldrich/head/search/external-link-icon.gif | |
| IPTG | Sigma Aldrich | PubChem Substance ID 329815691 | |
| IMAC buffer | Medicago | 09-1010-10 | |
| EDTA-free protease inhibitors | Sigma Aldrich | 11873580001 | |
| TALON resin | Clonetech | ||
| Nickel sepharose | GE Healthcare | ||
| S200 16/60 column | GE Healthcare | ||
| Glass bottom 8 well observation dish | Ibidi | 80827 |
References
- Voith von Voithenberg, L., Lamb, D. C. Single pair forster resonance energy transfer: A versatile tool to investigate protein conformational dynamics. BioEssays. 40, (2018).
- Padilla-Parra, S., Auduge, N., Coppey-Moisan, M., Tramier, M. Quantitative FRET analysis by fast acquisition time domain FLIM at high spatial resolution in living cells. Biophysical Journal. 95, 2976-2988 (2008).
- Padilla-Parra, S., Auduge, N., Coppey-Moisan, M., Tramier, M. Dual-color fluorescence lifetime correlation spectroscopy to quantify protein-protein interactions in live cell. Microscopy Research and Technique. 74, 788-793 (2011).
- Muller, J. D., Chen, Y., Gratton, E. Fluorescence correlation spectroscopy. Methods in Enzymology. 361, 69-92 (2003).
- Tramier, M., Coppey-Moisan, M. Fluorescence anisotropy imaging microscopy for homo-FRET in living cells. Methods in Cell Biology. 85, 395-414 (2008).
- Digman, M. A., Dalal, R., Horwitz, A. F., Gratton, E. Mapping the number of molecules and brightness in the laser scanning microscope. Biophysical Journal. 94, 2320-2332 (2008).
- Nolan, R., Iliopoulou, M., Alvarez, L., Padilla-Parra, S. Detecting protein aggregation and interaction in live cells: A guide to number and brightness. Methods. , (2017).
- Dalal, R. B., Digman, M. A., Horwitz, A. F., Vetri, V., Gratton, E. Determination of particle number and brightness using a laser scanning confocal microscope operating in the analog mode. Microscopy Research and Technique. 71, 69-81 (2008).
- Nolan, R., et al. nandb-number and brightness in R with a novel automatic detrending algorithm. Bioinformatics. , (2017).
- Hur, K. H., et al. Quantitative Measurement of Brightness from Living Cells in the Presence of Photodepletion. PLoS One. 9, (2014).
- Amara, J. F., et al. A versatile synthetic dimerizer for the regulation of protein-protein interactions. Proceedings of the National Academy of Sciences of the United States of America. 94, 10618-10623 (1997).
- Clackson, T., et al. Redesigning an FKBP-ligand interface to generate chemical dimerizers with novel specificity. Proceedings of the National Academy of Sciences of the United States of America. 95, 10437-10442 (1998).
- Rollins, C. T., et al. A ligand-reversible dimerization system for controlling protein-protein interactions. Proceedings of the National Academy of Sciences of the United States of America. 97, 7096-7101 (2000).
- Schindelin, J., Rueden, C. T., Hiner, M. C., Eliceiri, K. W. The ImageJ ecosystem: An open platform for biomedical image analysis. Molecular Reproduction and Development. 82, 518-529 (2015).
- . R: A Language and Environment for Statistical Computing. R Core. , (2017).
- . RStudio: Integrated Development Environment for R. R Team. , (2016).
- . nandb R package Available from: https://CRAN.R-project.org/package=nandb (2017)
- Jung, G., Wiehler, J., Zumbusch, A. The photophysics of green fluorescent protein: influence of the key amino acids at positions 65, 203, and 222. Biophysical Journal. 88, 1932-1947 (2005).
- Shaner, N. C., Steinbach, P. A., Tsien, R. Y. A guide to choosing fluorescent proteins. Nature Methods. 2, 905-909 (2005).
- Butkevich, A. N., et al. Hydroxylated fluorescent dyes for live-cell labeling: synthesis, spectra and super-resolution STED. Chemistry: A European Journal. 23, 12114-12119 (2017).

