Kalibrering-fri In Vitro kvantificering af Protein Homo-oligomerisering ved hjælp af kommercielle instrumentering og gratis, Open Source lysstyrke analyse Software
Summary
Denne protokol beskriver en kalibrering-fri tilgang til kvantificering af protein homo-oligomerisering in vitro- baseret på fluorescens udsving spektroskopi ved hjælp af kommercielle lys scanning mikroskopi. Den korrekte anskaffelse indstillinger og analysemetoder, der er vist.
Abstract
Antallet og lysstyrke er en kalibrering-fri fluorescens udsving spektroskopi (FFS) teknik til påvisning af protein homo-oligomerisering. Det kan være ansat, ved hjælp af en konventionel Konfokal mikroskop udstyret med digitale detektorer. En protokol til brug af teknik i vitro er vist ved hjælp af en use case, hvor antallet og lysstyrke kan ses at nøjagtigt kvantificere mVenus-mærket FKBP12F36V oligomere tilstand før og efter tilsætning af det dimerizing stof AP20187. Betydningen af at anvende korrekte mikroskop erhvervelse parametre og de korrekte data forbehandling og analysemetoder, der er diskuteret. Navnlig understreges betydningen af valget af photobleaching korrektion. Denne billig metode kan anvendes til at undersøge protein-protein interaktioner i mange biologiske sammenhænge.
Introduction
Protein-protein interaktioner, jeg n in Vitro
Traditionelt er krystallografi og Kernemagnetisk resonans eksperimenter kombineret med kryo-elektronmikroskopi (cryoEM) de teknologier, der er valgt til præcist beskriver den tre-dimensionelle arkitektur af proteiner og at udlede deres funktion af granske deres strukturelle detaljer i høj opløsning. Proteiner, men er ikke statiske strukturer og kan gennemgå en række konformationelle ændringer og vibrationer i tid og rum. Dette er grunden til strukturelle oplysninger fra krystallografiske eller CryoEM data skal suppleres med andre teknikker (fx molecular dynamics simulationer og enkelt molekyle teknikker): funktionen af et protein, der er relateret til dets konformationelle ændringer og interaktioner, og disse oplysninger er ikke til stede i en statisk struktur. For at sende forespørgsler om intra molecular dynamics, er teknikker baseret på enkelt molekyle Forster resonans energi Transfer (smFRET) meget effektiv1. Disse metoder er i stand til at vurdere forskellige delpopulationer af molekyler i komplekse medier. Dette er meget vigtigt, da disse ændringer er hurtig og opstå under erhvervelse af data (dvs. nanosekund til andet område).
To vigtigste tilgange er almindeligvis ansat til at detektere og kvantificere disse ændringer: proteiner i løsning og overflade-immobilisering. Til påvisning af indbyrdes molekylære interaktioner og navnlig processen af dimerization induceret af ligander, er smFRET ikke altid det bedste værktøj. Faktisk, FRET afhænger ikke kun af afstanden (≈10 nm), men også på orientering af de to dipoler (donor og acceptor, χ2), og overlapningen af donor emission med de acceptor absorptionsspektre2, men måske denne sidste betingelse er mindre forudsat at experimentalist kan valgte vigtigt, den rigtige FRET par. En særlig ulempe ved smFRET for sondering homo-dimerization kommer fra mærkning af protein af interesse: for hetero smFRET, dimerization kan kun blive registreret op til 50% (dvs., hetero-FRET vil kun kunne opdage donor-acceptor og acceptor-donor homo-dimerer men ikke donor-donor eller acceptor-acceptor, som de andre 50% af dimerer). Brugen af fluorescens-korrelations-spektroskopi (FCS) og derivater (FCCS, etc.3) at undersøge protein diffusion konstanter og bindende konstanter i vitro er et andet alternativ. Disse tilgange er ikke i stand til fuldt kvantificere homo-dimerization enten, som i FCS en foranstaltninger koncentration og diffusion, og radius og diffusion koefficient af et spreder partikel er meget dårligt afhængige molekylvægt; for eksempel vil en 10-fold stigning i molekylvægt kun medføre en ændring af 2,15 fold i diffusion koefficient4. To-farvet FCS eller FCCS, vil kun 50% af homo-dimerer ses af samme grund som ovenfor. Den mest praktiske og kvantitative tilgange til at registrere homo-dimerization i in vitro og i vivo er homo-FRET5 og antallet og lysstyrke (N & B)6. Betragtning af at homo-FRET kræver særlige instrumentering inddrivelse af anisotropy værdi (dvs. optiske elementer/analysatorer til at gendanne den parallelle med og vinkelrette polarisering), N & B præsenteres her som en gunstig teknik til at opdage protein homo-dimerization og sammenlægning. Det kan være ansat både in vitro- og i vivo med en kommerciel set-up.
Antallet og lysstyrke
N & B har været for nylig gennemgik7. Denne revision fokuseret på anvendelsen af teknikken i levende celler. Det er værd at gengive den matematiske formalisme her som disse ligninger vil blive anvendt til data indsamlet in vitro. For det første er det nødvendigt at fastlægge nogle begreber og matematiske mængder:
- En enhed er et sæt af molekyler, der bindes sammen.
- Lysstyrke ε af en enhed, er antallet af fotoner det udsender pr. tidsenhed (per frame).
- n er antallet af enheder, der er til stede.
- For en given pixel i løbet af et billede-serien er dets gennemsnitlige intensitet og σ2 er variansen i dens intensitet.
Derefter, med photon-optælling detektorer og under forudsætning af mobile enheder og ingen baggrund,
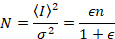
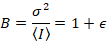
hvor N er tilsyneladende nummer og B er tilsyneladende lysstyrke. Dette resulterer i
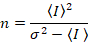
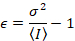
Anne Juul et al. 8 viste, at med analogt udstyr, man behøver tre korrektion vilkår: S faktor, baggrund forskydningog udlæsning støj σ02. Så igen under forudsætning af mobile enheder,
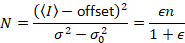
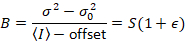
at give
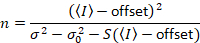
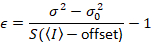
Bemærk, at ovenstående ligning for forskellige, givet i Anne Juul et al. 8 og en efterfølgende revision. 7 i Anne Juul et al. S i nævneren blev udeladt på grund af et slåfejl og denne fejl blev gengivet i forbindelse med revisionen. Ligningen ovenfor er den rigtige. Instruktioner for måling af S, forskydning og σ02 – sammen med en forklaring af deres betydning – er givet ved Anne Juul et al. 8
Lysstyrke ε er proportional med de sprede enheder oligomere tilstand: ε vil blive dobbelt så stor for dimerer som for monomerer, tre gange så stor for trimere som for monomerer, to gange så stor for hexamers, som det er for trimere og så videre. På denne måde, måling af lysstyrke ε, kan en kvantificering af enhver form for multimerization.
Hvis der er en blanding af oligomere stater nuværende, er antallet og lysstyrke ikke i stand til at inddrive de oligomere enkeltstater stede. Dette er en begrænsning af teknikken.
Detrend algoritme og nandb software
Betydningen af at korrigere for photobleaching har været understreget tidligere9. Photobleaching uundgåeligt opstår under lysmikroskopi eksperimenter i time-lapse tilstand; både i levende celler og in vitro. Mange tilgange er blevet beskrevet i litteraturen til at korrigere for blegning7. Den eksponentielle filtrering teknik med automatisk valg af detrending parameter T er den nuværende bedste. Det er integreret i gratis, open source software nandb9. Faktisk, software, der kræver, at brugeren kan manuelt vælge deres detrending parameter kan føre til forkerte resultater, fordi denne parameter valg vil sandsynligvis blive vilkårligt og forkert. Den automatiske algoritme undersøger data og bestemmer parameteren passende for det, uden behov for brugeren intervention9. Selv med den bedste valg af udjævning parameter, detrending har sine begrænsninger og fungerer godt kun med photobleaching procent lavere end 25%, som vist med simuleringer9. Interessant, når du bruger den automatiske detrending rutine, dets nøjagtighed er sådan, at man kan arbejde med lav lysstyrke værdier (selv B < 1,01), og dermed lav intensitet, og stadig være tilstrækkeligt præcise til at kvantificere homo-dimerization.
Photobleaching medfører også et andet problem: tilstedeværelsen af photobleached fluorophores i en kompleks multimer. Dette gør fx en trimer vises som en dimer, når en af de tre enheder i trimer er ikke-fluorescerende. Hur og Mueller10 viste hvordan man kan korrigere for dette, og denne korrektion blev også understreget i en efterfølgende anmeldelse7. Nandb software indeholder denne rettelse9.
Af FKBP12 F36V system
FKBP12F36V er et protein, som naturligvis ikke oligomerize men er kendt for at dimerize ved tilsætning af AP20187 stof (i daglig tale kendt som BB dimerizing ligand)11,12. Dette gør det en ideel prøvesag for antallet og lysstyrke: med mærket FKBP12F36V, en fordobling af oligomere stat bør overholdes ved tilsætning af BB.
Protocol
Representative Results
Discussion
N & B er en teknik til at opdage multimerization ved hjælp af kommercielle lys scanning Konfokal mikroskoper udstyret med digitale detektorer. Denne fremgangsmåde er ganske attraktiv i forhold til enkelt punkt FCS, FCCS og smFRET fordi det er kalibrering gratis og beregningen af lysstyrke er ligetil og koncentration uafhængige6. Det er af stor betydning, men at korrigere for blegning og langvarig intensitet udsving før du udfører lysstyrke beregninger9; en svag stignin…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Dette arbejde er blevet støttet af Wellcome Trust give R.N. 105278/Z/14/2 Wellcome Trust Center for humangenetik er finansieret af Wellcome Trust Core Award 203852/Z/16/2. Arbejde i gruppen C.S. understøttes af Cancer Research UK (C20724/A14414) og det Europæiske Forskningsråd under EUs Horisont 2020 forskning og Innovation program tilskud 647278.
Materials
| RosettaTM (DE3) pLysS cells | Novagen | 70956-3 | |
| Ampicillin | Sigma Aldrich | PubChem Substance ID 329824407 | |
| Chloramphenicol | Sigma Aldrich | PubChem Substance ID: 24892250 | |
| LB starter culture | QIAGEN | ||
| LB medium | QIAGEN | https://www.sigmaaldrich.com/content/dam/sigma-aldrich/head/search/external-link-icon.gif | |
| IPTG | Sigma Aldrich | PubChem Substance ID 329815691 | |
| IMAC buffer | Medicago | 09-1010-10 | |
| EDTA-free protease inhibitors | Sigma Aldrich | 11873580001 | |
| TALON resin | Clonetech | ||
| Nickel sepharose | GE Healthcare | ||
| S200 16/60 column | GE Healthcare | ||
| Glass bottom 8 well observation dish | Ibidi | 80827 |
References
- Voith von Voithenberg, L., Lamb, D. C. Single pair forster resonance energy transfer: A versatile tool to investigate protein conformational dynamics. BioEssays. 40, (2018).
- Padilla-Parra, S., Auduge, N., Coppey-Moisan, M., Tramier, M. Quantitative FRET analysis by fast acquisition time domain FLIM at high spatial resolution in living cells. Biophysical Journal. 95, 2976-2988 (2008).
- Padilla-Parra, S., Auduge, N., Coppey-Moisan, M., Tramier, M. Dual-color fluorescence lifetime correlation spectroscopy to quantify protein-protein interactions in live cell. Microscopy Research and Technique. 74, 788-793 (2011).
- Muller, J. D., Chen, Y., Gratton, E. Fluorescence correlation spectroscopy. Methods in Enzymology. 361, 69-92 (2003).
- Tramier, M., Coppey-Moisan, M. Fluorescence anisotropy imaging microscopy for homo-FRET in living cells. Methods in Cell Biology. 85, 395-414 (2008).
- Digman, M. A., Dalal, R., Horwitz, A. F., Gratton, E. Mapping the number of molecules and brightness in the laser scanning microscope. Biophysical Journal. 94, 2320-2332 (2008).
- Nolan, R., Iliopoulou, M., Alvarez, L., Padilla-Parra, S. Detecting protein aggregation and interaction in live cells: A guide to number and brightness. Methods. , (2017).
- Dalal, R. B., Digman, M. A., Horwitz, A. F., Vetri, V., Gratton, E. Determination of particle number and brightness using a laser scanning confocal microscope operating in the analog mode. Microscopy Research and Technique. 71, 69-81 (2008).
- Nolan, R., et al. nandb-number and brightness in R with a novel automatic detrending algorithm. Bioinformatics. , (2017).
- Hur, K. H., et al. Quantitative Measurement of Brightness from Living Cells in the Presence of Photodepletion. PLoS One. 9, (2014).
- Amara, J. F., et al. A versatile synthetic dimerizer for the regulation of protein-protein interactions. Proceedings of the National Academy of Sciences of the United States of America. 94, 10618-10623 (1997).
- Clackson, T., et al. Redesigning an FKBP-ligand interface to generate chemical dimerizers with novel specificity. Proceedings of the National Academy of Sciences of the United States of America. 95, 10437-10442 (1998).
- Rollins, C. T., et al. A ligand-reversible dimerization system for controlling protein-protein interactions. Proceedings of the National Academy of Sciences of the United States of America. 97, 7096-7101 (2000).
- Schindelin, J., Rueden, C. T., Hiner, M. C., Eliceiri, K. W. The ImageJ ecosystem: An open platform for biomedical image analysis. Molecular Reproduction and Development. 82, 518-529 (2015).
- . R: A Language and Environment for Statistical Computing. R Core. , (2017).
- . RStudio: Integrated Development Environment for R. R Team. , (2016).
- . nandb R package Available from: https://CRAN.R-project.org/package=nandb (2017)
- Jung, G., Wiehler, J., Zumbusch, A. The photophysics of green fluorescent protein: influence of the key amino acids at positions 65, 203, and 222. Biophysical Journal. 88, 1932-1947 (2005).
- Shaner, N. C., Steinbach, P. A., Tsien, R. Y. A guide to choosing fluorescent proteins. Nature Methods. 2, 905-909 (2005).
- Butkevich, A. N., et al. Hydroxylated fluorescent dyes for live-cell labeling: synthesis, spectra and super-resolution STED. Chemistry: A European Journal. 23, 12114-12119 (2017).

