면역형광에 의한 DNA 수리 단백질 상호작용의 시각화
Summary
DNA 손상에 따라 인간 세포는 게놈의 무결성을 회복하기 위해 필수적인 수리 경로를 활성화합니다. 여기서, 우리는 DNA 복구 단백질을 검출하고, 그들의 공간 및 측두모집을 분석하고, DNA 손상의 사이트에서 단백질 단백질 상호 작용을 심문하는 것을 돕는 수단으로 간접적인 면역 형광의 방법을 기술합니다.
Abstract
포유류 세포는 화학 물질, 방사선 및 자연적으로 발생하는 신진 대사 부산물, DNA 모욕의 특정 유형을 생성합니다. 독독성 제는 DNA 백본을 손상시키거나, 부러뜨리거나, 개별 기지의 화학적 특성을 수정할 수 있습니다. DNA 모욕에 따라 DNA 손상 반응(DDR) 경로가 활성화되고 수리에 관련된 단백질이 채용됩니다. 손상 유형을 감지하고 적절한 수리 응답을 활성화하는 데 많은 요인이 관여합니다. DDR 요인을 올바르게 활성화하고 모집하지 못하면 유전체 불안정으로 이어질 수 있으며 암을 포함한 많은 인간의 병리학에 기초가 됩니다. DDR 단백질의 연구는 약물 내성의 물질독성 약물 반응 과 세포 메커니즘에 대한 통찰력을 제공 할 수 있습니다.
생체 내에서단백질을 시각화하는 두 가지 주요 방법이 있습니다 : 직접 관찰, 형광 단백질로 관심있는 단백질을 태그하고 라이브 이미징에 의해 다음, 또는 고정 된 샘플에 간접 적인 면역 형광. 형광 태그 단백질의 시각화는 시간이 지남에 따라 정밀한 모니터링을 허용하지만, N-또는 C 종면에서 직접 태그는 단백질 국소화 또는 기능을 방해할 수 있습니다. 수정되지 않은 내생성 버전에서 단백질을 관찰하는 것이 바람직합니다. DNA 복구 단백질이 DNA 모욕에 모집될 때, 그들의 농도는 현지에서 증가하고 특정 항체를 사용하여 간접적인 면역 형광에 의해 가시화될 수 있는 그룹, 또는 “foci”를 형성합니다.
단백질 foci의 검출은 직접적인 상호 작용의 확실한 증거를 제공하지 않지만, 세포에 있는 단백질의 공동 현지화는 손상의 사이트에 다시 그룹화하고 복잡한 형성을 위해 요구되는 사건의 순서를 알릴 수 있다는 것을 나타냅니다. 단백질의 야생 모형 또는 돌연변이 버전을 표현하는 세포에 있는 foci 공간 중첩의 주의깊은 분석은 DNA 복구 기능에 중요한 기능적인 도메인에 귀중한 단서를 제공할 수 있습니다. 마지막으로, 단백질의 공동 국소화는 세포의 공동 면역 침전성 또는 정제 된 단백질을 사용하여 직접 풀다운하여 검증 될 수있는 가능한 직접적인 상호 작용을 나타냅니다.
Introduction
인간 세포는 다양한 기원의 다양한 DNA 손상 제에 지속적으로 노출됩니다. 외인성 근원은 주로 방사선, 화학물질 (화학요법 에이전트 및 몇몇 항생제를 포함하여), 바이러스에 노출로 이루어져 있고, 주요 내인성 근원은 DNA 복제 및 산화 긴장에 있는 오류를 포함하는 동안. 물질독성 노출의 직접적인 영향은 응력과 노출 량에 따라 변형된 베이스에서 잠재적으로 치명적인 DNA 이중 가닥 브레이크(DSB)까지 다양할 수 있습니다. 궁극적으로, 수리되지 않거나 잘못 수리된 DNA 손상은 돌연변이, 게놈 재배치, 게놈 불안정의 축적으로 이어질 수 있고 결국 발암발생 1으로이어질 수 있다. 포유류 세포는 DNA손상2,,3의 특정 유형을 인식하고 세포 주기 진행과 동기화된 적시 방식으로 복구하기 위해 복잡한 경로를 진화시켰다.
이온화 방사선 (IR)은 DNA 이중 나선을 손상시키고 DNA 손상의 가장 해로운 형태 중 하나 인 이중 가닥 휴식 (DSBs)을 만듭니다. MRN(MRE11, RAD50, NBS1) 복합기능은 DNA의 센서로서 기능하며 단백질 키나제 실조(ATM)4,,5를활성화한다. DNA 에 의해 ATM의 초기 활성화에 따라, ATM은 휴식의 사이트에서 DDR 이벤트의 폭포를 트리거, 키 이벤트, 히스톤 변형 H2AX6의인산화를 시작. 잔류물 S139에 H2AX 인산화는 dna 병변,,6,7,8,9주위에 메가베이스까지 영역까지 에 걸쳐, γH2AX로 활성화한다.8 이 이벤트는 DNA 접근성을 증가시켜 다른 DNA 수리 단백질7의모집 및 축적으로 이어진다. γH2AX는 주변 DSB를 풍부하고 구체적으로 유도하기 때문에 특정 항체를 사용하여 쉽게 시각화할 수 있으며 DNA 수리 분야에서 DSBs에 대한 대리 마커로 일반적으로 사용됩니다. 휴식이 신호되면 세포는 DNA 복구 경로를 활성화하고 DNA 손상을 처리합니다. 단백질 MDC1(DNA 손상 검사점 단백질 1의 중재자)은 γH2AX10을직접 결합하고 ATM11과 상호 작용하며 NBS112,,13과도상호 작용한다. DSB에서 MRN 단지의 농도를 높이고 양성 ATM 피드백 루프를 시동하는 데 기여합니다. γH2AX는 휴식이 수리되면 빠르게 제거되므로 DSB 클리어런스를 모니터링할 수 있습니다. 현미경 검사법에 이어, 시간이 지남에 따라 γH2AX의 감소는 잔류 휴식 및 DNA 수리 효율의 간접적인 측정을 제공합니다.
진핵 세포는 여러 경로에 의해 DSB를 복구 할 수 있으며, 두 가지 주요 것들은 비 동상화 종조 (NHEJ) 및 상동성 재조합 (HR)(도1)입니다. NHEJ는 본질적으로 확장 된 상동성의 사용없이 DNA 이중 가닥 끝을 ligates 및 세포 주기 에 걸쳐 작동14,,15. HR은 S 및 G2 단계에서 우세하게 되고, 그렇지 않으면 억압된다, 그것은수리를위한상동성 템플릿으로 자매 크로마티드를 필요로하기 때문에14,16. NHEJ와 HR 사이의 통로 선택은 자매 크로마티드의 물리적 근접성뿐만 아니라 NHEJ를 억제하는 DNA 끝 절제술17의연장에 달려 있습니다.
편집 의존DSB 복구는 휴식 끝에서 5’가닥의 뉴클레오분해성 분해에 의해 시작되어 3’단일 가닥 DNA(ssDNA) 꼬리를 생성하며, 이는 5′-3’절제라고 하는 과정이다. MRN 복합체는 DNA 엔드 절제술을 개시하고 추가 절제술은 BLM/EXO1(블룸 증후군 단백질/엑소뉴클레아제 1) 또는 BLM/DNA2(DNA 복제 ATP 의존헬리케이스/뉴클레아제)18,,19,,20,21,22와함께 처리된다.,, DNA 최종 절제술은 MRN복합체(23)와의 직접적인 상호작용및 BRCA1(유방암형 1형 감수성 단백질),24,25의모집을 통해 CtIP(CtBP-상호작용 단백질)에 의해 강화된다. 복제 단백질 A(RPA)는 즉시 ssDNA에 노출되어 재조합 단백질 RAD51에 의해 변위되어 상동성 검색 및 가닥침입(26,,27,,28)을촉매화하는 뉴클레오단백질 필라멘트를 형성한다.
절제술의 개시는 수리 통로 선택을 위한 중요한 단계입니다. 일단 절제술이 시작되면, DNA 끝은 Ku70/Ku80 이종수(NHEJ 통로의 성분)에 의해 결합을 위한 가난한 기판이 되고 세포는 HR17,,29,,30에투입된다. Ku70/Ku80 이성애는 DSB에 결합하여 DNA-PKcs 및 p53 결합 단백질 1(53BP1)29,,30을모집한다. 53BP1은 G1에서 절제하는 장벽역할을 하여 NHEJ31,,32를홍보하면서 HR을 차단하지만, S 단계에서 BRCA1 의존적 방식으로 제거되어,33,34의절제술이 발생할 수 있다. 따라서 53BP1과 BRCA1은 DSB 수리에서 반대 역할을 하며, 53BP1은 NHEJ 촉진제이며 BRCA1은 HR을 통해 파손을 복구할 수 있습니다.
실험실에서, DSB 형성은 이온화 방사선(IR)에 의해 유도될 수 있다. 이 예는 4 Gy의 고용량을 활용하지만, 1 Gy 및 2 Gy는 또한 풍부한 단백질에 의한 포시 형성의 분석에 적합한 상당한 양의 DSB를 생성한다. 사용되는 방사선의 종류와 투여량은 DNA와 세포에서 다른 병변으로 이어질 수 있다는 점에 유의하는 것이 중요합니다: IR은 DSB를 유도하는 동안, 또한 단일 가닥 파손 또는 염기 수정을 일으킬 수 있습니다 (조사 선형 에너지 전송에 대한참조35,,36 및 DNA 손상의 유형). 이온화 방사선 유도 포시(IRIF) 형성 및 이들의 클리어런스의 운동을 결정하기 위해, 이는 활성DDR8,9,,37,,38의손상 및 반전의 수리를 나타내며, 이온화 후 다른 시점에서 포시 형성을 모니터링할 수 있다., 모든 주요 DNA 손상 단백질의 활성화 및 클리어런스의 타이밍은39로알려져 있으며, 많은 사람들이 주요 사건의 대리 마커로 조사된다. 예를 들어, ssDNA에 대한 높은 친화력을 지닌 pRPA는 휴식 절제의 대리로서 사용되며, MRN 단백질(MRE11, RAD50, NBS1) 및 엑소뉴클레아제도 절제술 효율을 평가하는데 사용될 수 있다. RAD51, BRCA1, BRCA2(유방암 형 2형 감수성 단백질) 및 PALB2(BRCA2의 파트너 및 국소화)가 HR 효율을 평가하기 위해 모니터링될 수 있지만, 쿠 단백질 또는 53BP1의 존재는 NHEJ(도1)의마커로 사용된다.
DNA 수리 기계의 단백질이 서로 를 모집하여 슈퍼 복합체에서 서로 모집하고 조립함에 따라, DNA 단백질 및 단백질 단백질 상호 작용은 시간이 지남에 따라 개별 적인 국소화를 따르고 단백질의 공동 국소화를 분석함으로써 추론 될 수 있으며, 세포40,,41,,42에서신호가 겹쳐서 시각화됩니다. 세포주에서, 게놈 편집을 통해 또는 플라스미드 기반 돌연변이의 과발현에 의해 특정 DNA 복구 유전자에 포인트 돌연변이 또는 삭제의 도입은 DNA 손상(예를 들어, γH2AX를 가진 공동 국소화) 또는 복잡한 조립(예를 들어, 다른 단백질과 공동 국소화) 또는 복잡한 조립(다른 단백질과 공동 국소화) DNA 에 대한 영향을 받아 특정 잔류물 및 가능한 역할에 대한 조사를 가능하게 합니다. 여기서, 우리는 시간이 지남에 따라 γH2AX foci를 따라 DSB의 형성 및 해상도를 조사하기 위한 의미로 간접 면역 형광을 사용합니다. 또한 DSB 수리의 주요 플레이어에 의한 포시 형성 및 공동 국소화 분석의 한 가지 예를 제시합니다: p53 결합 단백질 1 (53BP1)32. 앞에서 언급했듯이, 53BP1은 DNA 수리 경로 선택의 중심으로 간주됩니다. 53BP1 축적 및 γH2AX와의 공동 국소화에 따라 세포 주기 단계, DNA 손상 축적 및 DSB를 복구하는 데 사용되는 경로에 대한 귀중한 정보를 제공합니다. 간접 면역국화의 목적은 세포주에서 DNA 손상 복구의 효율을 평가하는 것입니다, 이 연구에서와 같은 IR에 이어, 또는 세포의 다양한 스트레스에 노출 후, DNA 교차링크에서 복제 포크의 막힘에 이르기까지(DNA 손상 제의 목록은 표 1에제공된다).
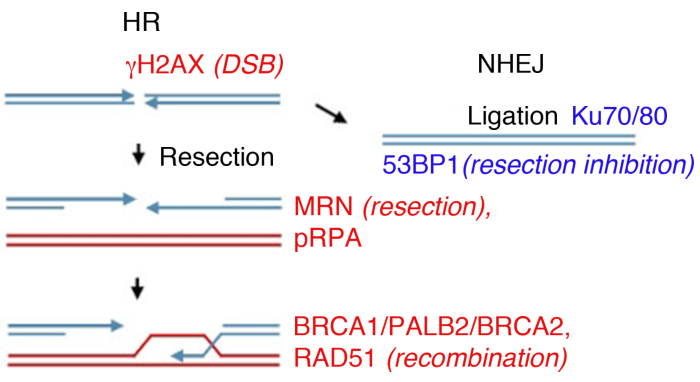
그림 1: DNA 이중 가닥 브레이크(DSB) 수리 경로.
DSB 수리에는 두 가지 주요 경로가 포함됩니다: 동종 재조합(HR, 왼쪽) 및 비동호동성 최종 결합(NHEJ, 오른쪽). 휴식 후, 단백질은 휴식(γH2AX)을 표시하고, 최종 절제술(MRN)에 참여하고, 절제된 ssDNA(pRPA)를 코팅하고, 재조합(BRCA1, PALB2, BRCA2, RAD51) 또는 제한 절제술을 촉진하고 NHEJ(53BP1)를 촉진한다. 그밖 단백질은 손상 복구에 참여합니다, 그러나 열거된 단백질은 간접적인 면역 형광에 선행됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
| DNA 손상 에이전트 | 행동 메커니즘 | 권장 용량 |
| γ 선/엑스레이 | 방사선 일부 통제되지 않는 세포 효과와 이중 좌초 휴식의 형성 |
1-4 Gy |
| 36 아르 이온 | 방사선 이중 좌초 휴식의 형성 |
270 케브/μm |
| α 입자 | 방사선 이중 좌초 휴식의 형성 |
116 케브/μm |
| 블레오마이신 | DNA 합성 억제제 | 0.4-2 μg/mL |
| 캄프토테신 | 포포아소아제I의 억제제 I | 10-200 nM |
| 시스플라틴 | 알키라팅 에이전트 (인트라스트랜드 크로스링크 유도) |
0.25-2 μM |
| 독소루비신 | 상호 작용 에이전트 토포이소머라제 II의 억제제 |
10-200 nM |
| 에토포사이드 | 토포이소머라제 II의 억제제 | 10 μM |
| 하이드록수레아 | DNA 합성 억제제 (리보뉴클레오티드 환원효소) |
10-200 μM |
| 메틸 메탄술포네이트 | 알키라팅 에이전트 | 0.25-2 mM |
| 미토마이신 C | 알키라팅 에이전트 | 0.25-2 μM |
| 자외선(UV) | 티미딘 디머 의 형성 (DNA 사슬의 왜곡 생성) |
50-100 mJ/cm2 |
표 1: 제독독성 제고품. DNA 손상 에이전트의 예, 행동의 그들의 기계장치 및 제안된 작업 농도에 근거를 둔 유도된 손상.
Protocol
Representative Results
Discussion
현미경 검사법에 의한 DNA 손상 수리의 타이밍 그리고 효율의 분석은 DNA 복구 기계가 일반적인 세포 및 암과 같은 인간 병리에서 어떻게 작동하는지 이해하는 데 필수적임이 입증되었습니다.
인계판에서 활성단백질을 검출할 수 있는 특정 항체의 개발(예: γH2AX, pRPA, pRAD50 등10,,23,,39,,43…
Disclosures
The authors have nothing to disclose.
Acknowledgements
이 작품은 샌안토니오 지역 재단의 보조금에 의해 지원되었다. 메이스 암 센터는 NCI 암 센터 지원 코어 보조금 P30 CA054174에 의해 지원됩니다. 시약을 조달하는 데 도움을 준 스티븐 할로웨이와 우주 및 현미경 용량 제공에 대한 성 연구소에 감사드립니다.
Materials
| 16% (v/v) paraformaldehyde (PFA) aqueous solution | Electron Microscopy Sciences | 15710 | Microscopy quality of the PFA ensures best images. If using "home-made PFA", filter prior to use. |
| Bovine serum albumin (BSA) | Sigma-Aldrich | A3059 | Heat-shock fraction is recommended, to avoid precipitation/background. |
| Coverglass #1, 18 mm round (coverslips) | Neuvitro | NC0308920 | Coverslips need to be cleaned and sterilized prior using, with HCl or ethanol. |
| DMEM, High Glucose [(+) 4.5 g/L D-Glucose, (+) L-Glutamine, (-) Sodium Pyruvate] | Gibco | 11965118 | Adjust the growing media to the needs of cell line used. |
| DPBS, no calcium, no magnesium | Gibco | 14190144 | PBS for tissue culture. |
| Ethylene glycol-bis(β-aminoethyl ether)-N,N,N′,N′-tetraacetic acid (EGTA) | Research Products International | E57060 | Nuclear extraction buffer. |
| Fetal Bovine Serum (FBS) | Life Technologies | 104370028 | The quality of FBS can be assessed by testing gH2AX foci formation. If traces of genotoxic endotoxin are present in the batch, gH2AX will be positive in the absence of stress. |
| Magnesium chloride (MgCl2) | Research Products International | M24000 | Nuclear extraction buffer. |
| Piperazine-N,N′-bis(2-ethanesulfonic acid) (PIPES) | Research Products International | P40150 | Nuclear extraction buffer. |
| SlowFade Diamond Antifade Mountant with DAPI | Invitrogen | S36973 | 300 nM DAPI with VECTASHIELD Antifade Mounting Medium can be used instead. |
| Sodium chloride (NaCl) | Research Products International | S23020 | Nuclear extraction buffer. |
| Sucrose | Research Products International | S24060 | Nuclear extraction buffer. |
| Superfrost Plus Microscope Slides | Fisherbrand | 1255015 | Polysine Slides can be used instead. |
| TC-Treated Multiple Well Plates, size 12 wells | Costar | 3513 | Seeding on coverslips is done in 12-wells plate. |
| Triton X-100 | AmericanBio | AB02025 | Nuclear extraction buffer. |
| TrypLE Express Enzyme (1X), No Phenol Red | Gibco | 12604021 | Trypsin-EDTA can be used instead. |
| Trypsin-EDTA (0.5%), No Phenol Red | Gibco | 15400054 | TrypLE can be used instead. |
References
- Prakash, R., Zhang, Y., Feng, W., Jasin, M. Homologous recombination and human health: the roles of BRCA1, BRCA2, and associated proteins. Cold Spring Harbor Perspectives in Biology. 7 (4), 016600 (2015).
- Jalan, M., Olsen, K. S., Powell, S. N. Emerging Roles of RAD52 in Genome Maintenance. Cancers (Basel). 11 (7), (2019).
- Oh, J., Symington, L. S. Role of the Mre11 Complex in Preserving Genome Integrity. Genes (Basel). 9 (12), (2018).
- Uziel, T., et al. Requirement of the MRN complex for ATM activation by DNA damage. The EMBO Journal. 22 (20), 5612-5621 (2003).
- Lee, J. H., Paull, T. T. ATM activation by DNA double-strand breaks through the Mre11-Rad50-Nbs1 complex. Science. 308 (5721), 551-554 (2005).
- Rogakou, E. P., Pilch, D. R., Orr, A. H., Ivanova, V. S., Bonner, W. M. DNA double-stranded breaks induce histone H2AX phosphorylation on serine 139. Journal of Biological Chemistry. 273, 5858-5868 (1998).
- Kinner, A., Wu, W., Staudt, C., Iliakis, G. γ-H2AX in recognition and signaling of DNA double-strand breaks in the context of chromatin. Nucleic Acids Research. 36 (17), 5678-5694 (2008).
- Martin, O. A., Pilch, D. R., Redon, C., Bonner, W. M. Histone H2AX in DNA damage repair. Cancer Biology & Therapy. 2 (3), 233-235 (2003).
- Rogakou, E. P., Boon, C., Redon, C., Bonner, W. M. Megabase chromatin domains involved in DNA double-strand breaks in vivo. Journal of Cell Biology. 146 (5), 905-916 (1999).
- Stucki, M., et al. MDC1 directly binds phosphorylated histone H2AX to regulate cellular responses to DNA double-strand breaks. Cell. 123 (7), 1213-1226 (2005).
- Lou, Z., et al. MDC1 maintains genomic stability by participating in the amplification of ATM-dependent DNA damage signals. Molecular Cell. 21 (2), 187-200 (2006).
- Chapman, J. R., Jackson, S. P. Phospho-dependent interaction between NBS1 and MDC1 mediate chromatin retention of the MRN complex at sites of DNA damage. EMBO Reports. 9 (8), 795-801 (2008).
- Melander, F., et al. Phosphorylation of SDT repeats in the MDC1 N terminus triggers retention of NBS1 at the DNA damage-modified chromatin. Journal of Cell Biology. 181 (2), 213-226 (2008).
- Branzei, D., Foiani, M. Regulation of DNA repair throughout the cell cycle. Nature Review. Molecular Cell Biology. 9 (4), 297-308 (2008).
- Chiruvella, K. K., Liang, Z., Wilson, T. E. Repair of double-strand breaks by end joining. Cold Spring Harbor Perspectives in Biology. 5 (5), 012757 (2013).
- Mehta, A., Haber, J. E. Sources of DNA double-strand breaks and models of recombinational DNA repair. Cold Spring Harbor Perspectives in Biology. 6 (9), 016428 (2014).
- Symington, L. S., Gautier, J. Double-strand break end resection and repair pathway choice. Annual Review of Genetics. 45, 247-271 (2011).
- Huertas, P. DNA resection in eukaryotes: deciding how to fix the break. Nature Structural & Molecular Biology. 17 (1), 11-16 (2010).
- Nimonkar, A. V., et al. BLM-DNA2-RPA-MRN and EXO1-BLM-RPA-MRN constitute two DNA end resection machineries for human DNA break repair. Genes & Development. 25 (4), 350-362 (2011).
- Garcia, V., Phelps, S. E. L., Gray, S., Neale, M. J. Bidirectional resection of DNA double-strand breaks by Mre11 and Exo1. Nature. 479 (7372), 241-244 (2011).
- Sturzenegger, A., et al. DNA2 cooperates with the WRN and BLM RecQ helicases to mediate long-range DNA end resection in human cells. Journal of Biological Chemistry. 289 (39), 27314-27326 (2014).
- Daley, J. M., Niu, H., Miller, A. S., Sung, P. Biochemical mechanism of DSB end resection and its regulation. DNA Repair. 32, 66-74 (2015).
- Sartori, A. A., et al. Human CtIP promotes DNA end resection. Nature. 450 (7169), 509-514 (2007).
- Chen, L., Nievera, C. J., Lee, A. Y. L., Wu, X. Cell cycle-dependent complex formation of BRCA1-CtIP-MRN is important for DNA double-strand break repair. Journal of Biological Chemistry. 283, 7713-7720 (2008).
- Yun, M. H., Hiom, K. CtIP-BRCA1 modulates the choice of DNA double-strand break repair pathway throughout the cell cycle. Nature. 459 (7245), 460-463 (2009).
- Sung, P., Klein, H. Mechanism of homologous recombination: mediators and helicases take on regulatory functions. Nature Review. Molecular Cell Biology. 7, 739-750 (2006).
- San Filippo, J., Sung, P., Klein, H. Mechanisms of eukaryotic homologous recombination. Annual Review of Biochemistry. 77, 229-257 (2008).
- Jasin, M., Rothstein, R. Repair of strand breaks by homologous recombination. Cold Spring Harbor Perspectives in Biology. 5 (11), 012740 (2013).
- Dynan, W. S., Yoo, S. Interaction of Ku protein and DNA-dependent protein kinase catalytic subunit with nucleic acids. Nucleic Acids Research. 26 (7), 1551-1559 (1998).
- Lieber, M. R. The mechanism of double-strand DNA break repair by the nonhomologous DNA end-joining pathway. Annual Review of Biochemistry. 79, 181-211 (2010).
- Cejka, P. DNA end resection: nucleases team up with the right partners to initiate homologous recombination. Journal of Biological Chemistry. 290 (38), 22931-22938 (2015).
- Mirman, Z., de Lange, T. 53BP1: a DSB escort. Genes & Development. 34, 7-23 (2020).
- Cao, L., et al. A selective requirement for 53BP1 in the biological response to genomic instability induces by BRCA1 deficiency. Molecular Cell. 35 (4), 534-541 (2009).
- Zimmermann, M., de Lange, T. 53BP1: Pro choice in DNA repair. Trends in Cell Biology. 24 (2), 108-117 (2014).
- Mavragani, I. V., Nikitaki, Z., Kalospyros, S. A., Georgakilas, A. G. Ionizing Radiation and Complex DNA Damage: From Prediction to Detection Challenges and Biological Significance. Cancers (Basel). 11 (11), (2019).
- Nikitaki, Z., et al. Measurement of complex DNA damage induction and repair in human cellular systems after exposure to ionizing radiations of varying linear energy transfer (LET). Free Radical Research. 50, 64-78 (2016).
- Redon, C., et al. Histone H2A variants H2AX and H2AZ. Current Opinion in Genetics & Development. 12 (2), 162-169 (2002).
- Fernandez-Capetillo, O., Lee, A., Nussenzweig, M., Nussenzweig, A. H2AX: the histone guardian of the genome. DNA Repair. 3 (8-9), 959-967 (2004).
- Paull, T. T., et al. A critical role for histone H2AX in recruitment of repair factors to nuclear foci after DNA damage. Current Biology. 10 (15), 886-895 (2000).
- Sy, S. M. H., Huen, M. S. Y., Chen, J. PALB2 is an integral component of the BRCA complex required for homologous recombination repair. Proceedings of the National Academy of Sciences. 106 (17), 7155-7160 (2009).
- Buisson, R., Masson, J. Y. PALB2 self-interaction controls homologous recombination. Nucleic Acids Research. 40 (20), 10312-10323 (2012).
- Belotserkovskaya, R., et al. PALB2 chromatin recruitment restores homologous recombination in BRCA1-deficient cells depleted of 53BP1. Nature Communications. 11 (1), 819 (2020).
- Betts, J. A., et al. Long noncoding RNAs CUPID1 and CUPID2 mediate breast cancer risk at 11q13 by modulating the response to DNA damage. American Journal of Human Genetics. 101 (2), 255-266 (2017).
- Dray, E., et al. Molecular basis for enhancement of the meiotic DMC1 recombinase by RAD51 associated protein 1 (RAD51AP1). Proceedings of the National Academy of Sciences. 108 (9), 3560-3565 (2011).

